Details of Host Protein
| Host Protein General Information (ID: PT0773) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name |
Prelamin-A/C (LMNA)
|
Gene Name |
LMNA
|
||||||
| Host Species |
Homo sapiens
|
Uniprot Entry Name |
LMNA_HUMAN
|
||||||
| Protein Families |
Intermediate filament family
|
||||||||
| Subcellular Location |
Nucleus envelope Nucleus lamina
|
||||||||
| External Link | |||||||||
| NCBI Gene ID | |||||||||
| Uniprot ID | |||||||||
| Ensembl ID | |||||||||
| HGNC ID | |||||||||
| Function in Host |
Lamins are components of the nuclear lamina, a fibrous layeron the nucleoplasmic side of the inner nuclear membrane, which isthought to provide a framework for the nuclear envelope and may alsointeract with chromatin. Lamin A and C are present in equal amounts inthe lamina of mammals. Recruited by DNA repair proteins XRCC4 and IFFO1to the DNA double-strand breaks (DSBs) to prevent chromosometranslocation by immobilizing broken DNA ends. Playsan important role in nuclear assembly, chromatin organization, nuclearmembrane and telomere dynamics. Required for normal development ofperipheral nervous system and skeletal muscle and for muscle satellitecell proliferation. Required for osteoblastogenesis andbone formation. Also prevents fat infiltration of muscle and bone marrow, helping tomaintain the volume and strength of skeletal muscle and bone. Required for cardiac homeostasis.
[1-5]
Click to Show/Hide
|
||||||||
| Related KEGG Pathway | |||||||||
| Apoptosis | hsa04210 |
Pathway Map 
|
|||||||
| Hypertrophic cardiomyopathy | hsa05410 |
Pathway Map 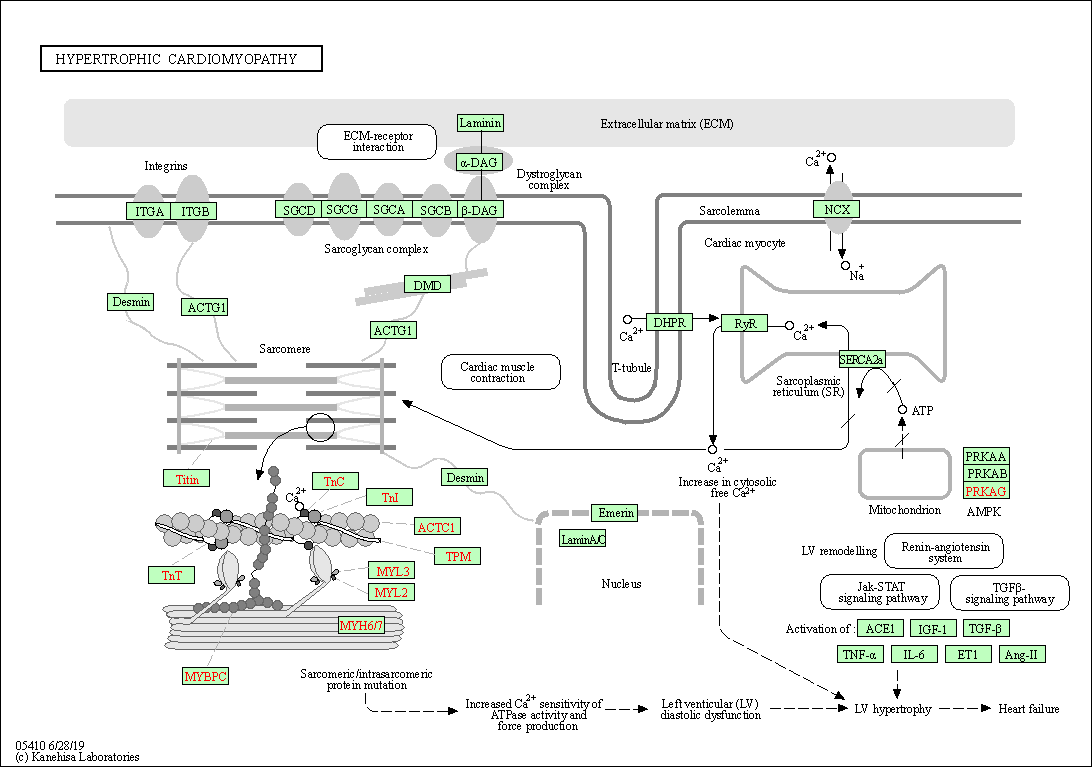
|
|||||||
| Arrhythmogenic right ventricular cardiomyopathy | hsa05412 |
Pathway Map 
|
|||||||
| Dilated cardiomyopathy | hsa05414 |
Pathway Map 
|
|||||||
| 3D Structure |
|
||||||||
| Function of This Protein During Virus Infection | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Virus Name | SARS-COV-2 | Protein Function | Pro-viral | [6] | |||||
| Infected Tissue | Lung | Infection Time | 7-9 Days | ||||||
| Infected Cell | Calu-3 Cells (Human epithelial cell line) | Cellosaurus ID | CVCL_0609 | ||||||
| Method Description | To detect the role of host protein LMNA in viral infection, LMNA protein knockout Calu-3 Cells were infected with SARS-COV-2 for 7 - 9 Days , and the effects on infection was detected through CRISPR-based genome-wide gene-knockout screen. | ||||||||
| Results | It is reported that knockout of LMNA leads to the decreased SARS-CoV-2 RNA levels compared with control group. | ||||||||
| Full List of Virus RNA Interacting with This Protien | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| RNA Region: 3'-UTR (hCoV-19/IPBCAMS-YL01/2020 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[7] | |||||||
| Strains Name |
hCoV-19/IPBCAMS-YL01/2020
|
||||||||
| Strains Family |
Beta (B.1.351)
|
||||||||
| RNA Binding Region |
3'-UTR
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Interaction Type | Unlikely to be direct binder | ||||||||
| Infection Cells | Huh7.5.1 cells (Hepatocyte derived cellular carcinoma cell) (CVCL_E049 ) | ||||||||
| Cell Originated Tissue | Liver | ||||||||
| Infection Time | 30 h | ||||||||
| Interaction Score | MIST = 0.685515867 | ||||||||
| Method Description | comprehensive identification of RNA-binding proteins by massspectrometry (ChIRP-MS) | ||||||||
| Differential Gene Expression During SARS-COV-2 Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Phosphorylation after Virus Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
S107
[8] |
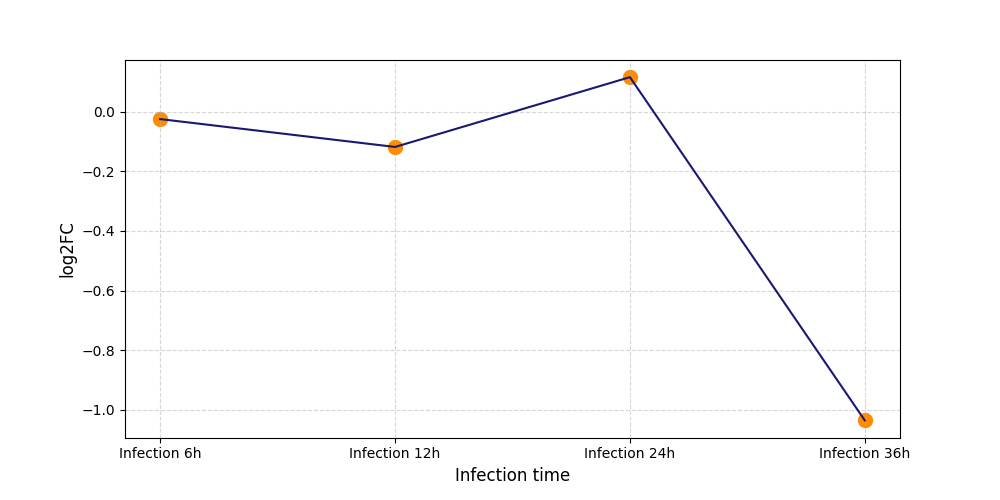 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S12
[8] |
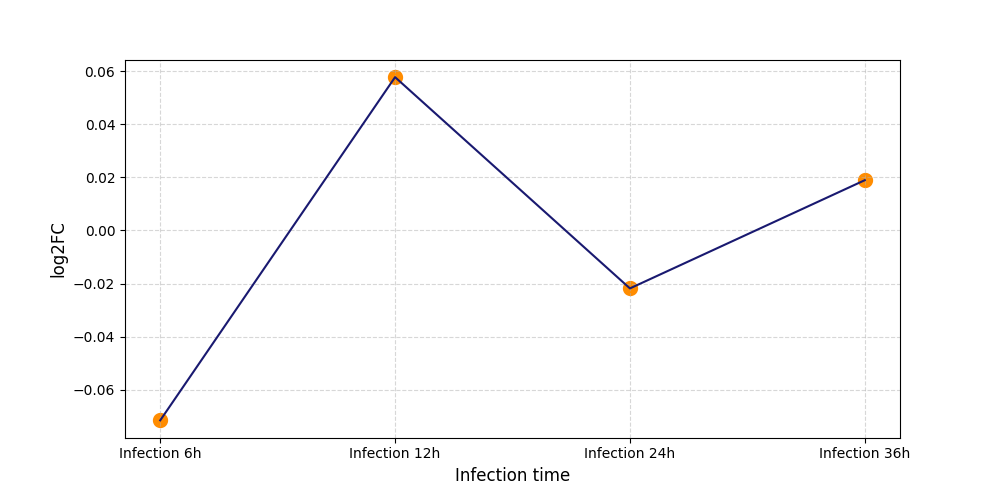 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S22
[9] |
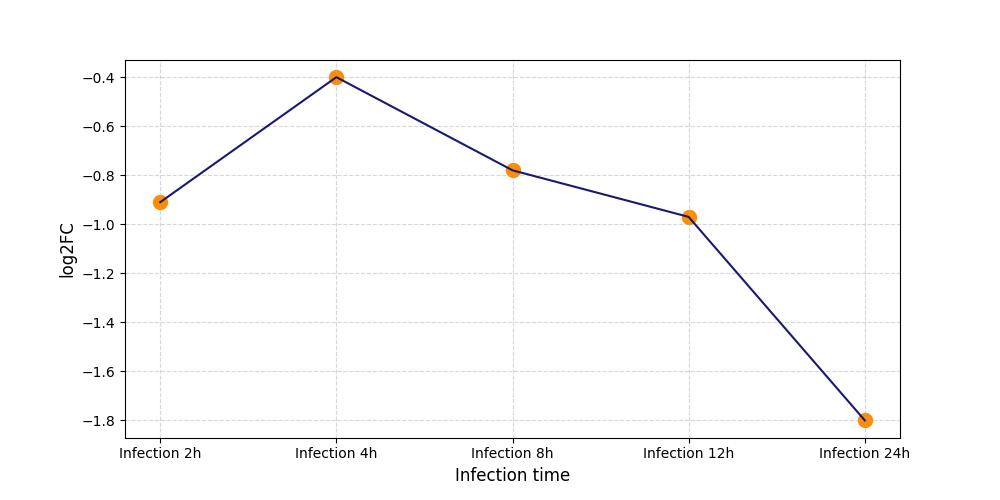 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S22
[8] |
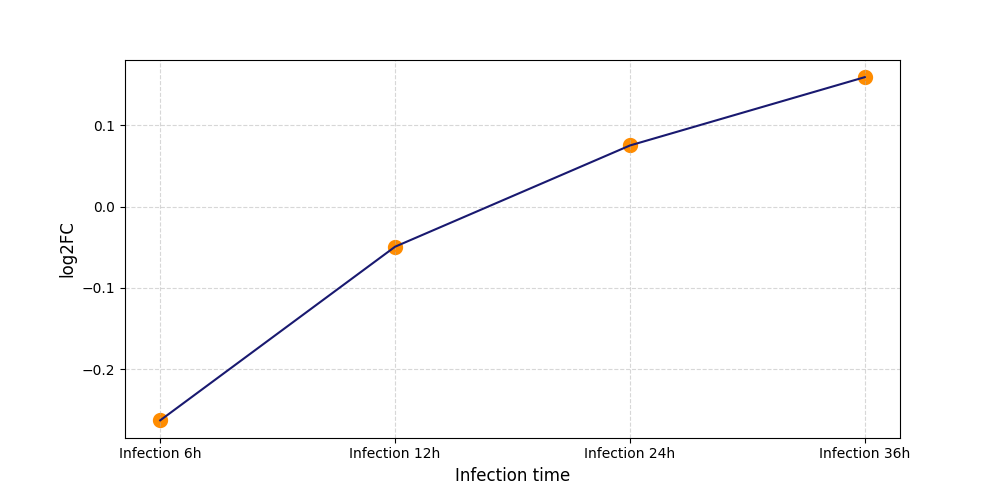 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S277
[8] |
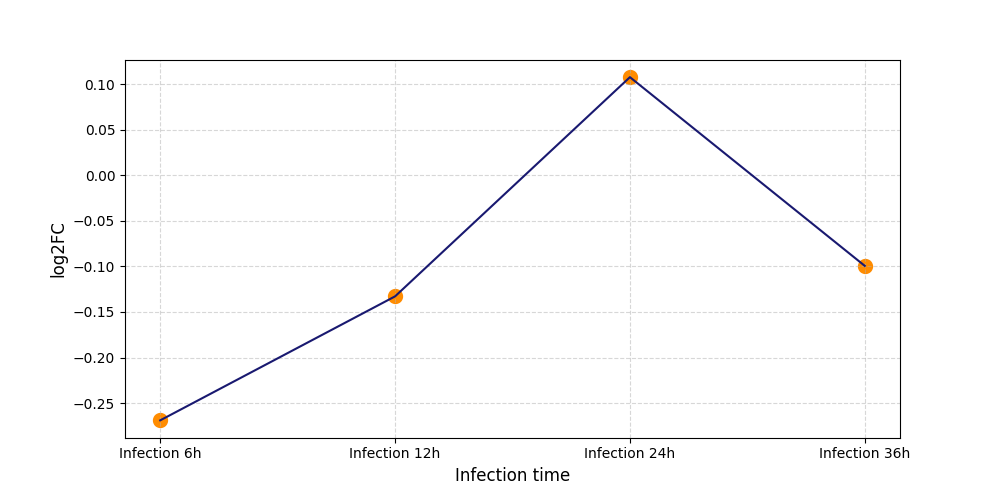 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S301
[8] |
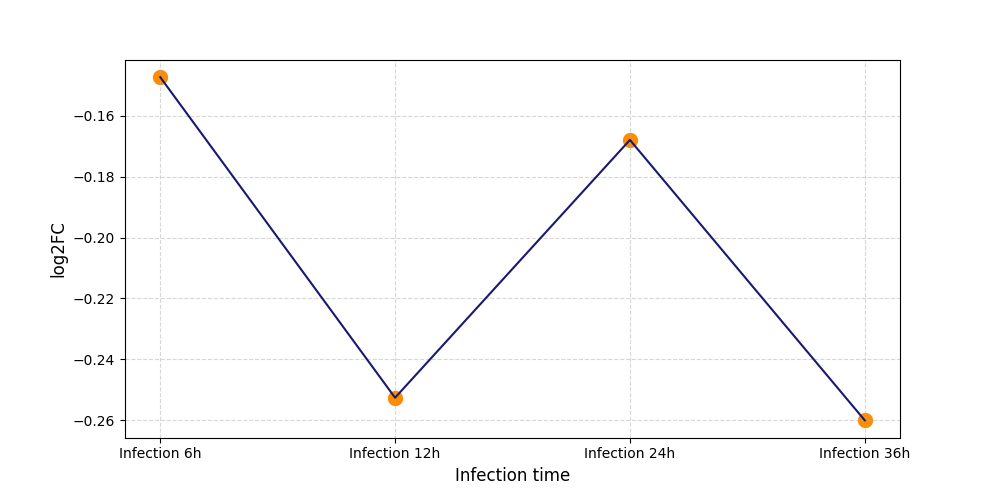 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S390
[9] |
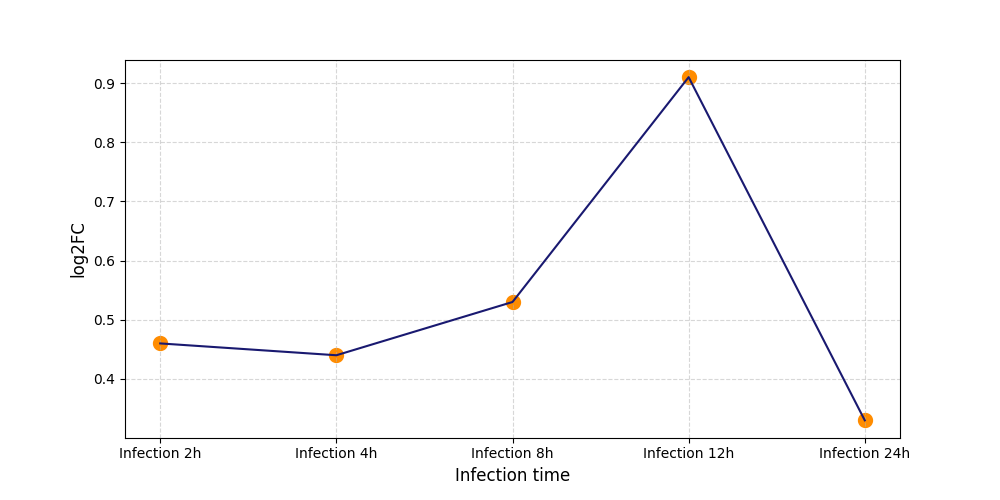 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S390
[8] |
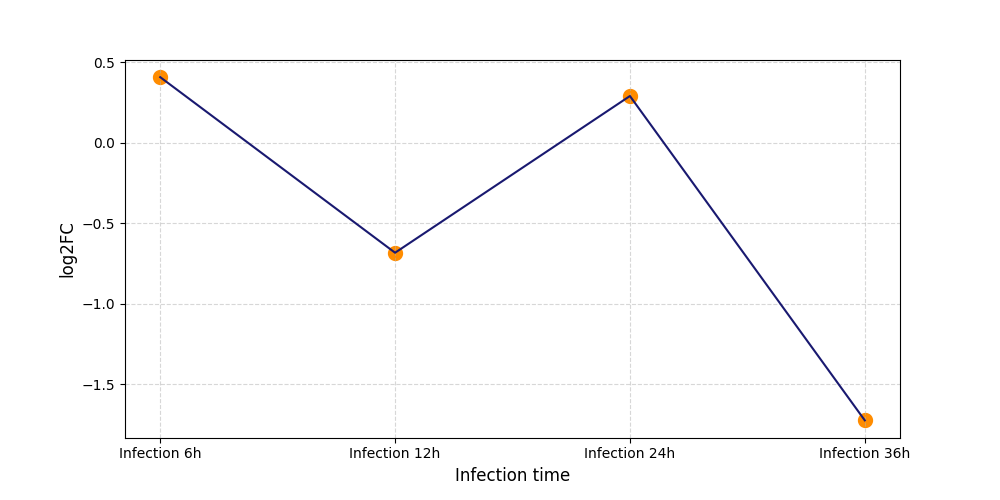 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S392
[9] |
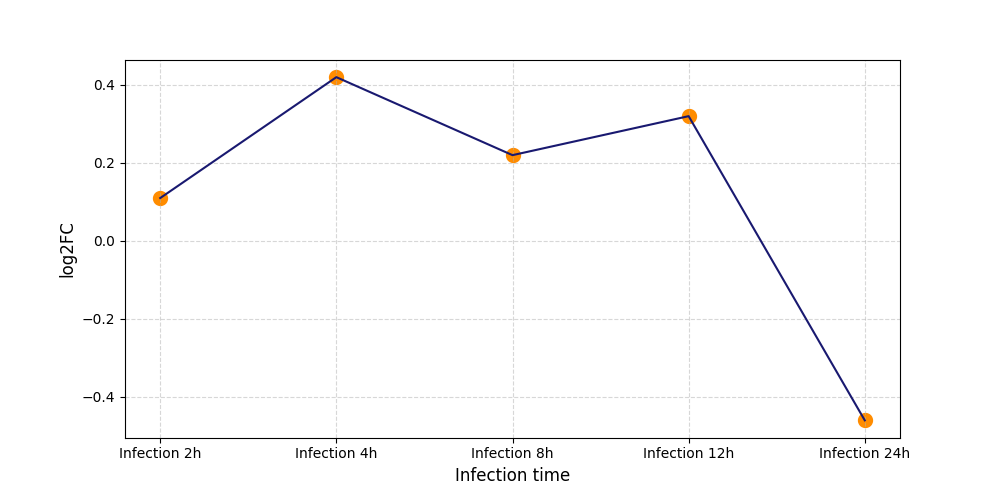 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S392
[8] |
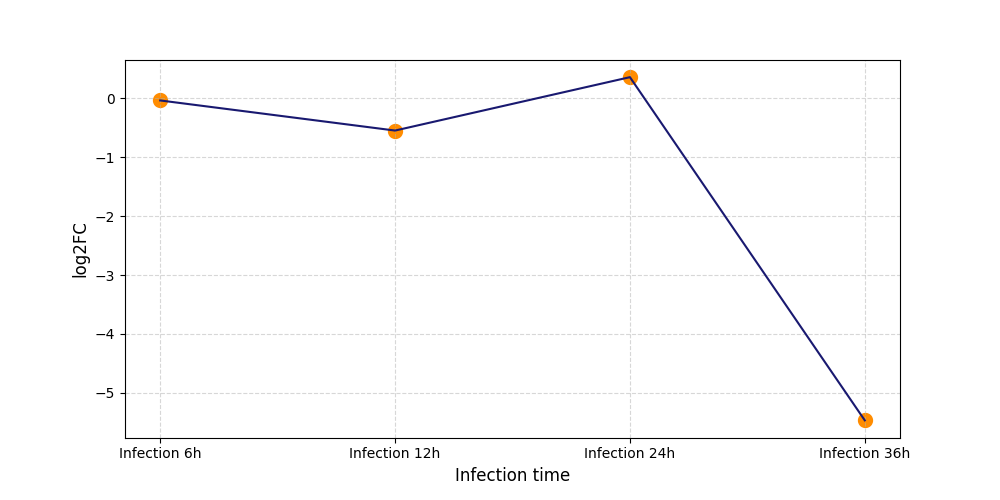 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
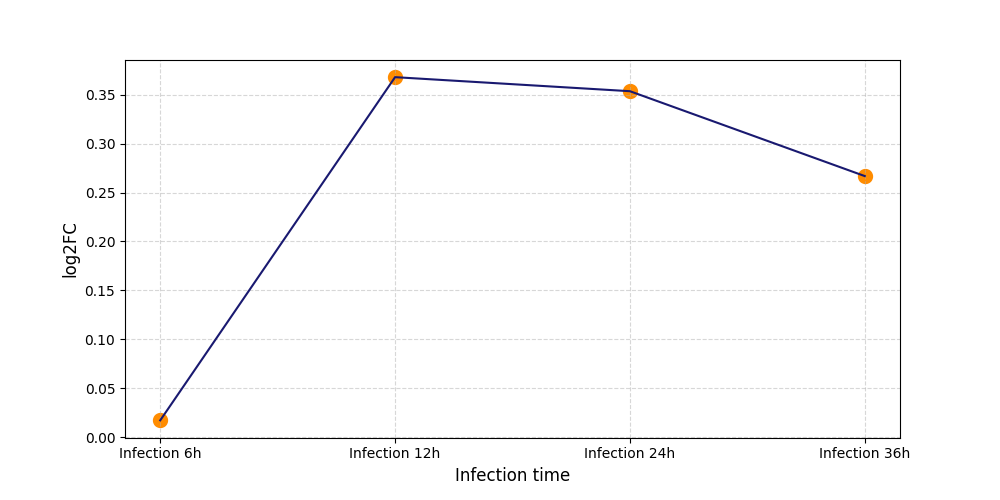
|
S395
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
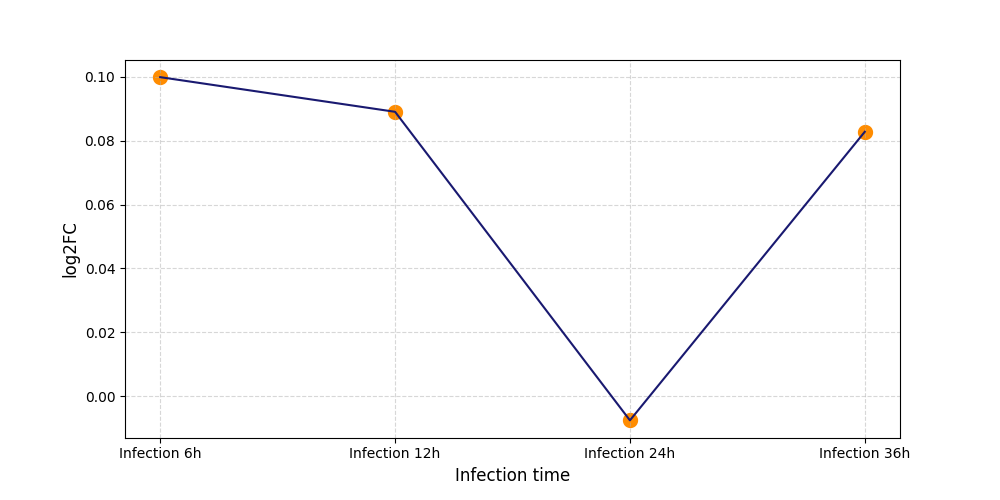
|
S403
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S404
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
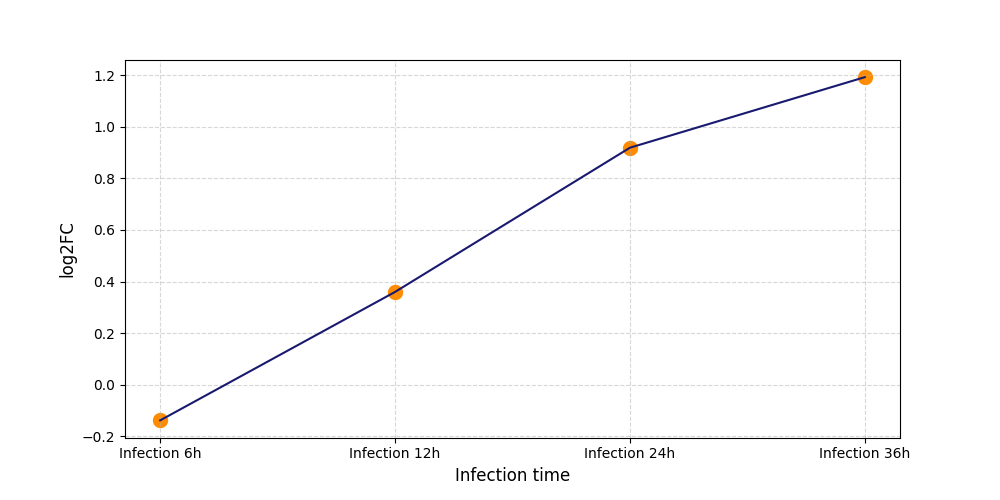
|
S404
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
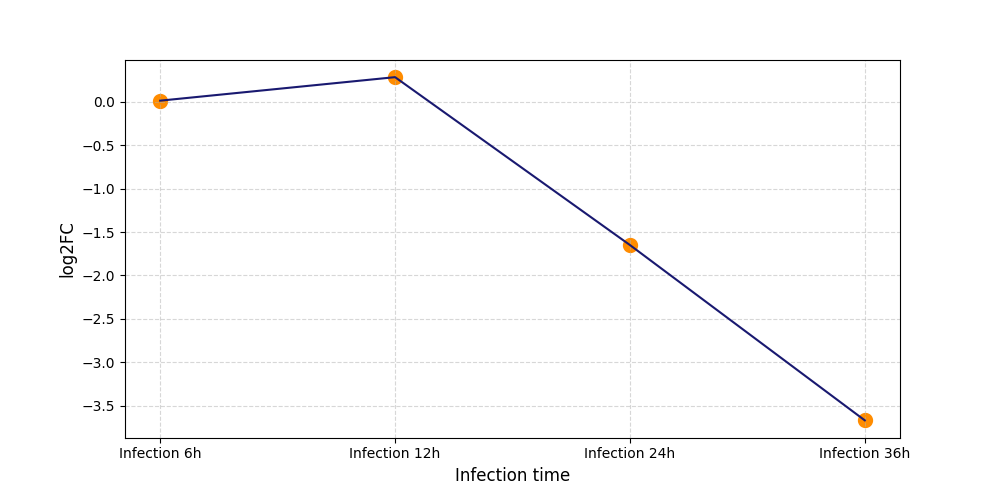
|
S406
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
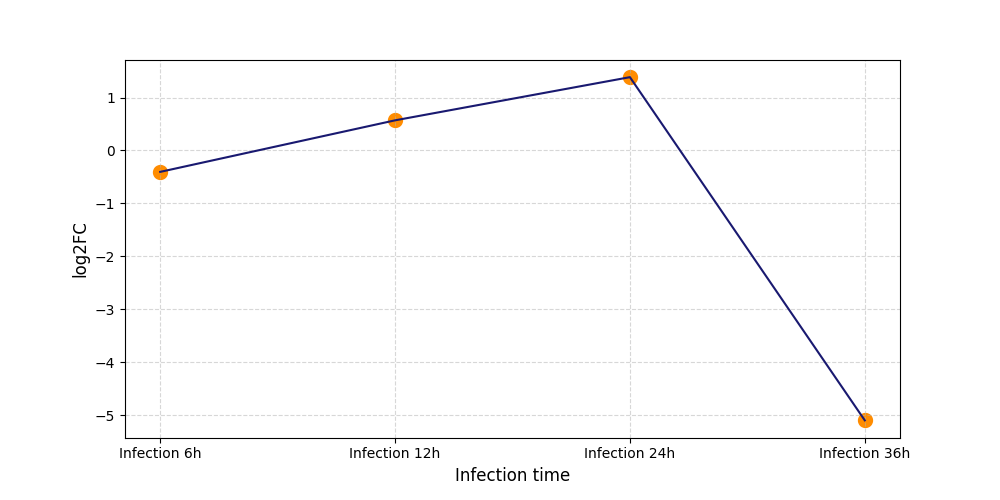
|
S407
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
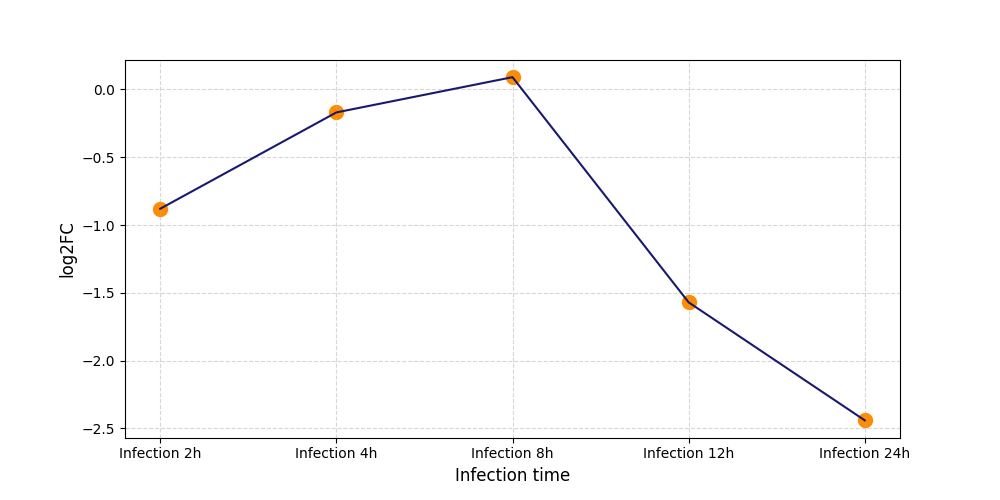
|
S414
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
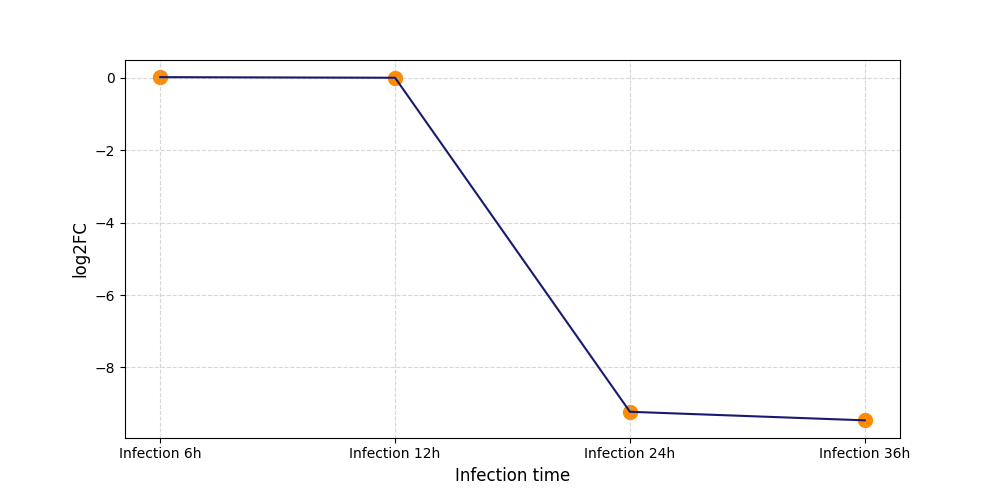
|
S414
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
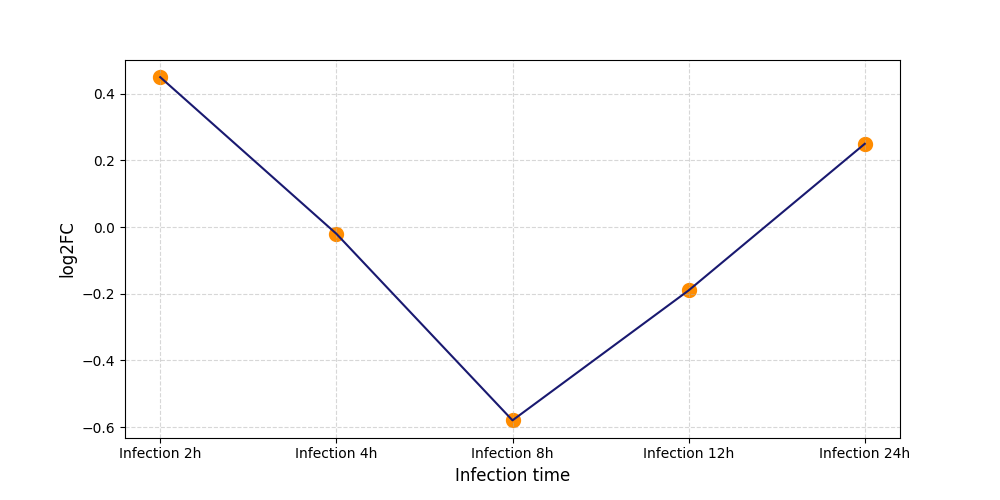
|
S423
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S423
[8] |
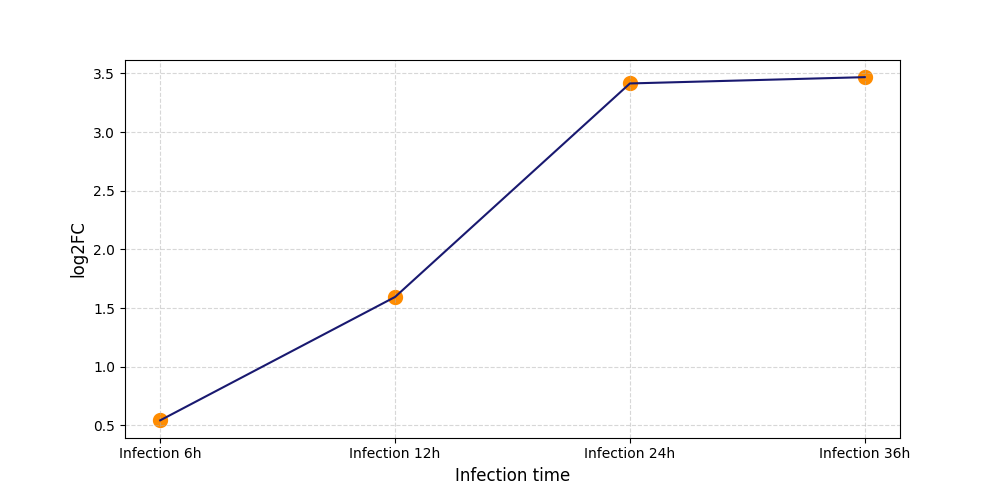 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S426
[8] |
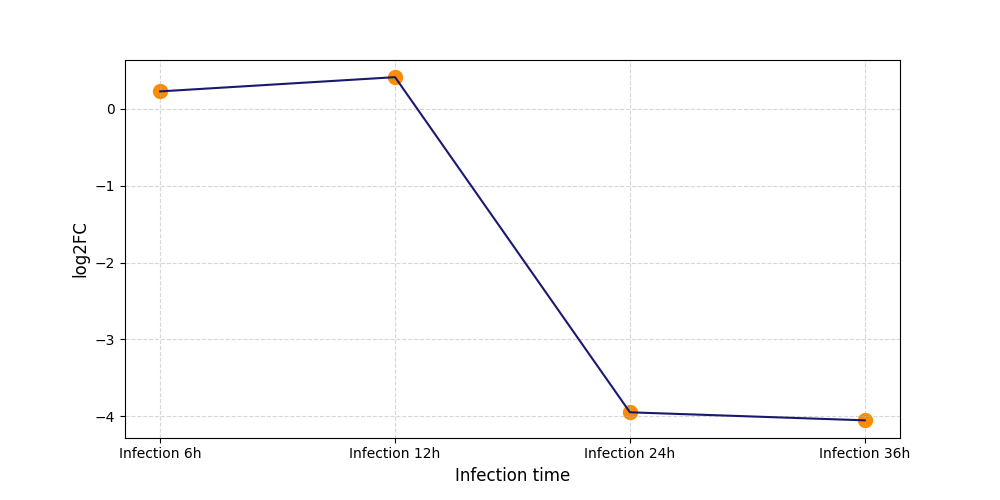 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S429
[8] |
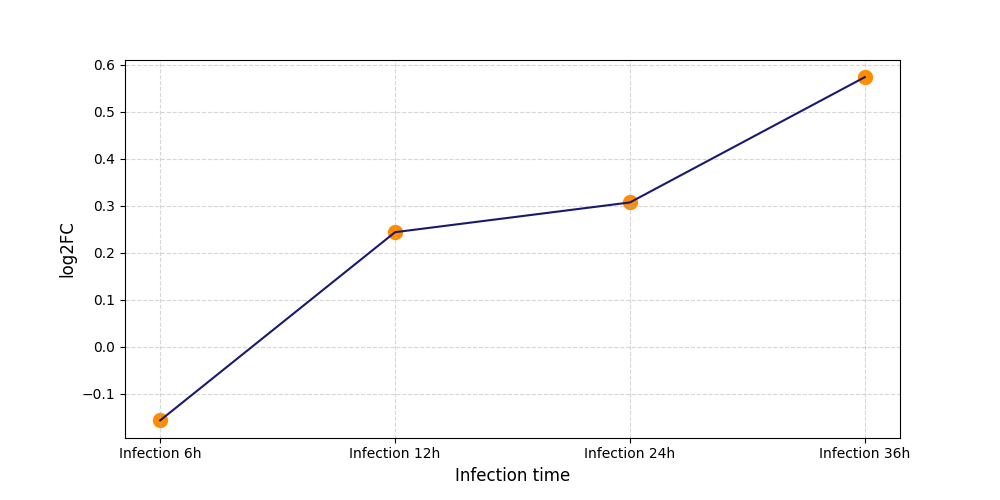 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S431
[8] |
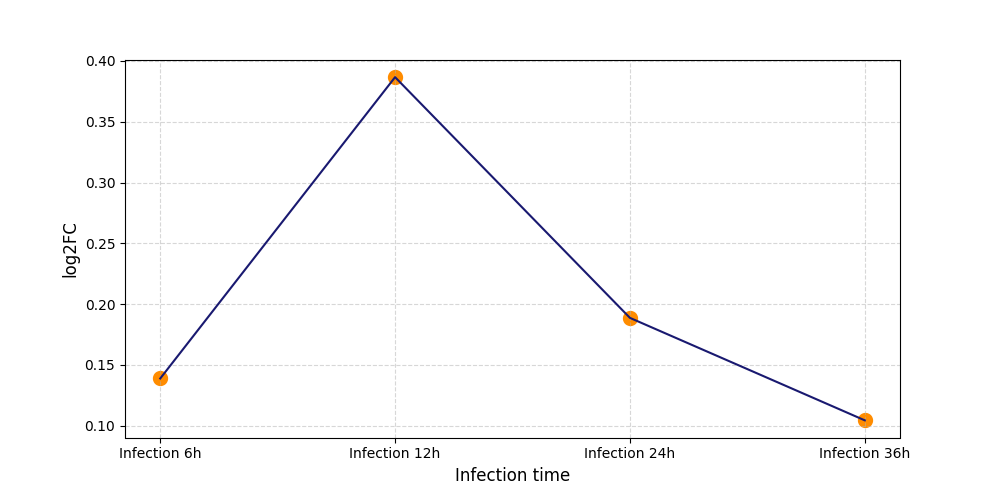 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S458
[9] |
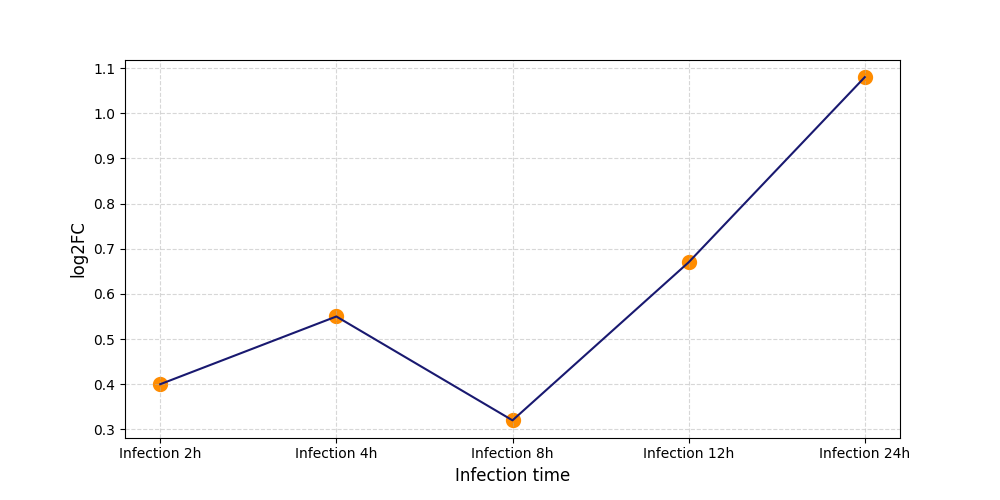 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S458
[8] |
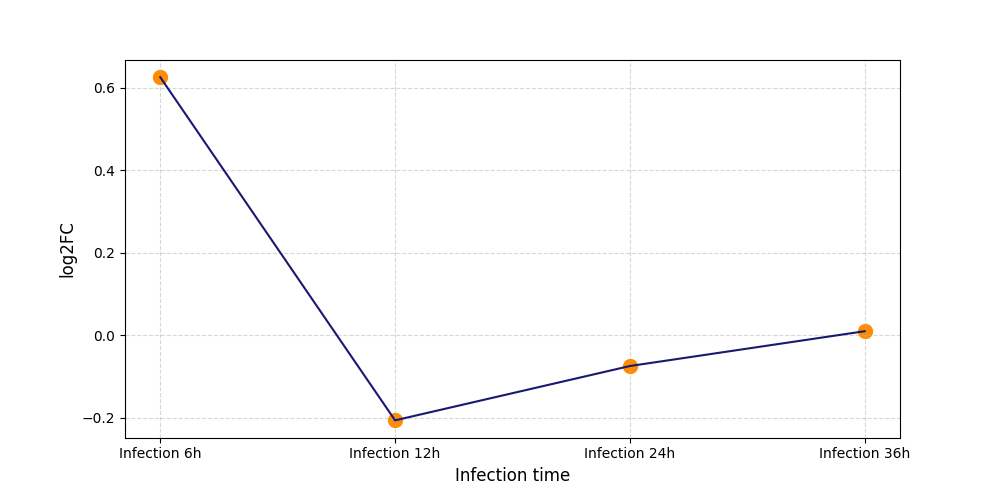 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S463
[8] |
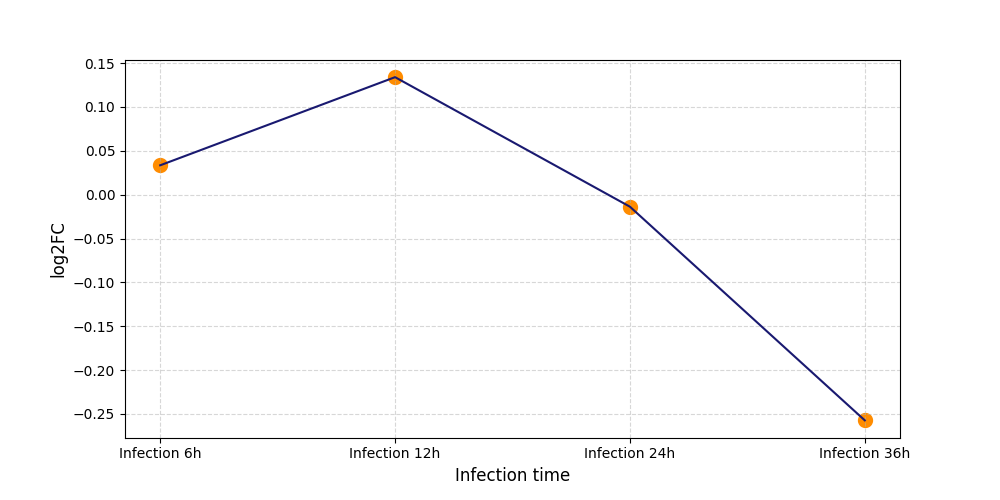 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S616
[8] |
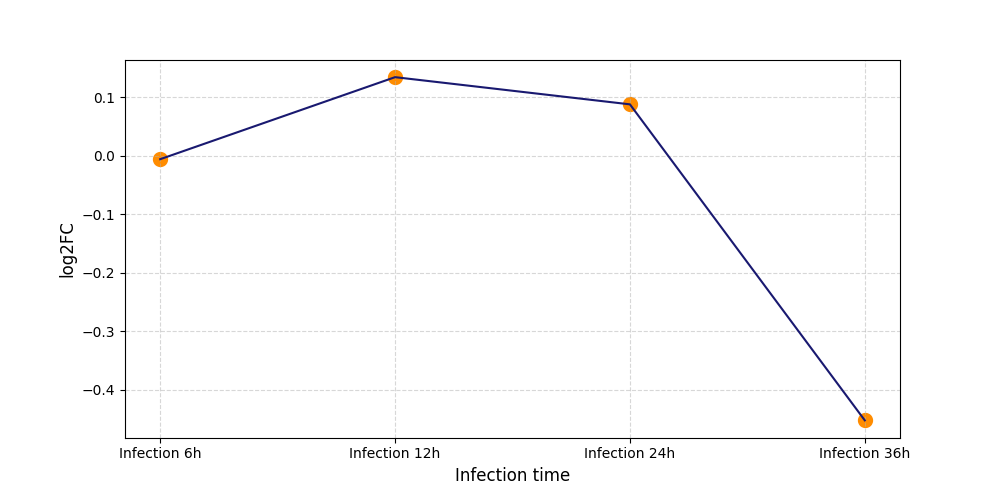 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S618
[8] |
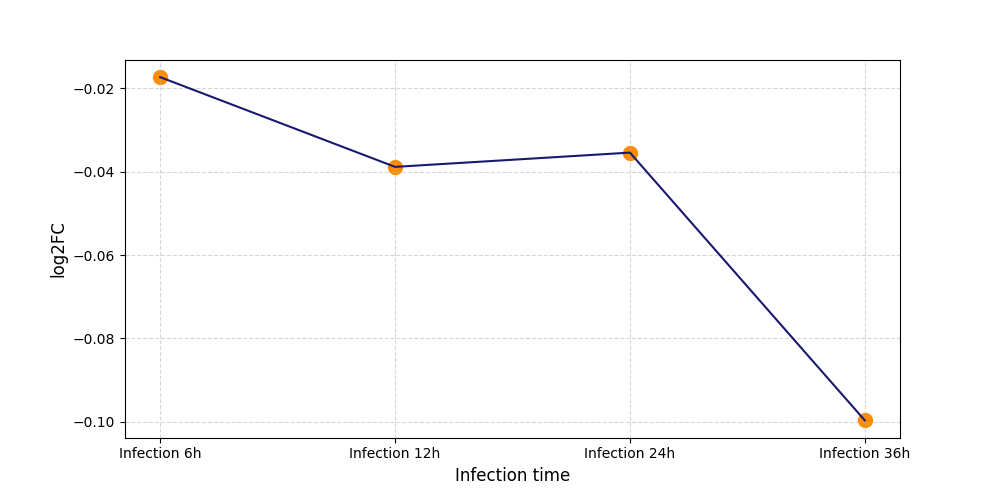 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S628
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S632
[8] |
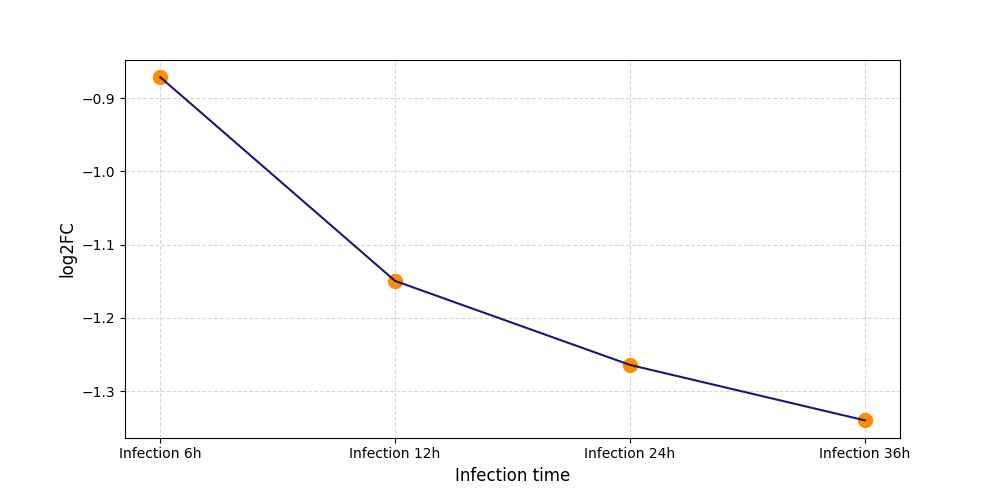 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S636
[8] |
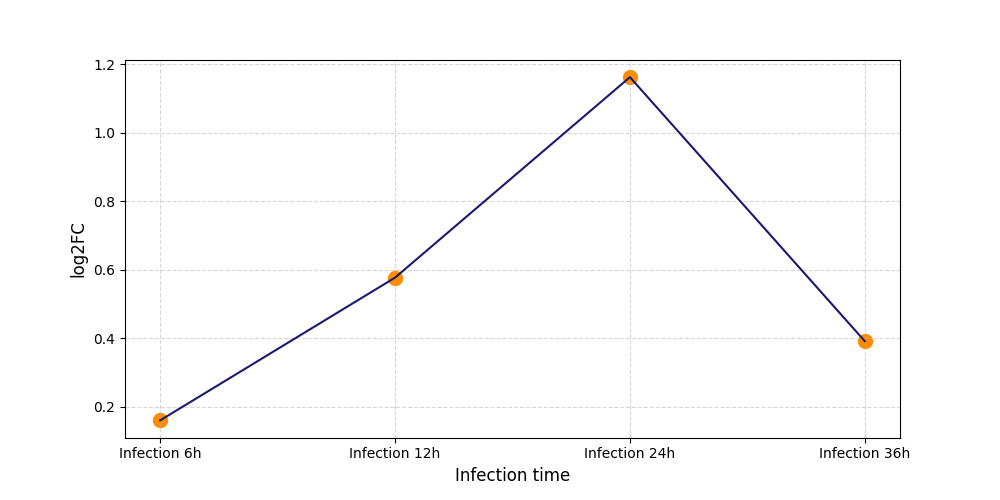 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S652
[8] |
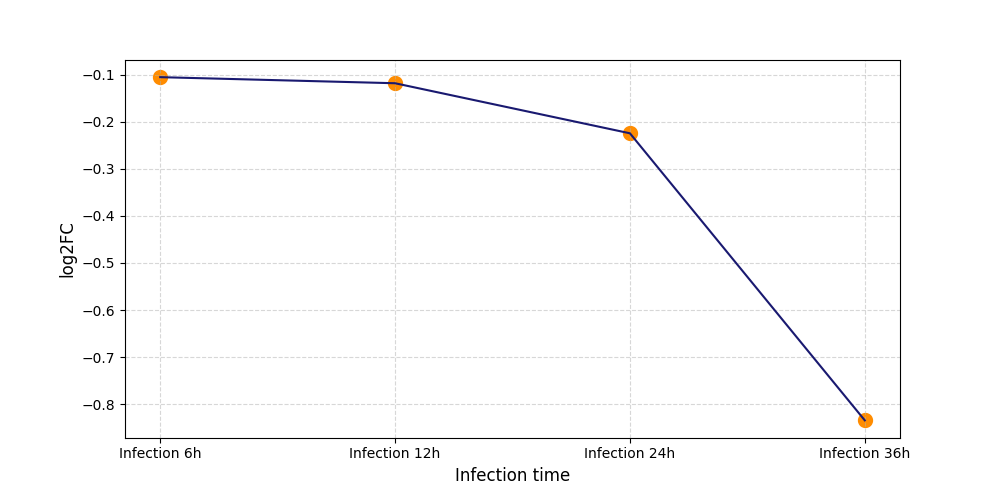 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T19
[8] |
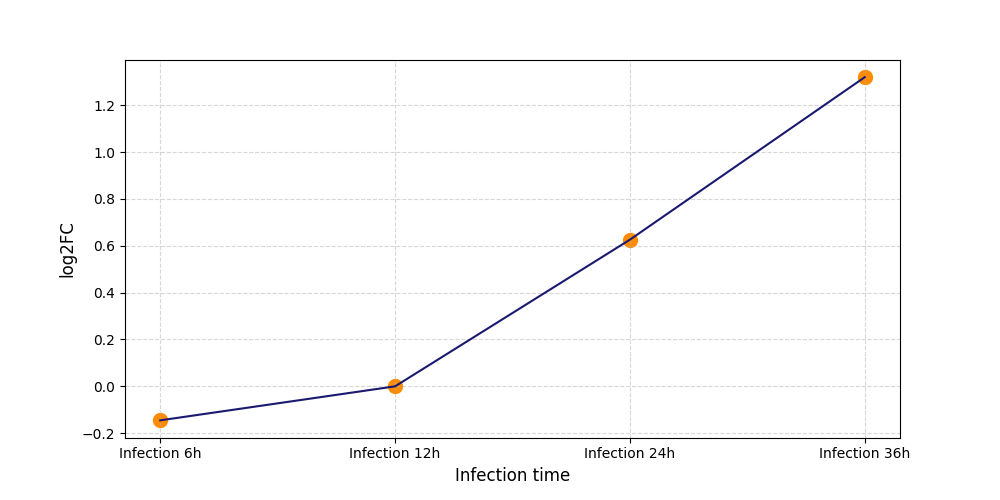 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T24
[8] |
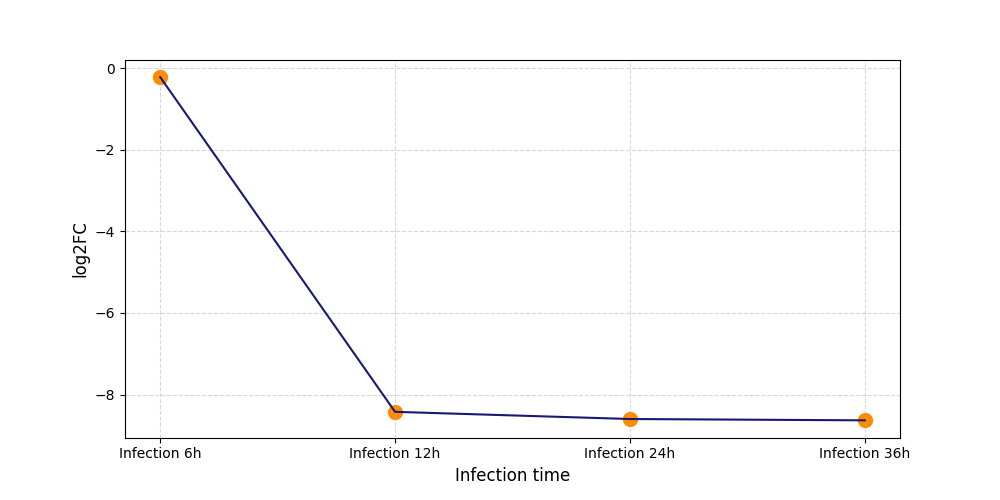 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T394
[8] |
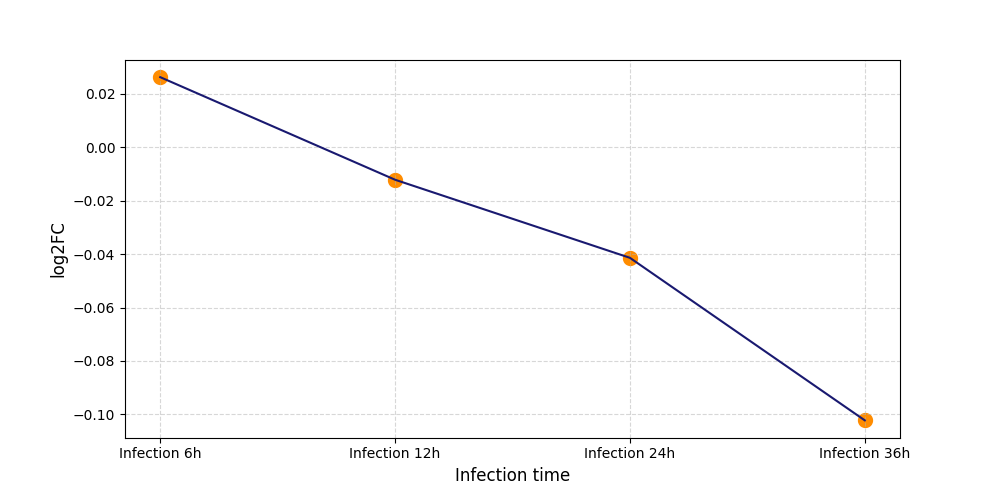 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T409
[8] |
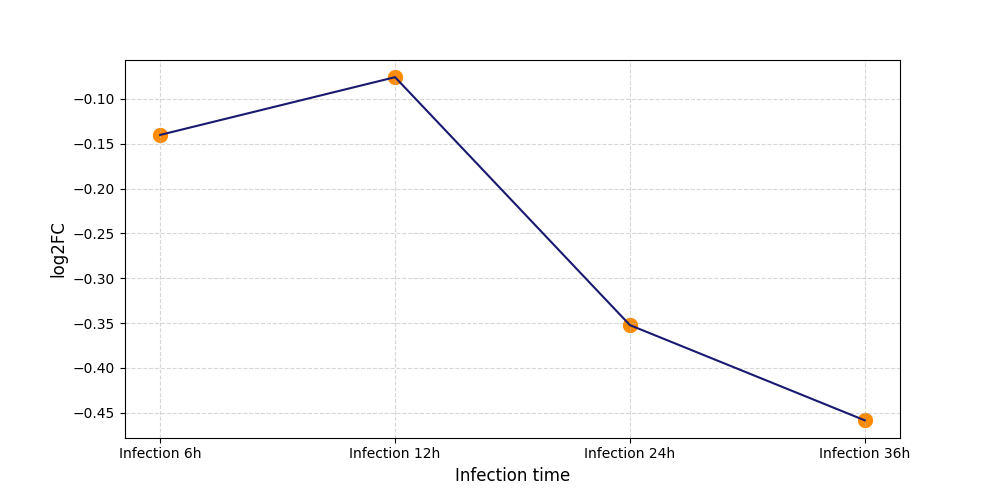 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T416
[8] |
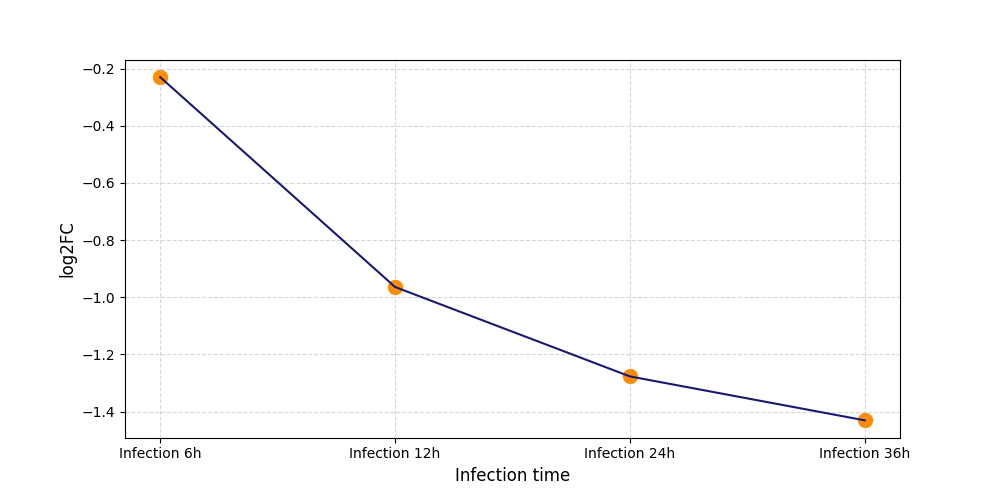 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T621
[8] |
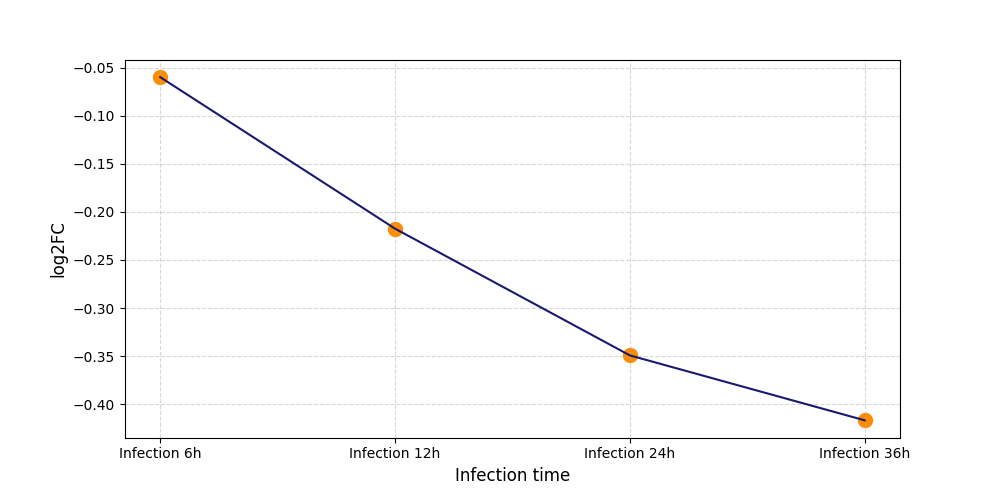 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Sequence Information |
METPSQRRATRSGAQASSTPLSPTRITRLQEKEDLQELNDRLAVYIDRVRSLETENAGLRLRITESEEVVSREVSGIKAAYEAELGDARKTLDSVAKERARLQLELSKVREEFKELKARNTKKEGDLIAAQARLKDLEALLNSKEAALSTALSEKRTLEGELHDLRGQVAKLEAALGEAKKQLQDEMLRRVDAENRLQTMKEELDFQKNIYSEELRETKRRHETRLVEIDNGKQREFESRLADALQELRAQHEDQVEQYKKELEKTYSAKLDNARQSAERNSNLVGAAHEELQQSRIRIDSLSAQLSQLQKQLAAKEAKLRDLEDSLARERDTSRRLLAEKEREMAEMRARMQQQLDEYQELLDIKLALDMEIHAYRKLLEGEEERLRLSPSPTSQRSRGRASSHSSQTQGGGSVTKKRKLESTESRSSFSQHARTSGRVAVEEVDEEGKFVRLRNKSNEDQSMGNWQIKRQNGDDPLLTYRFPPKFTLKAGQVVTIWAAGAGATHSPPTDLVWKAQNTWGCGNSLRTALINSTGEEVAMRKLVRSVTVVEDDEDEDGDDLLHHHHGSHCSSSGDPAEYNLRSRTVLCGTCGQPADKASASGSGAQVGGPISSGSSASSVTVTRSYRSVGGSGGGSFGDNLVTRSYLLGNSSPRTQSPQNCSIM
Click to Show/Hide
|
|---|