Details of Host Protein
| Host Protein General Information (ID: PT0197) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name |
Spinocerebellar ataxia type 2 protein (SCA2)
|
Gene Name |
ATXN2
|
||||||
| Host Species |
Homo sapiens
|
Uniprot Entry Name |
ATX2_HUMAN
|
||||||
| Protein Families |
Ataxin-2 family
|
||||||||
| Subcellular Location |
Cytoplasm
|
||||||||
| External Link | |||||||||
| NCBI Gene ID | |||||||||
| Uniprot ID | |||||||||
| Ensembl ID | |||||||||
| HGNC ID | |||||||||
| Function in Host |
Involved in EGFR trafficking, acting as negative regulator ofendocytic EGFR internalization at the plasma membrane.
[1]
Click to Show/Hide
|
||||||||
| Related KEGG Pathway | |||||||||
| Amyotrophic lateral sclerosis | hsa05014 |
Pathway Map 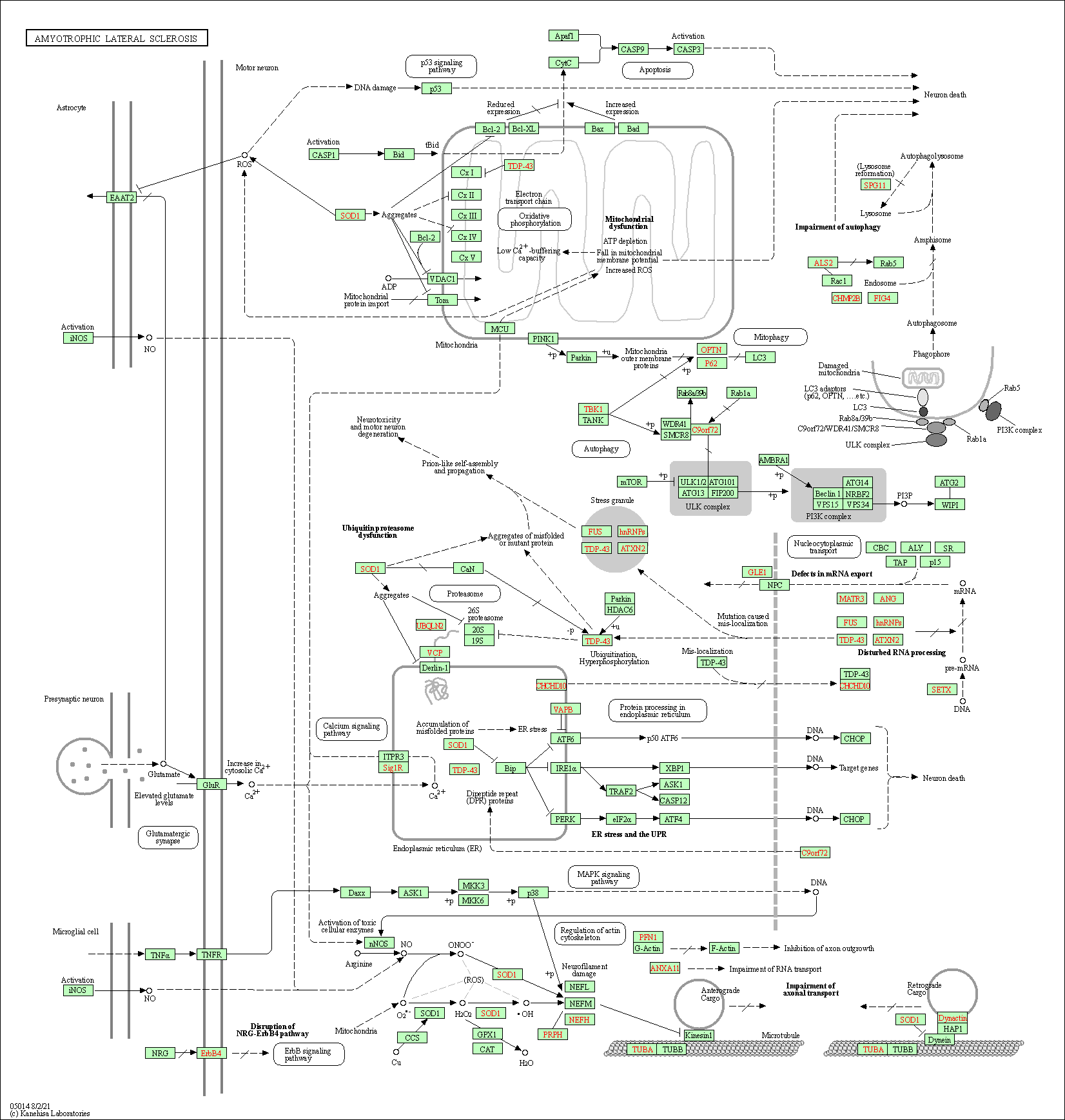
|
|||||||
| Spinocerebellar ataxia | hsa05017 |
Pathway Map 
|
|||||||
| Pathways of neurodegeneration - multiple diseases | hsa05022 |
Pathway Map 
|
|||||||
| 3D Structure |
|
||||||||
| Host Protein - Virus RNA Network | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Full List of Virus RNA Interacting with This Protien | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| RNA Region: 5'-UTR (hCoV-19/South Korea/KCDC03/2020 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[2] | |||||||
| Strains Name |
hCoV-19/South Korea/KCDC03/2020
|
||||||||
| Strains Family |
Alpha (B.1.1.7)
|
||||||||
| RNA Binding Region |
5'-UTR
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Interaction Type | Directly bind to SARS-CoV-23 RNA's 5' UTR region | ||||||||
| Infection Cells | Vero cells (epithelial kidney cell) (CVCL_0059 ) | ||||||||
| Cell Originated Tissue | kidney | ||||||||
| Infection Time | 24 h | ||||||||
| Interaction Score | P-adjust < 0.05 | ||||||||
| Method Description | Modified RNA antisense purification coupled with mass spectrometry (RAP-MS); label-free quantification (LFQ); liquid chromatography with tandem mass spectrometry (LC-MS/MS) | ||||||||
| RNA Region: Not Specified Virus Region (hCov-OC43/VR-759 Quebec/2019 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[2] | |||||||
| Strains Name |
hCov-OC43/VR-759 Quebec/2019
|
||||||||
| Strains Family |
Beta
|
||||||||
| RNA Binding Region |
Not Specified Virus Region
|
||||||||
| Virus Name |
Human coronavirus OC43 (HCov-OC43)
|
||||||||
| Infection Cells | HCT-8 cells (Human ileocaecal adenocarcinoma cell) HCT-8 cells (Human ileocaecal adenocarcinoma cell) (CVCL_2478 ) | ||||||||
| Cell Originated Tissue | Ileocecum | ||||||||
| Infection Time | 12h; 24; 36h; 48h | ||||||||
| Interaction Score | p_value < 0.05; FDR < 10% | ||||||||
| Method Description | Modified RNA antisense purification coupled with mass spectrometry (RAP-MS); label-free quantification (LFQ); liquid chromatography with tandem mass spectrometry (LC-MS/MS) | ||||||||
| Differential Gene Expression During SARS-COV-2 Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Phosphorylation after Virus Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
S466
[3] |
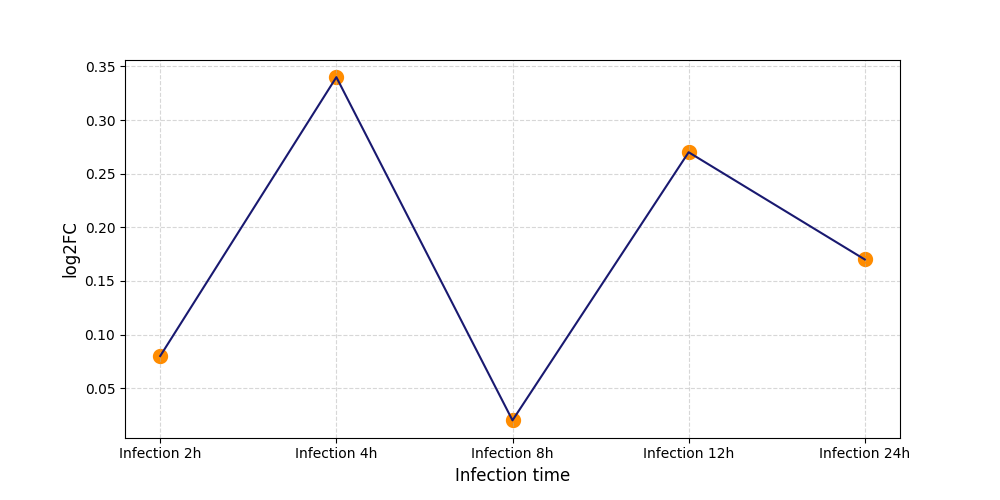 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S466
[4] |
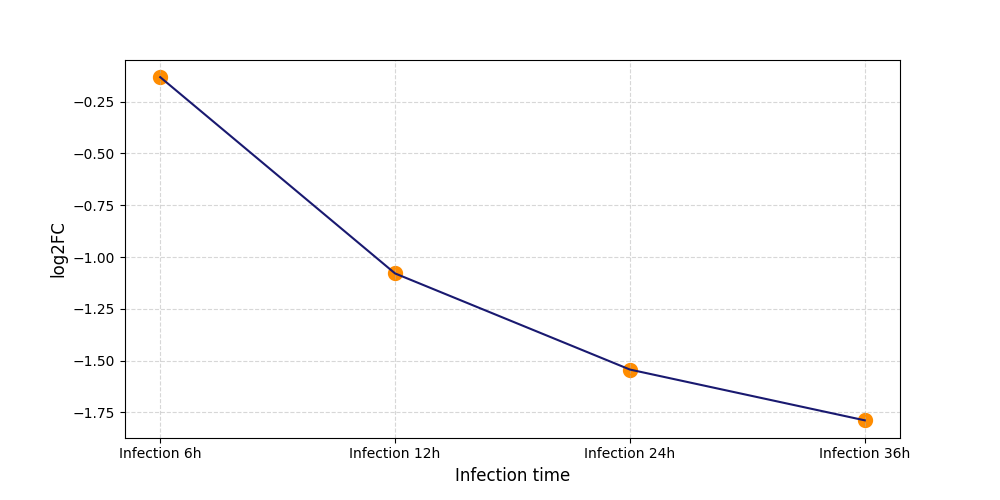 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
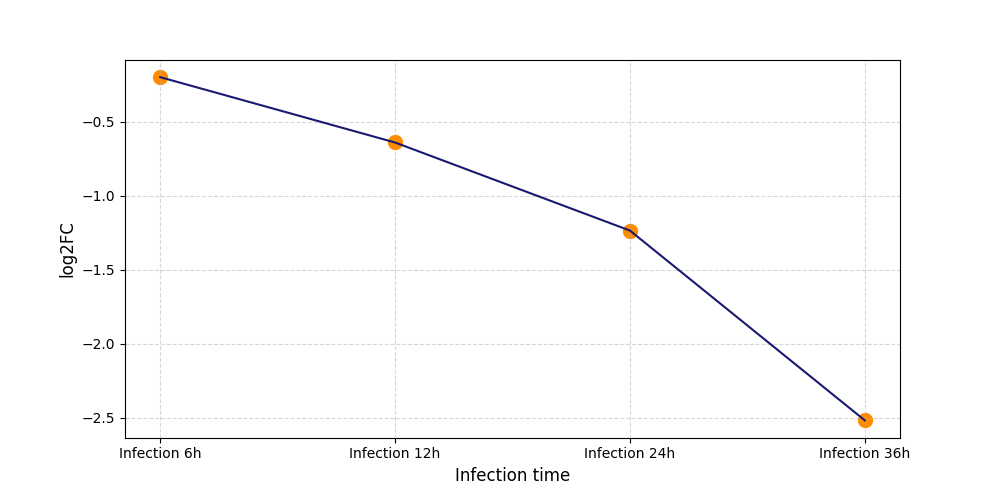
|
S593
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
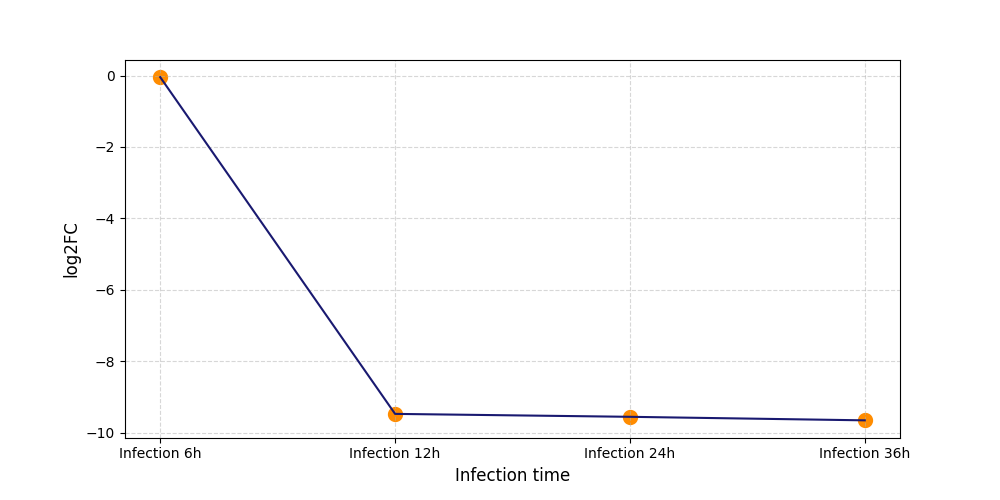
|
S597
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
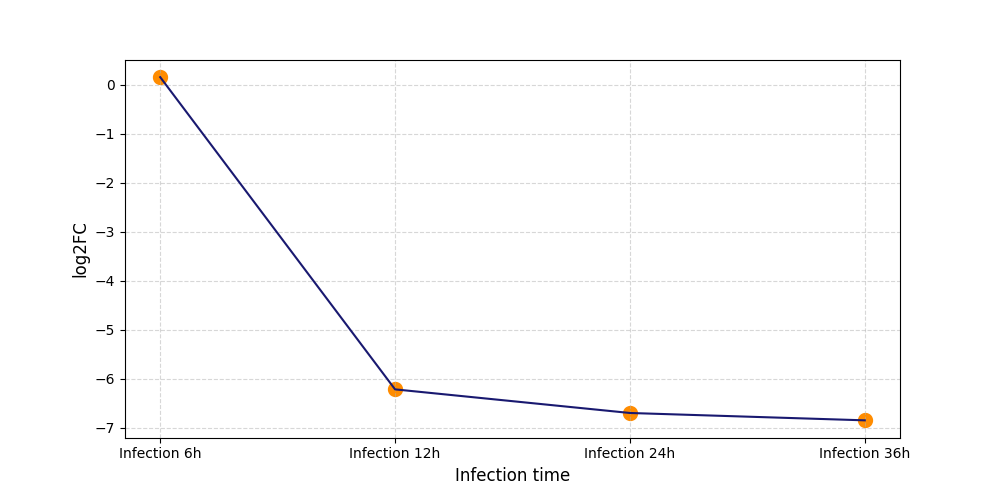
|
S600
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
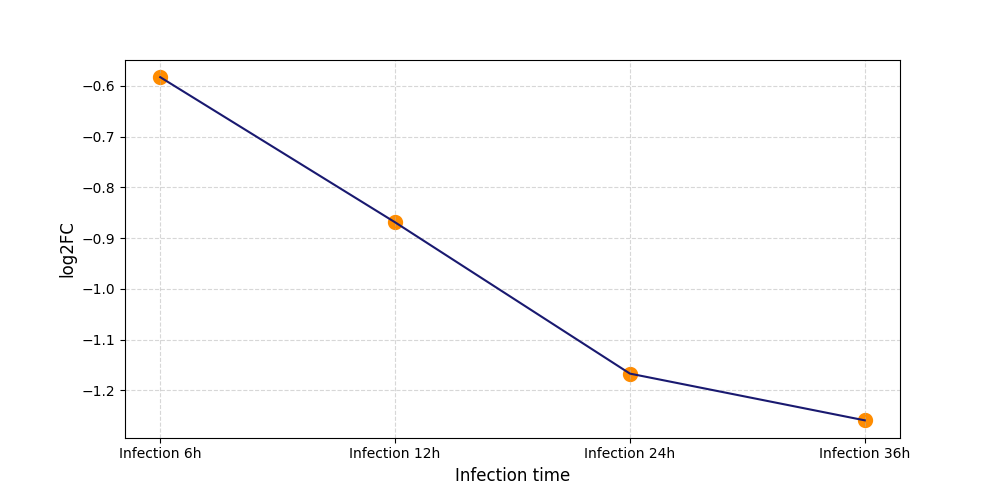
|
S604
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S624
[4] |
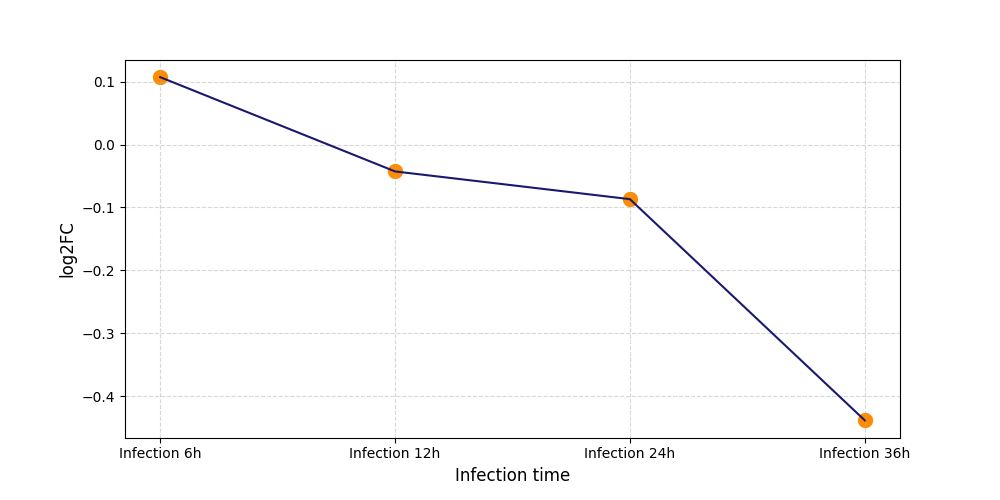 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S667
[4] |
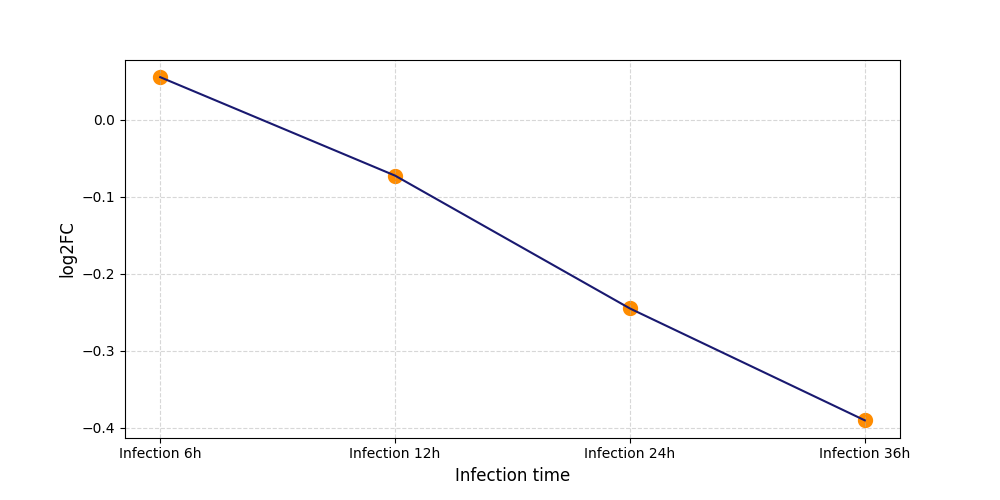 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S669
[4] |
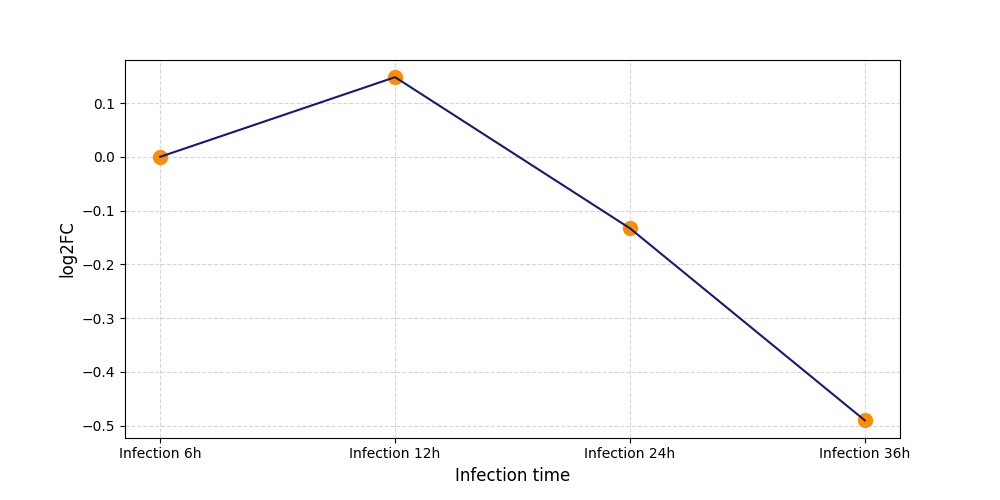 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S728
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S772
[4] |
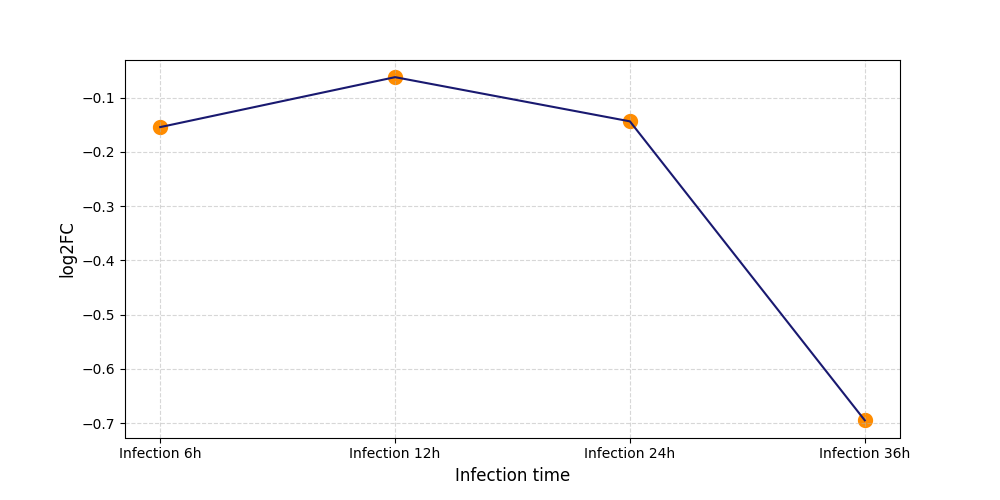 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S774
[4] |
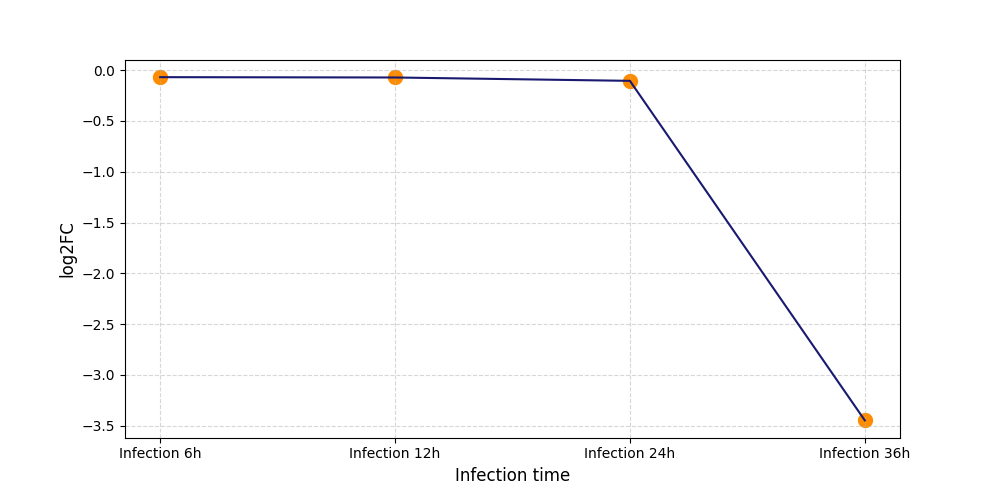 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S776
[4] |
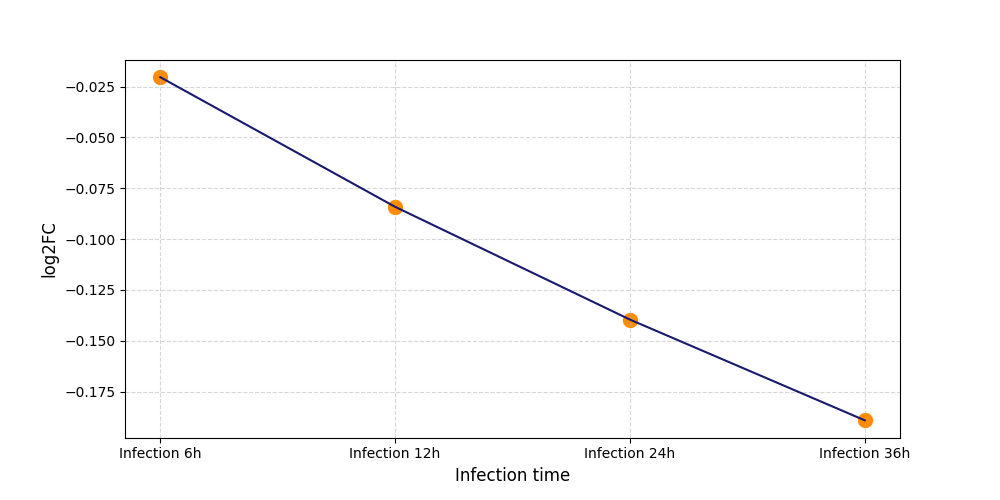 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S784
[4] |
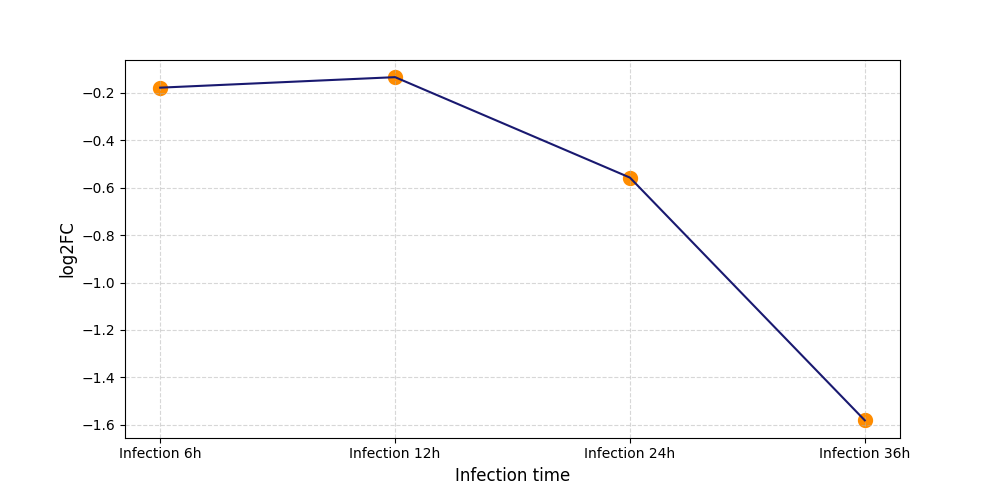 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S856
[4] |
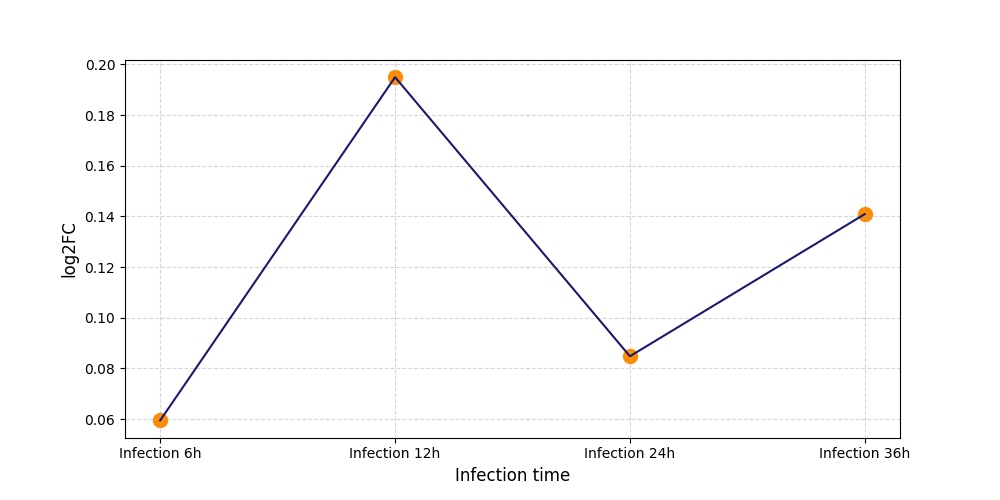 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
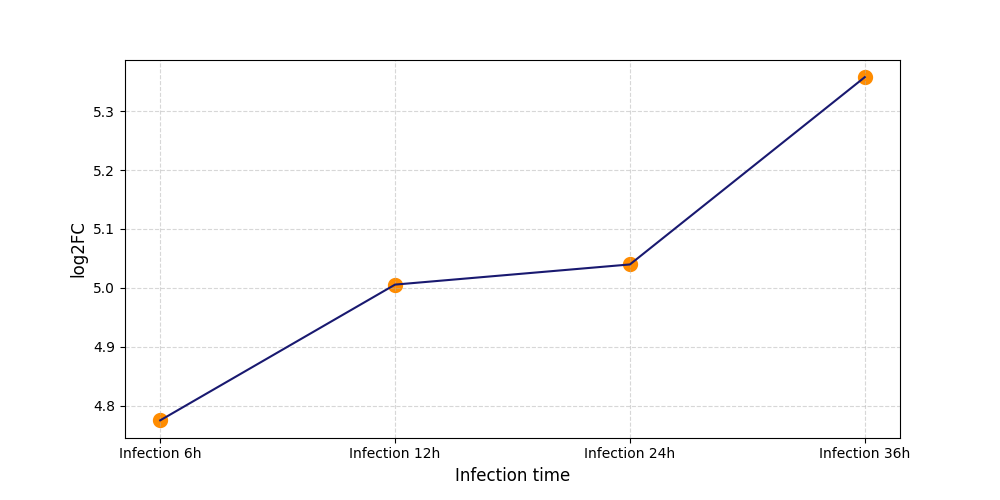
|
S857
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
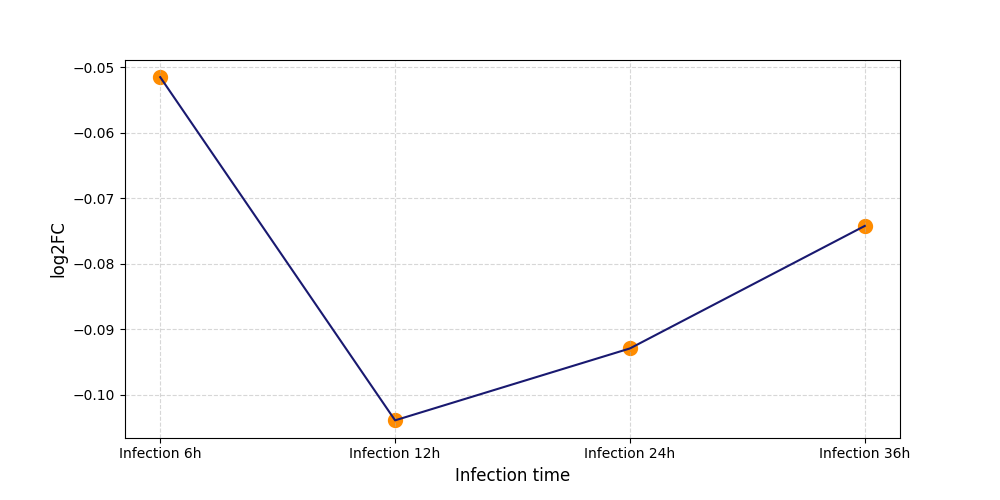
|
S861
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
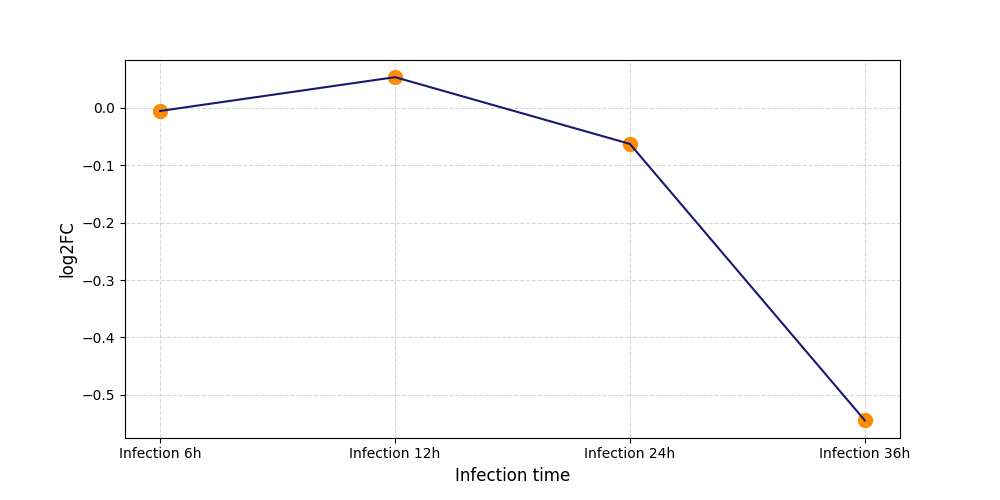
|
S863
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
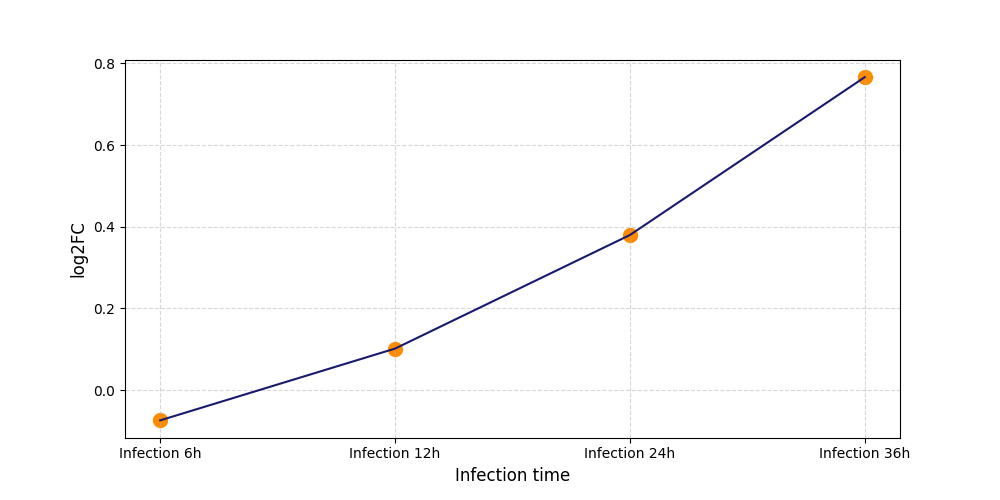
|
S865
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S867
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S889
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T532
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T660
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
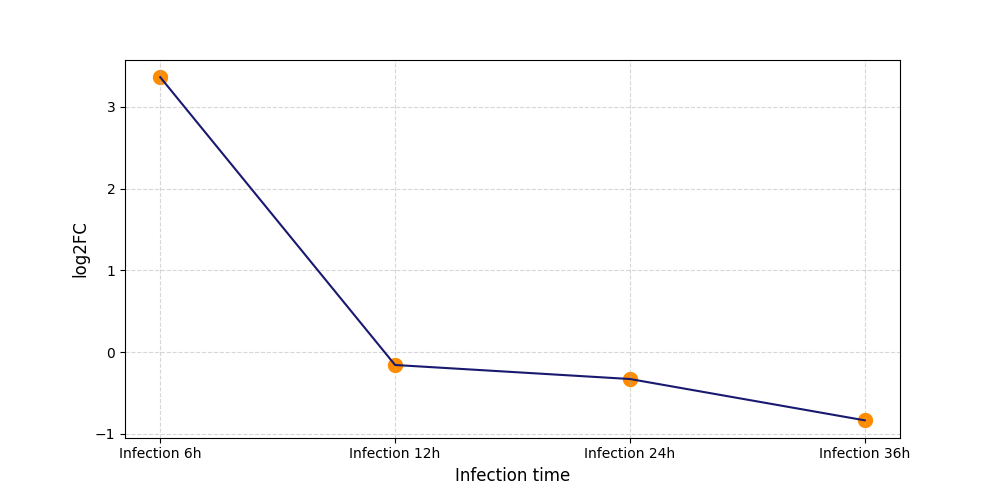
|
T666
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
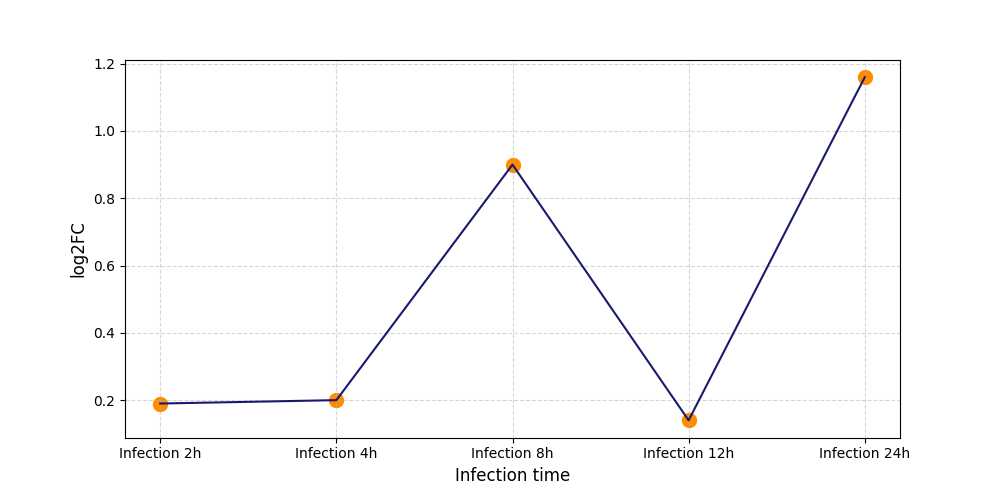
|
T730
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
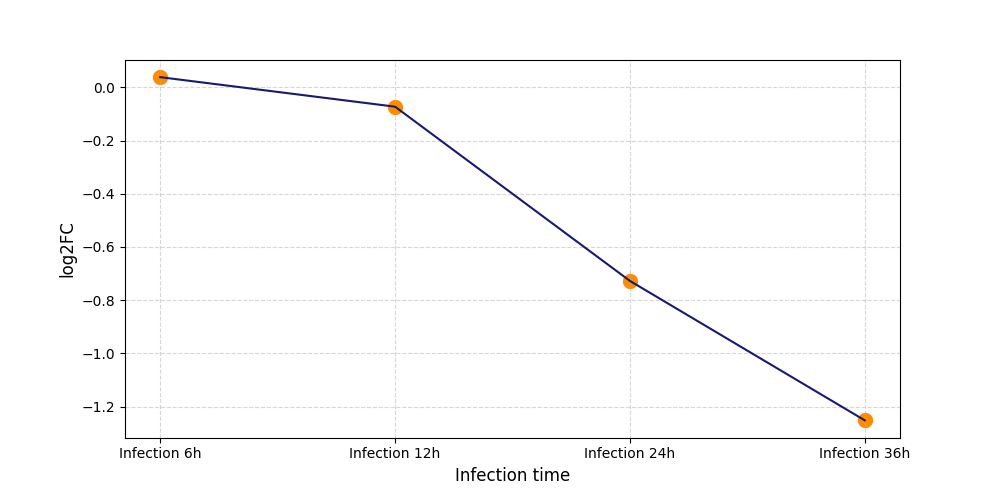
|
T741
[3] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
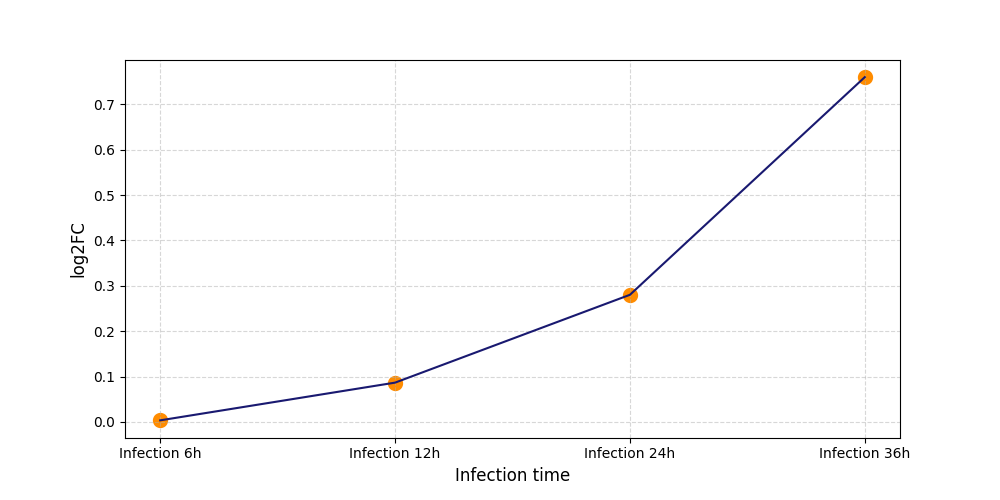
|
T741
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Sequence Information |
MRSAAAAPRSPAVATESRRFAAARWPGWRSLQRPARRSGRGGGGAAPGPYPSAAPPPPGPGPPPSRQSSPPSASDCFGSNGNGGGAFRPGSRRLLGLGGPPRPFVVLLLPLASPGAPPAAPTRASPLGARASPPRSGVSLARPAPGCPRPACEPVYGPLTMSLKPQQQQQQQQQQQQQQQQQQQQQQQPPPAAANVRKPGGSGLLASPAAAPSPSSSSVSSSSATAPSSVVAATSGGGRPGLGRGRNSNKGLPQSTISFDGIYANMRMVHILTSVVGSKCEVQVKNGGIYEGVFKTYSPKCDLVLDAAHEKSTESSSGPKREEIMESILFKCSDFVVVQFKDMDSSYAKRDAFTDSAISAKVNGEHKEKDLEPWDAGELTANEELEALENDVSNGWDPNDMFRYNEENYGVVSTYDSSLSSYTVPLERDNSEEFLKREARANQLAEEIESSAQYKARVALENDDRSEEEKYTAVQRNSSEREGHSINTRENKYIPPGQRNREVISWGSGRQNSPRMGQPGSGSMPSRSTSHTSDFNPNSGSDQRVVNGGVPWPSPCPSPSSRPPSRYQSGPNSLPPRAATPTRPPSRPPSRPSRPPSHPSAHGSPAPVSTMPKRMSSEGPPRMSPKAQRHPRNHRVSAGRGSISSGLEFVSHNPPSEAATPPVARTSPSGGTWSSVVSGVPRLSPKTHRPRSPRQNSIGNTPSGPVLASPQAGIIPTEAVAMPIPAASPTPASPASNRAVTPSSEAKDSRLQDQRQNSPAGNKENIKPNETSPSFSKAENKGISPVVSEHRKQIDDLKKFKNDFRLQPSSTSESMDQLLNKNREGEKSRDLIKDKIEPSAKDSFIENSSSNCTSGSSKPNSPSISPSILSNTEHKRGPEVTSQGVQTSSPACKQEKDDKEEKKDAAEQVRKSTLNPNAKEFNPRSFSQPKPSTTPTSPRPQAQPSPSMVGHQQPTPVYTQPVCFAPNMMYPVPVSPGVQPLYPIPMTPMPVNQAKTYRAVPNMPQQRQDQHHQSAMMHPASAAGPPIAATPPAYSTQYVAYSPQQFPNQPLVQHVPHYQSQHPHVYSPVIQGNARMMAPPTHAQPGLVSSSATQYGAHEQTHAMYACPKLPYNKETSPSFYFAISTGSLAQQYAHPNATLHPHTPHPQPSATPTGQQQSQHGGSHPAPSPVQHHQHQAAQALHLASPQQQSAIYHAGLAPTPPSMTPASNTQSPQNSFPAAQQTVFTIHPSHVQPAYTNPPHMAHVPQAHVQSGMVPSHPTAHAPMMLMTTQPPGGPQAALAQSALQPIPVSTTAHFPYMTHPSVQAHHQQQL
Click to Show/Hide
|
|---|