Details of Host Protein
| Host Protein General Information (ID: PT0639) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name |
Microtubule-associated protein 1B (MAP1B)
|
Gene Name |
MAP1B
|
||||||
| Host Species |
Homo sapiens
|
Uniprot Entry Name |
MAP1B_HUMAN
|
||||||
| Protein Families |
MAP1 family
|
||||||||
| Subcellular Location |
Cytoplasm; synapse Cell projection; dendritic spine
|
||||||||
| External Link | |||||||||
| NCBI Gene ID | |||||||||
| Uniprot ID | |||||||||
| Ensembl ID | |||||||||
| HGNC ID | |||||||||
| Function in Host |
Facilitates tyrosination of alpha-tubulin in neuronalmicrotubules. Phosphorylated MAP1B may play a role inthe cytoskeletal changes that accompany neurite extension. PossiblyMAP1B binds to at least two tubulin subunits in the polymer, and thisbridging of subunits might be involved in nucleating microtubulepolymerization and in stabilizing microtubules. Acts as a positivecofactor in DAPK1-mediated autophagic vesicle formation and membraneblebbing.
[1]
Click to Show/Hide
|
||||||||
| Related KEGG Pathway | |||||||||
| Viral life cycle - HIV-1 | hsa03250 |
Pathway Map 
|
|||||||
| 3D Structure |
|
||||||||
| Function of This Protein During Virus Infection | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Virus Name | SARS-COV-2 | Protein Function | Pro-viral | [2] | |||||
| Infected Tissue | Lung | Infection Time | 7-9 Days | ||||||
| Infected Cell | Calu-3 Cells (Human epithelial cell line) | Cellosaurus ID | CVCL_0609 | ||||||
| Method Description | To detect the role of host protein MAP1B in viral infection, MAP1B protein knockout Calu-3 Cells were infected with SARS-COV-2 for 7 - 9 Days , and the effects on infection was detected through CRISPR-based genome-wide gene-knockout screen. | ||||||||
| Results | It is reported that knockout of MAP1B leads to the decreased SARS-CoV-2 RNA levels compared with control group. | ||||||||
| Full List of Virus RNA Interacting with This Protien | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| RNA Region: 3'-UTR (hCoV-19/Wuhan-Hu-1/2019 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[3] | |||||||
| Strains Name |
hCoV-19/Wuhan-Hu-1/2019
|
||||||||
| Strains Family |
Beta (B.1.351)
|
||||||||
| RNA Binding Region |
3'-UTR
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Infection Cells | HEK293 Cells (Human embryonic kidney cell) (CVCL_0045 ) | ||||||||
| Cell Originated Tissue | Liver | ||||||||
| Infection Time | 48 h | ||||||||
| Interaction Score | Prot score = 22 | ||||||||
| Method Description | RNA-protein interaction detection (RaPID) assay; liquid chromatography with tandem mass spectrometry (LC-MS/MS) | ||||||||
| Differential Gene Expression During SARS-COV-2 Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Phosphorylation after Virus Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
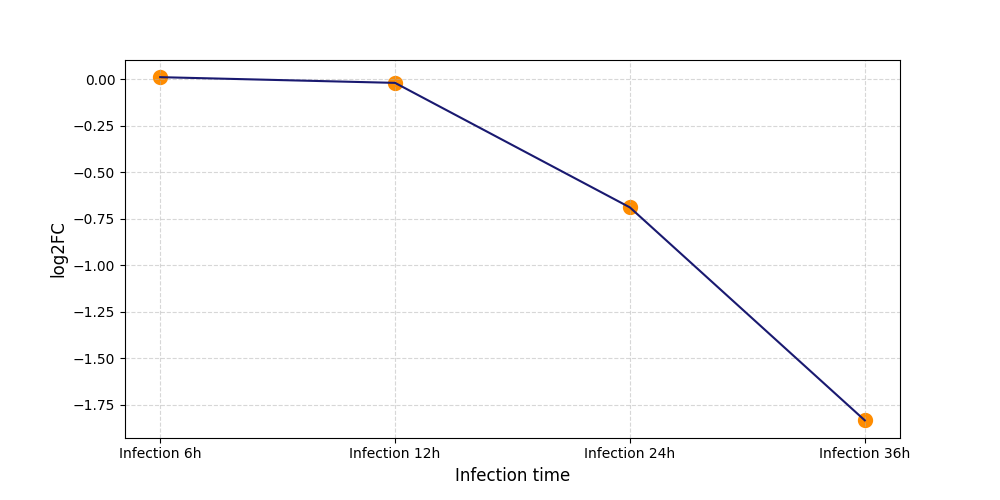
S1016
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
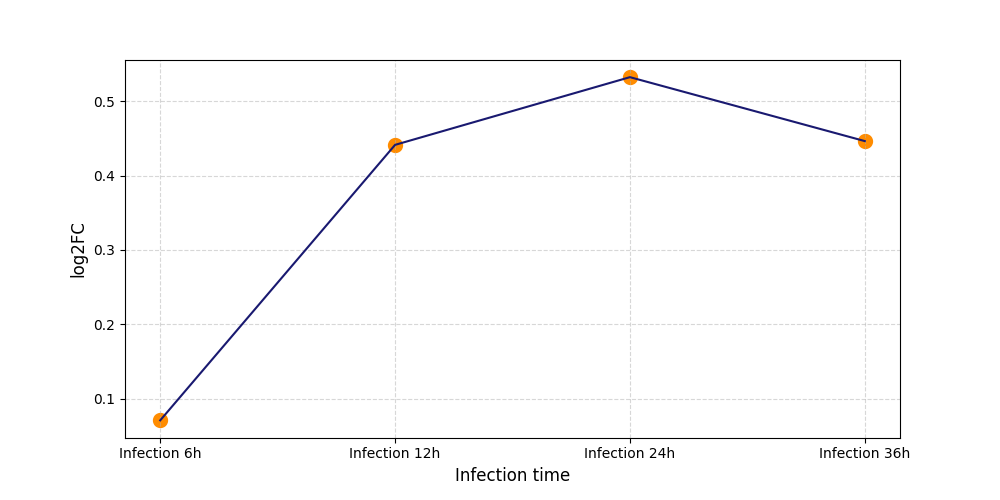
|
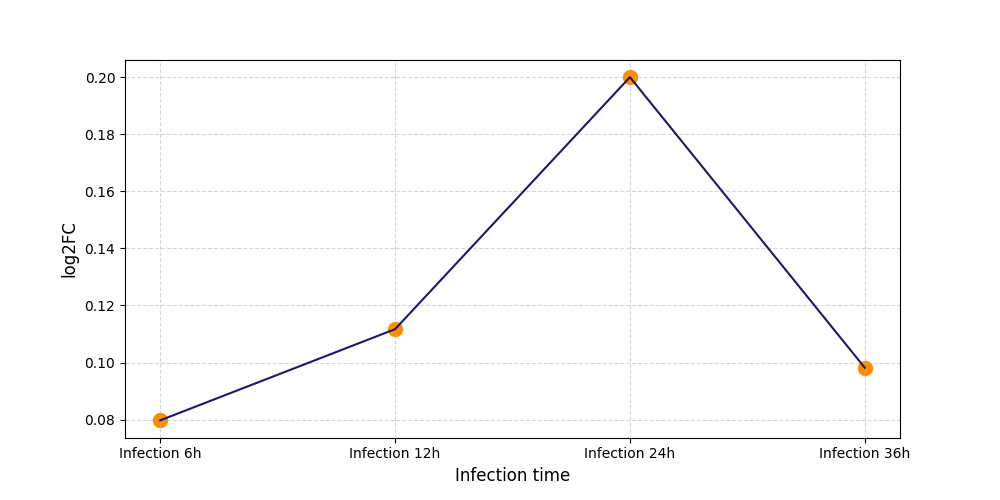
S1076
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
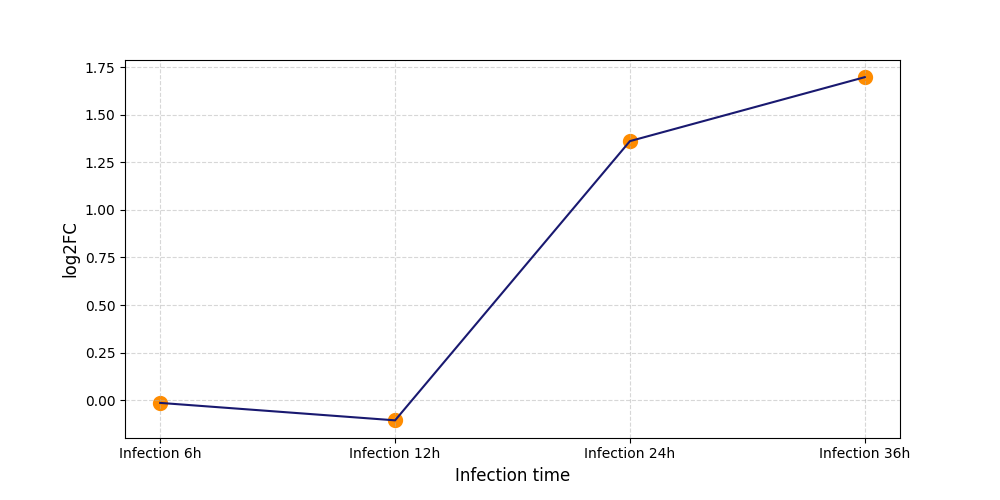
|
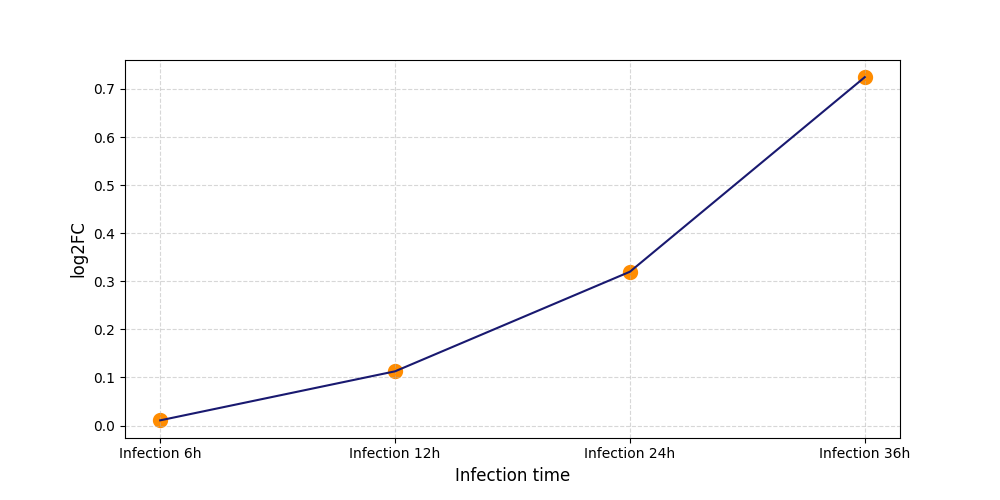
S1154
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
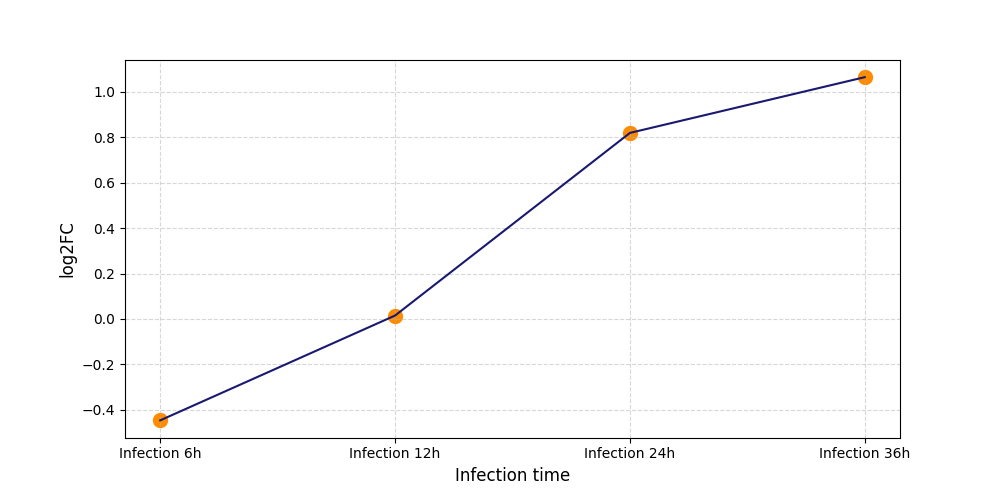
|
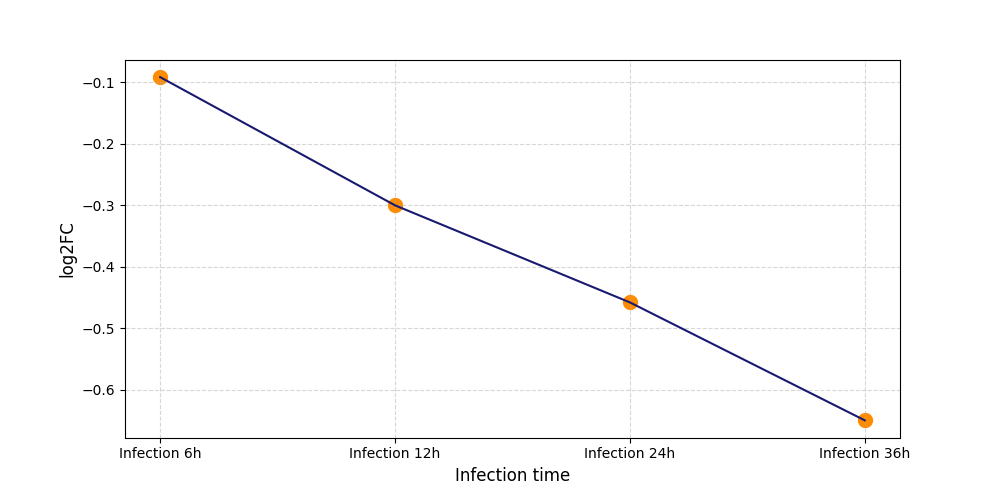
S1208
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
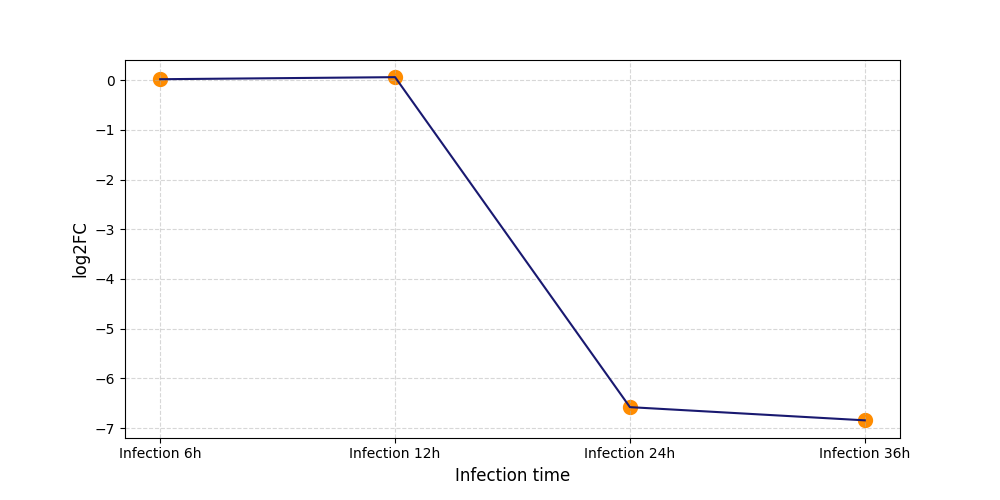
|
S1247
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1248
[4] |
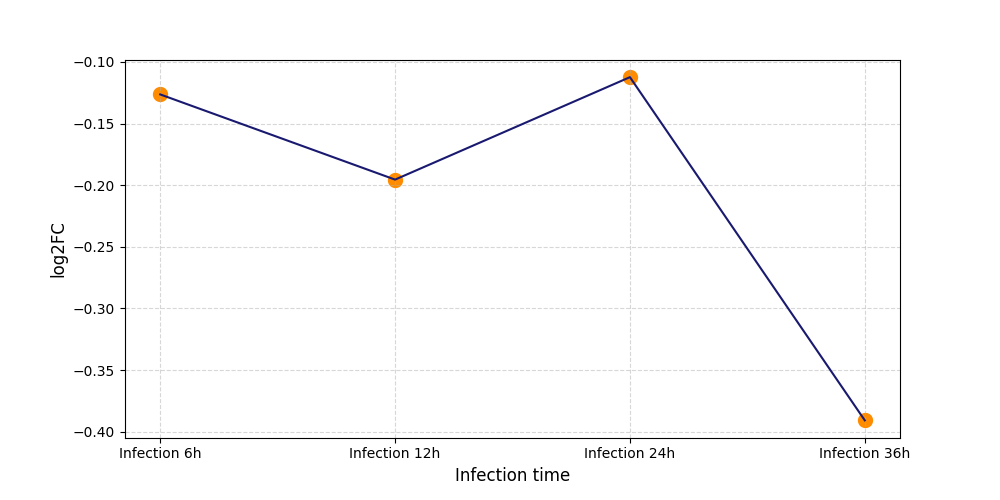 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1252
[4] |
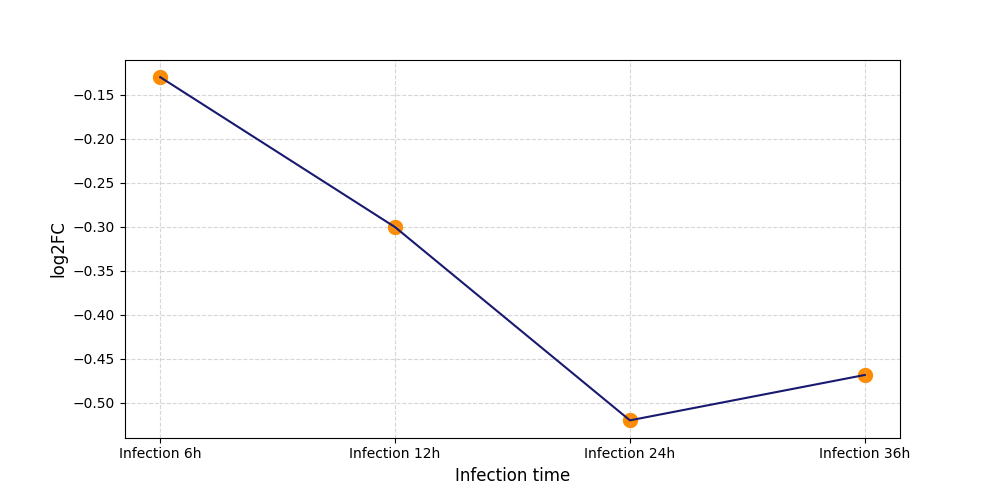 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1254
[4] |
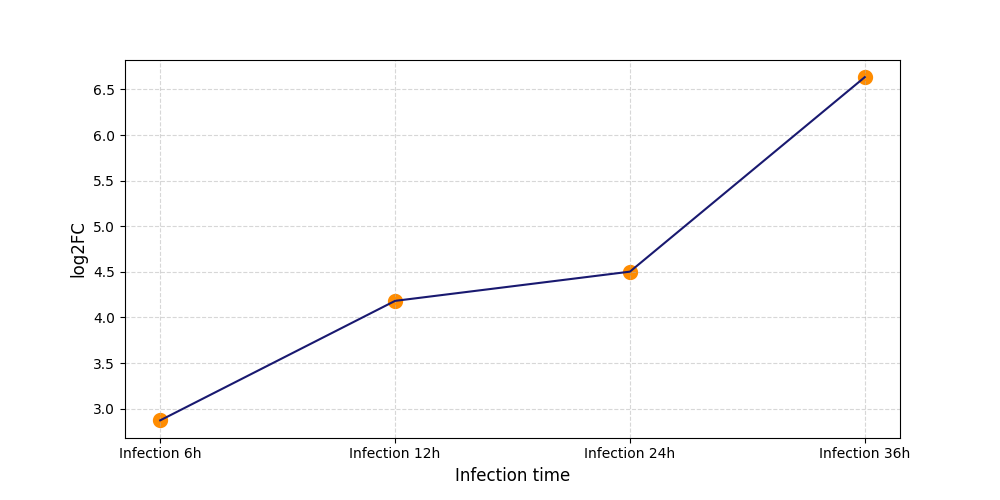 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1256
[4] |
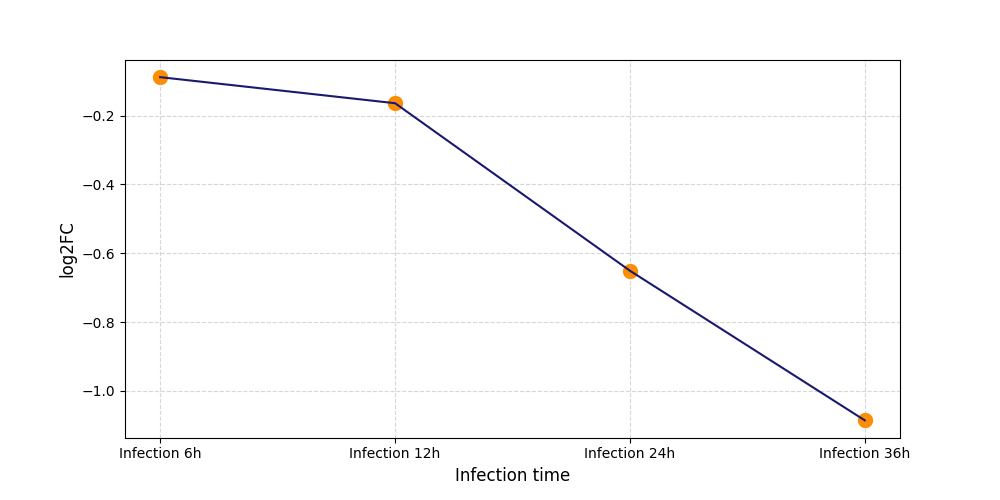 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1265
[5] |
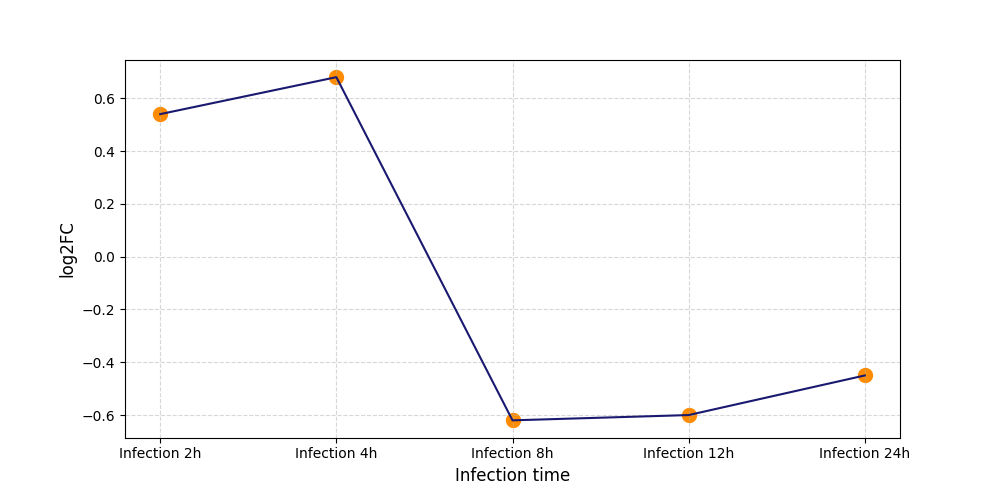 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1265
[4] |
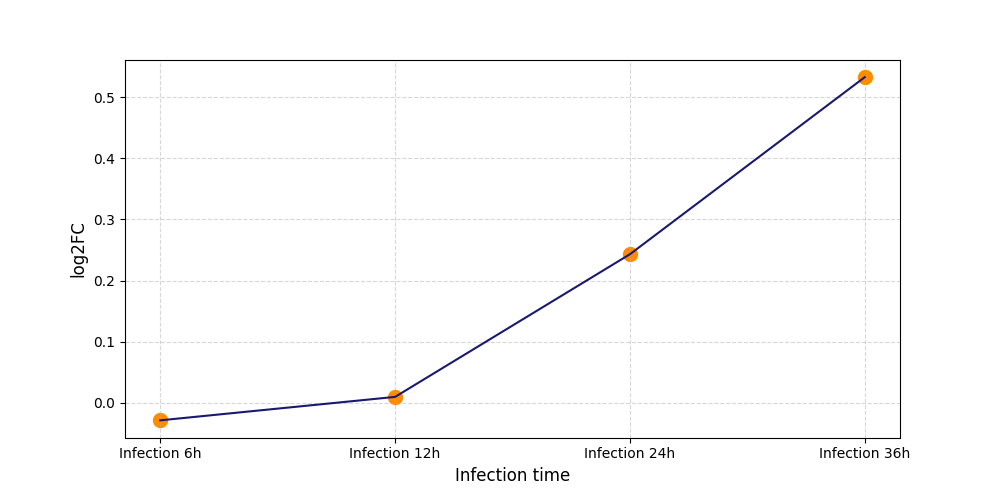 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1276
[4] |
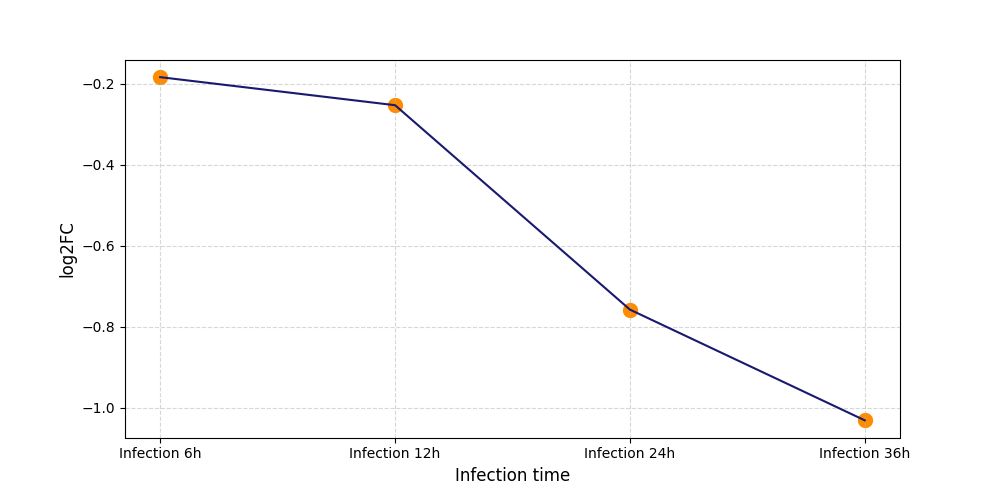 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1298
[4] |
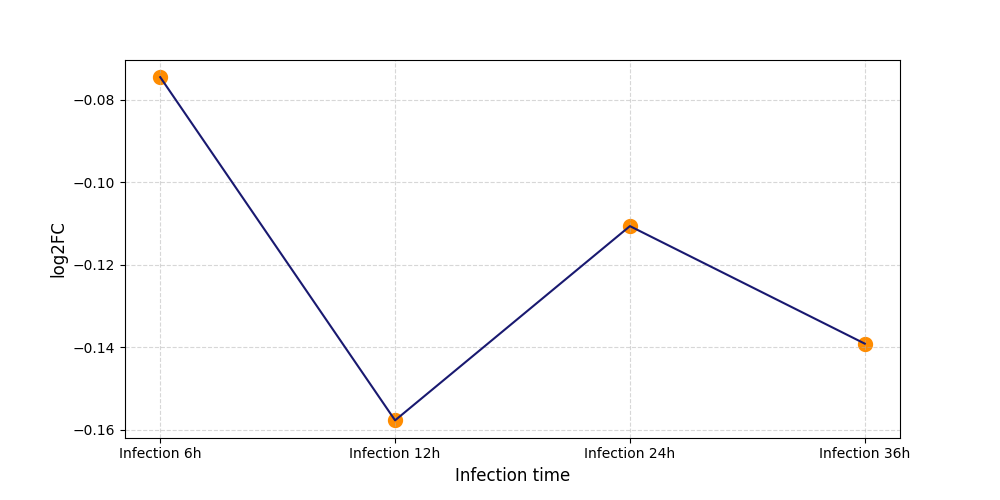 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
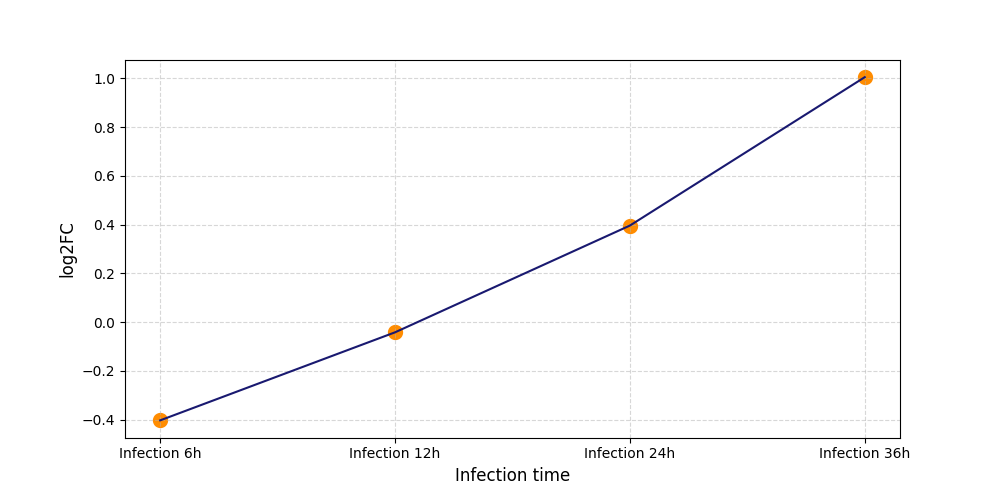
|
S1322
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
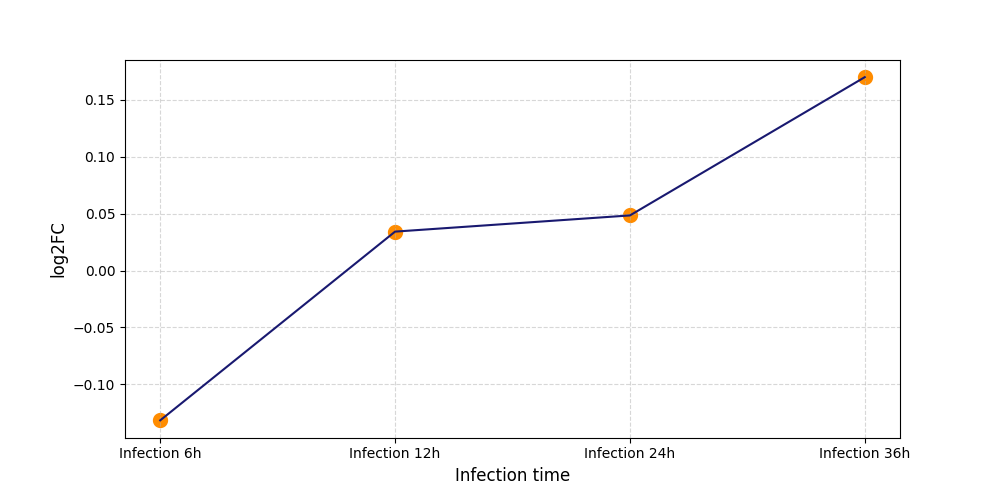
|
S1324
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
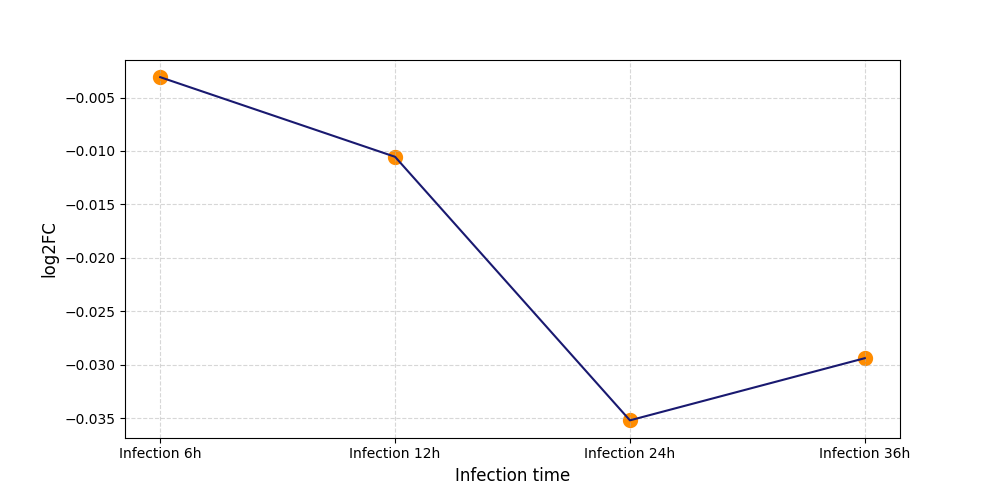
|
S1326
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
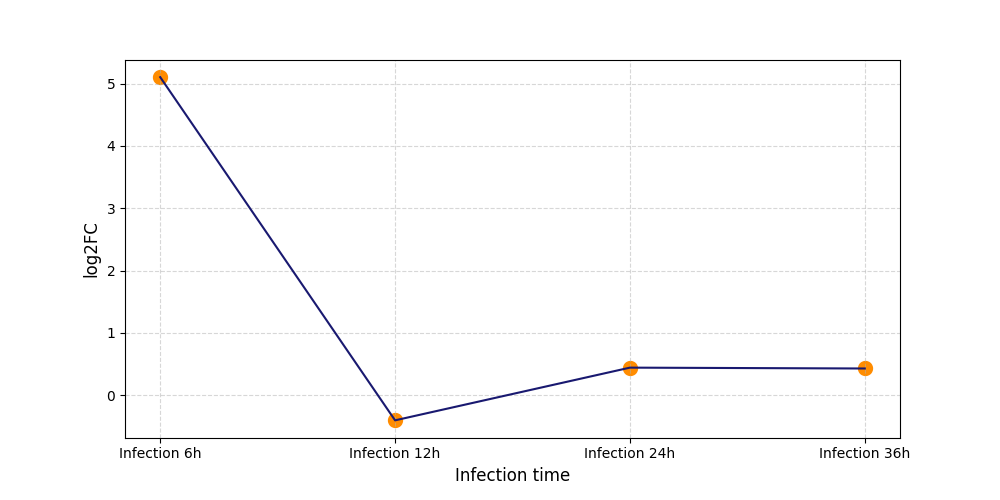
|
S1378
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
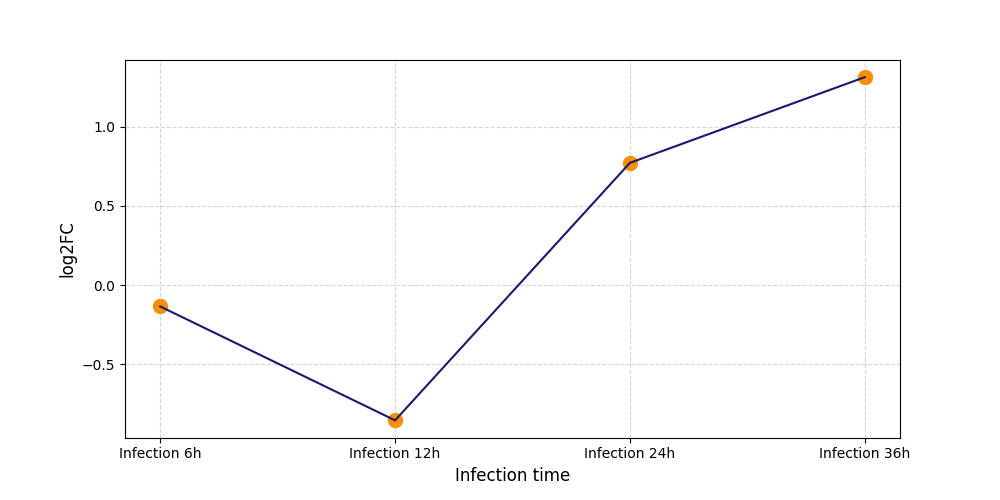
|
S1387
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
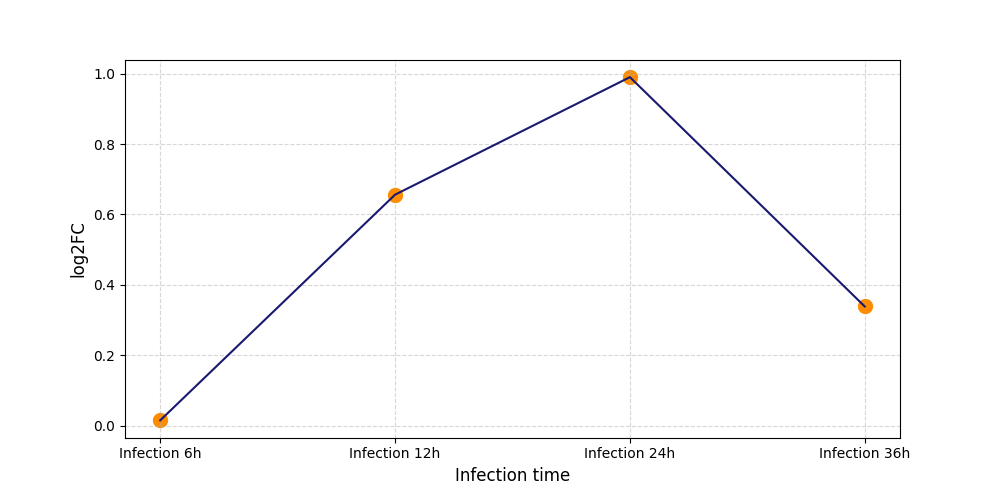
|
S1389
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
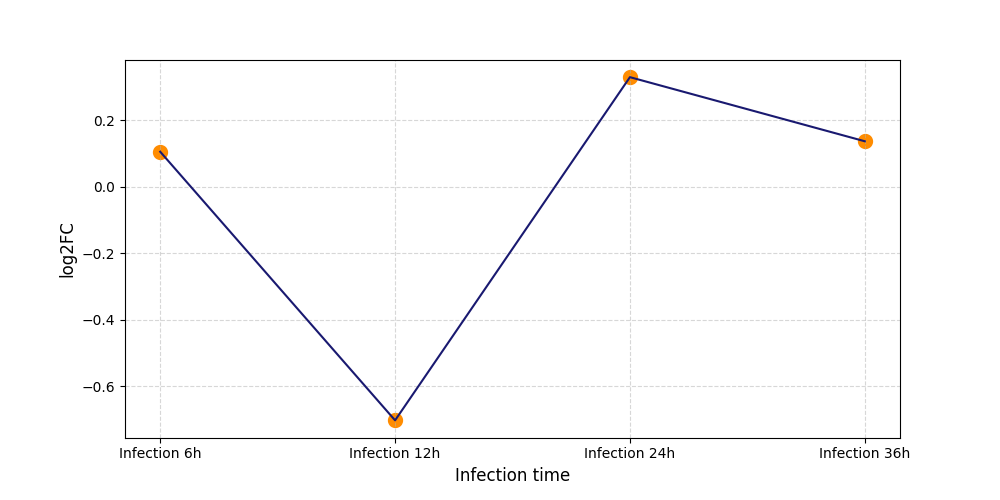
|
S1396
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
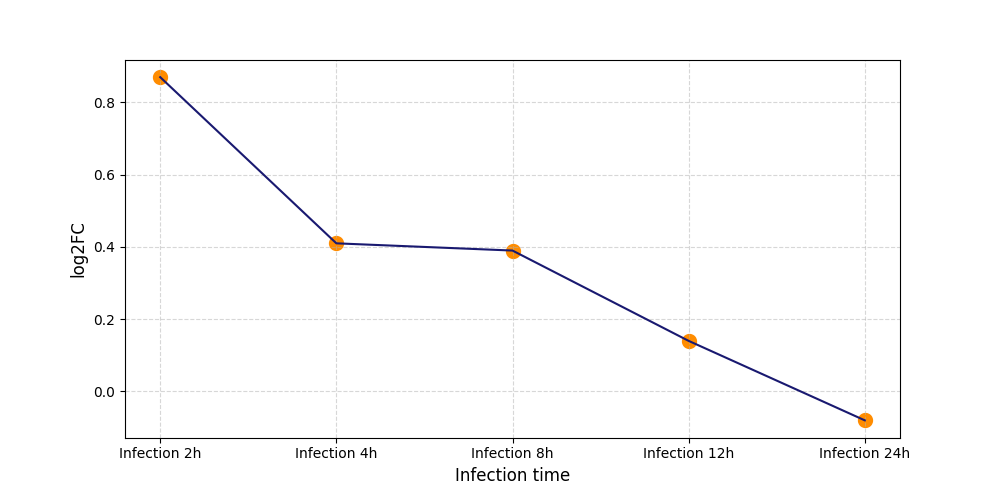
|
S1400
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1400
[4] |
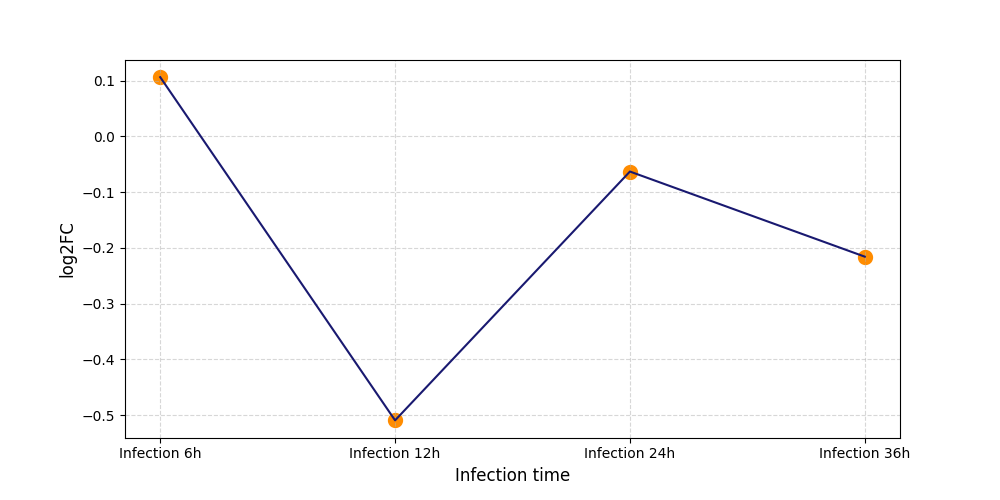 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1427
[5] |
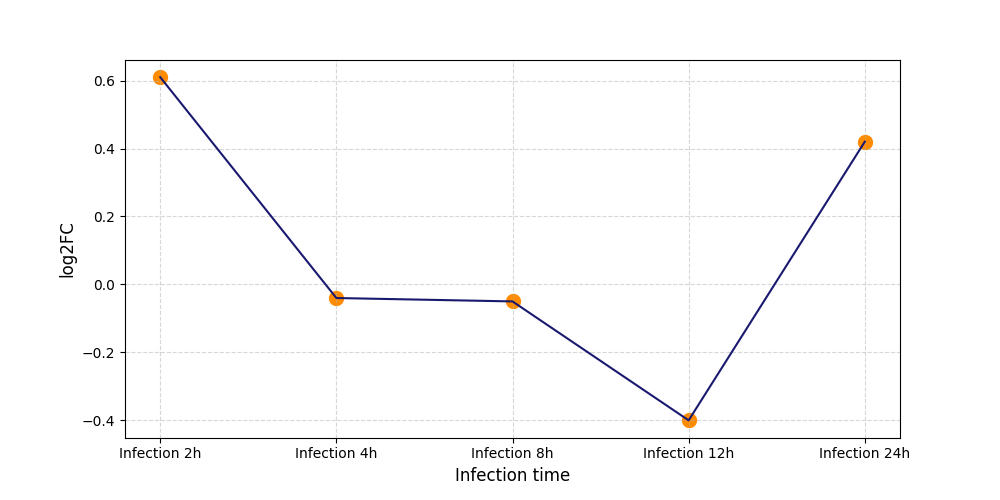 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1427
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1438
[4] |
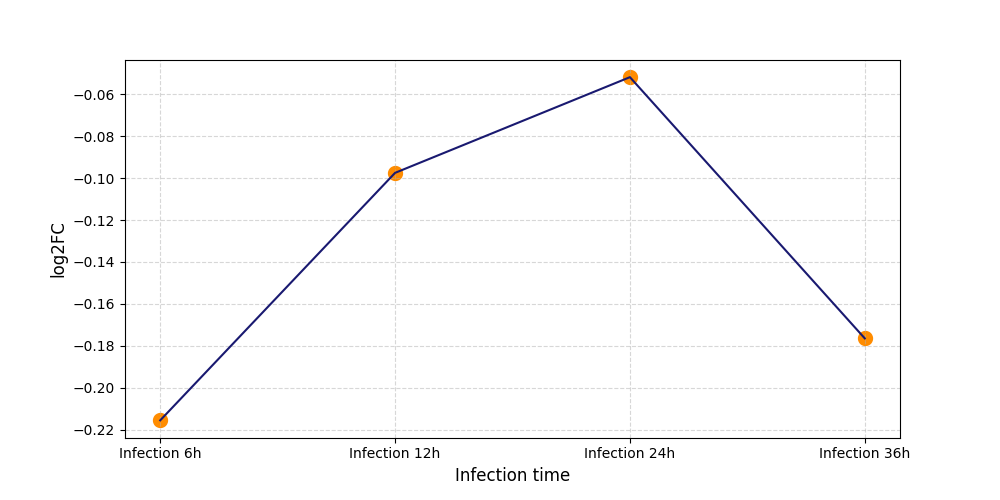 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1443
[4] |
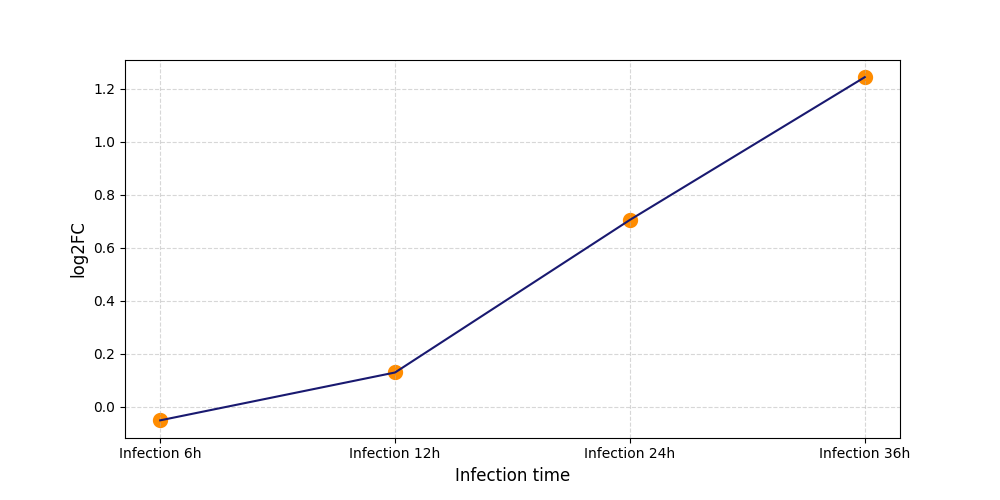 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
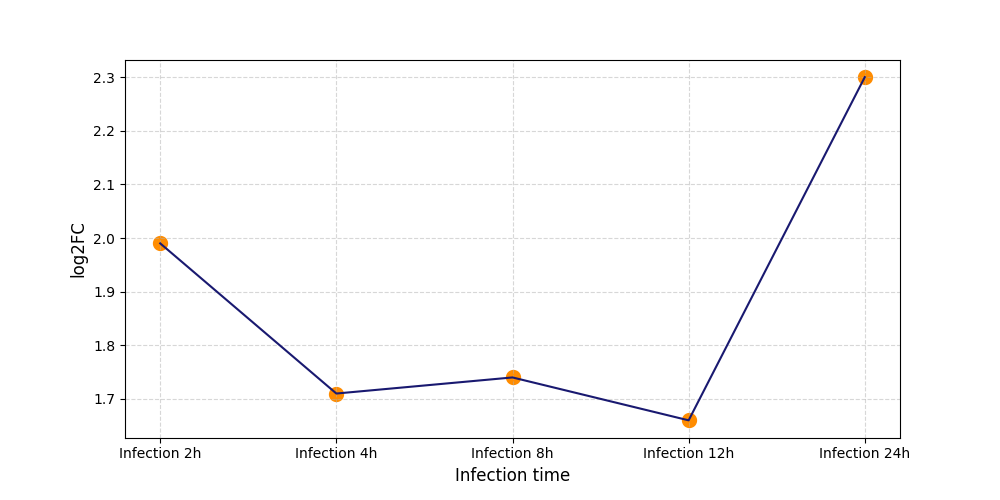
|
S1501
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
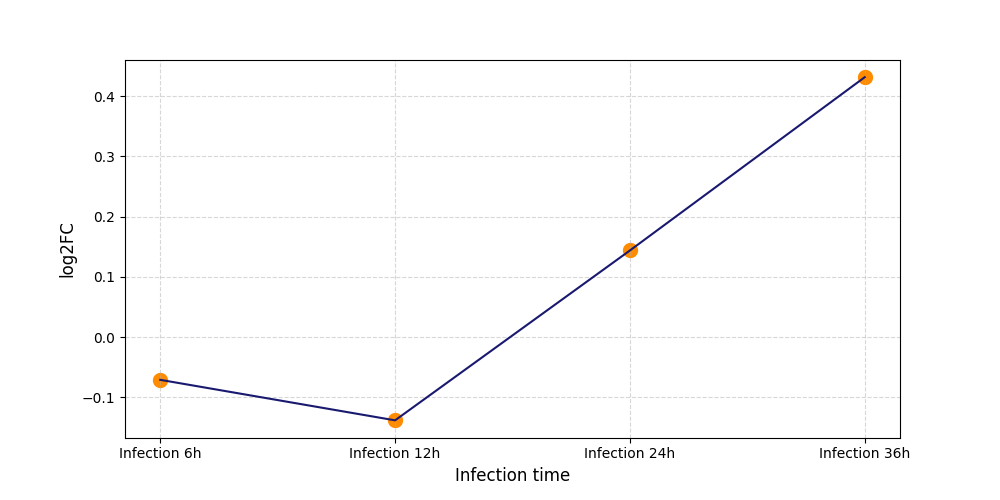
|
S1501
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
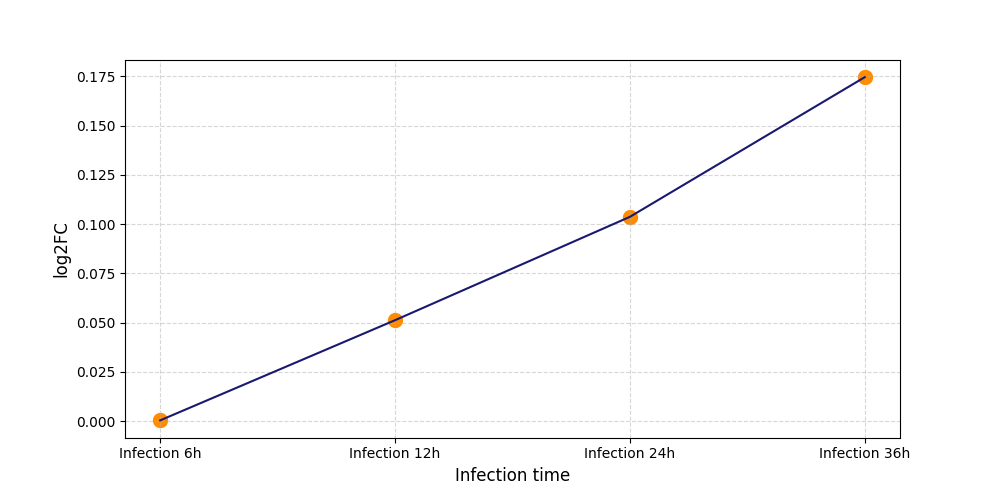
|
S1618
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
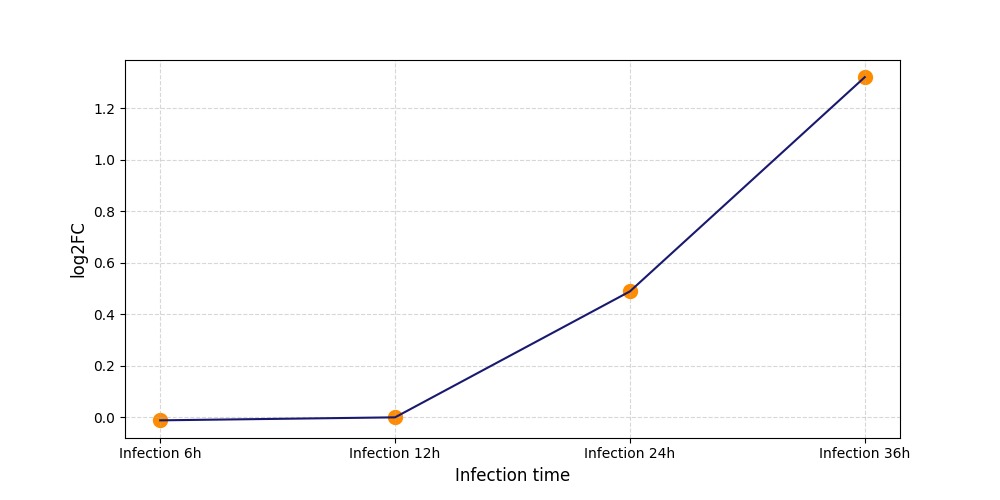
|
S1620
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1625
[4] |
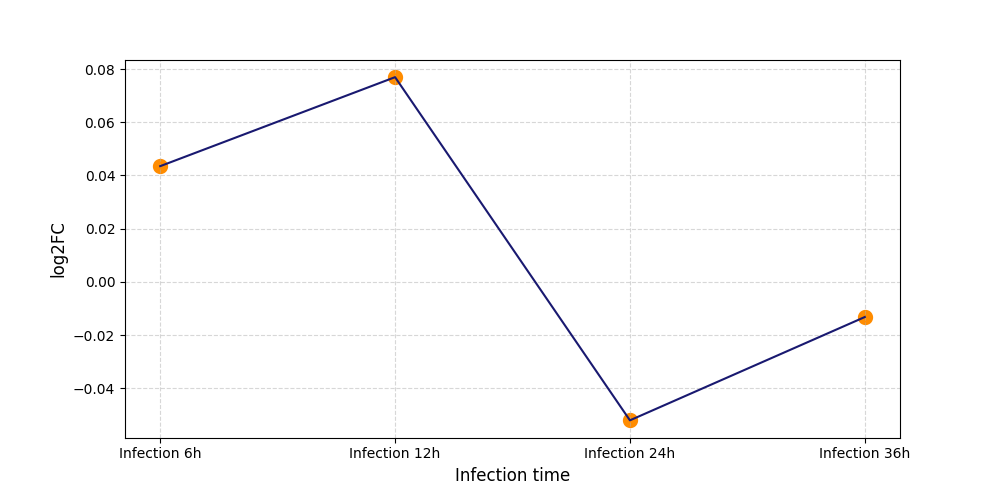 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1653
[5] |
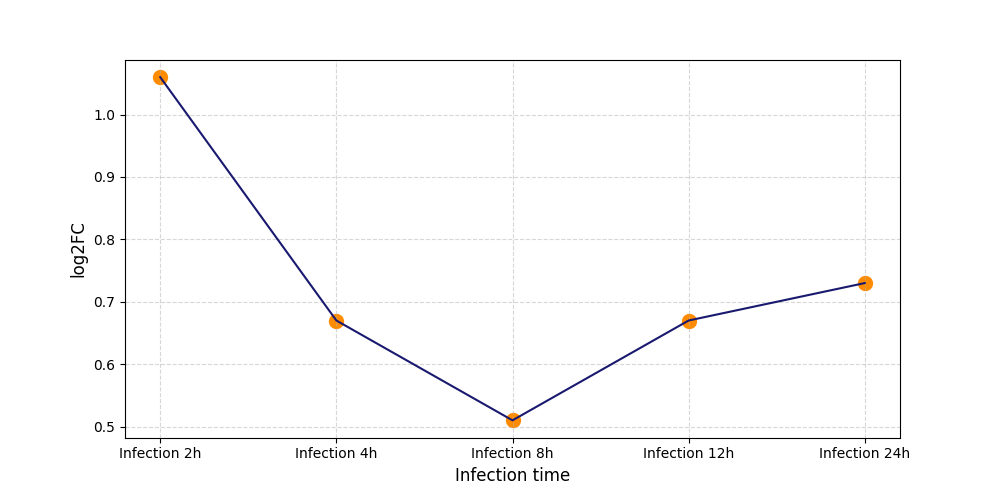 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1653
[4] |
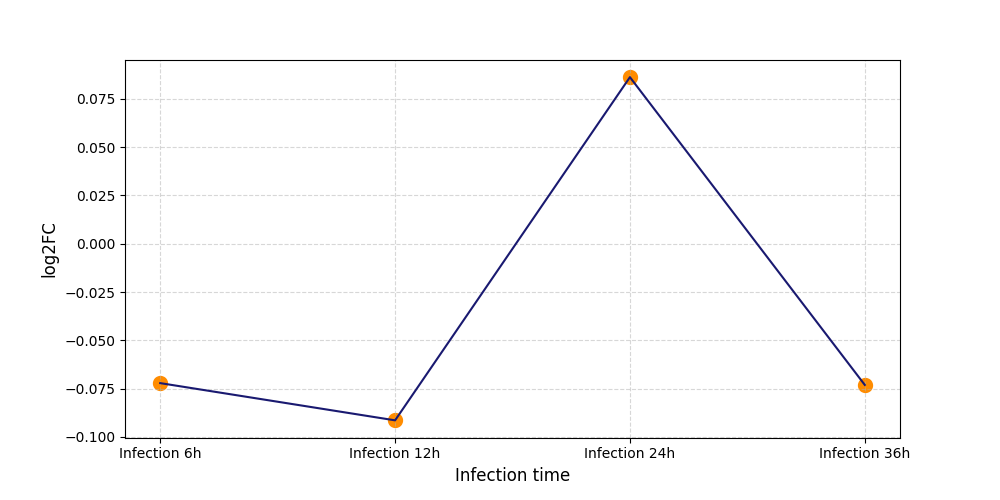 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1666
[4] |
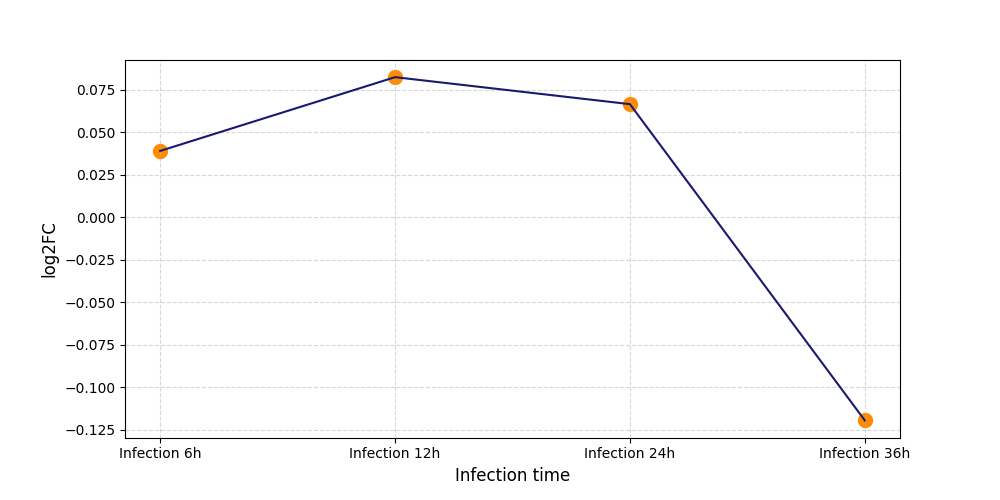 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1772
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1779
[5] |
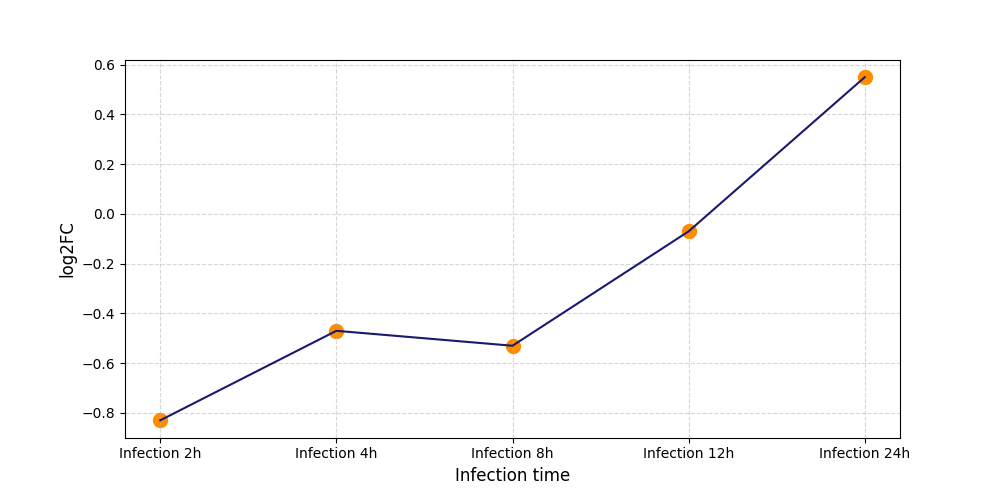 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1779
[4] |
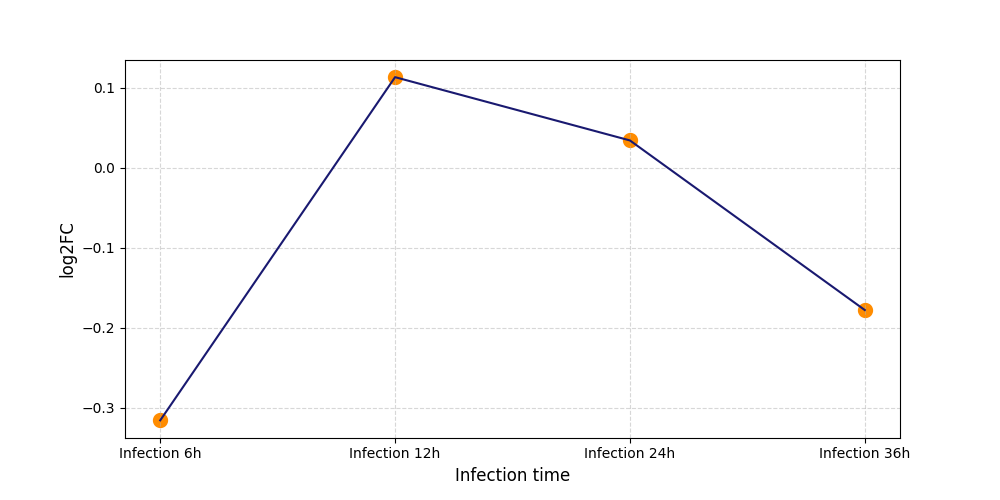 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1782
[4] |
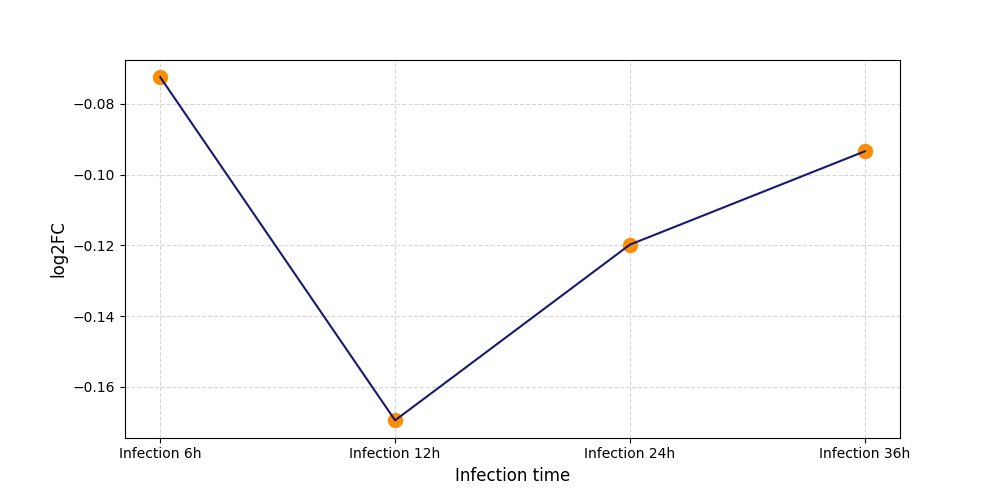 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1785
[5] |
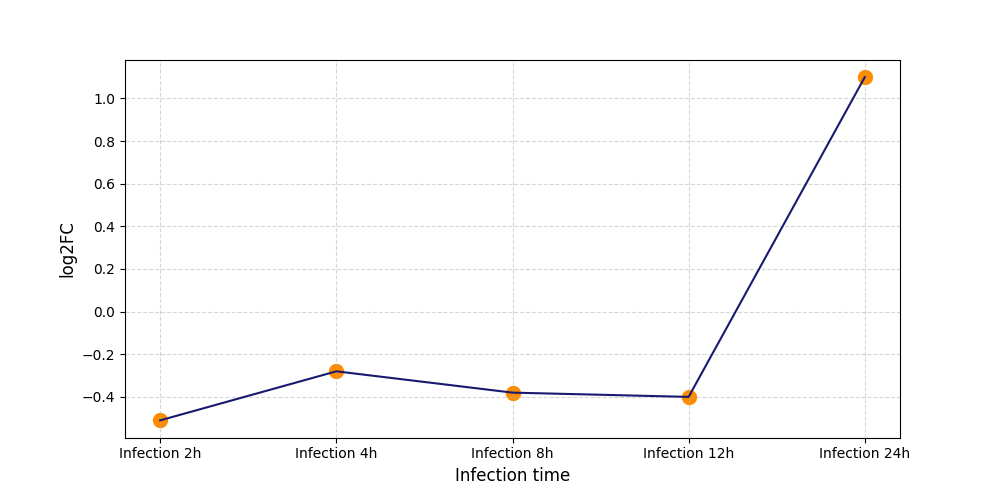 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
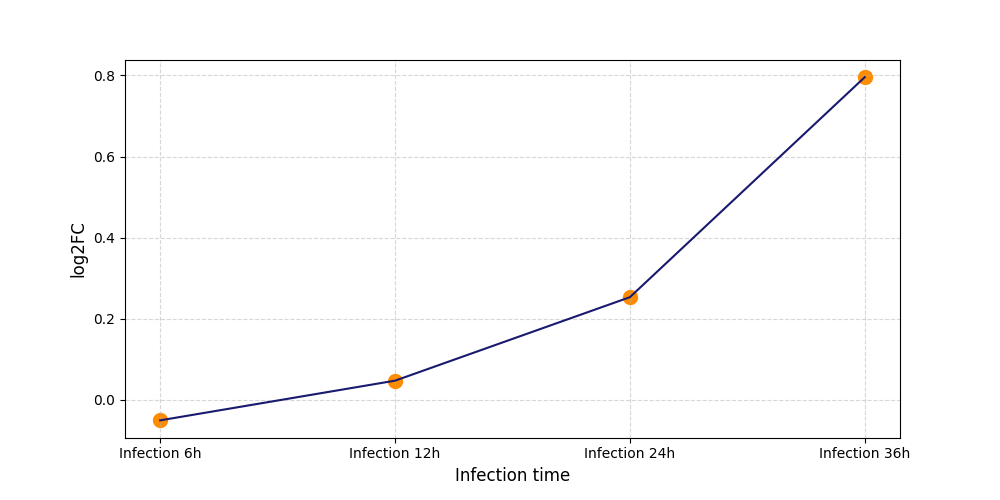
S1785
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
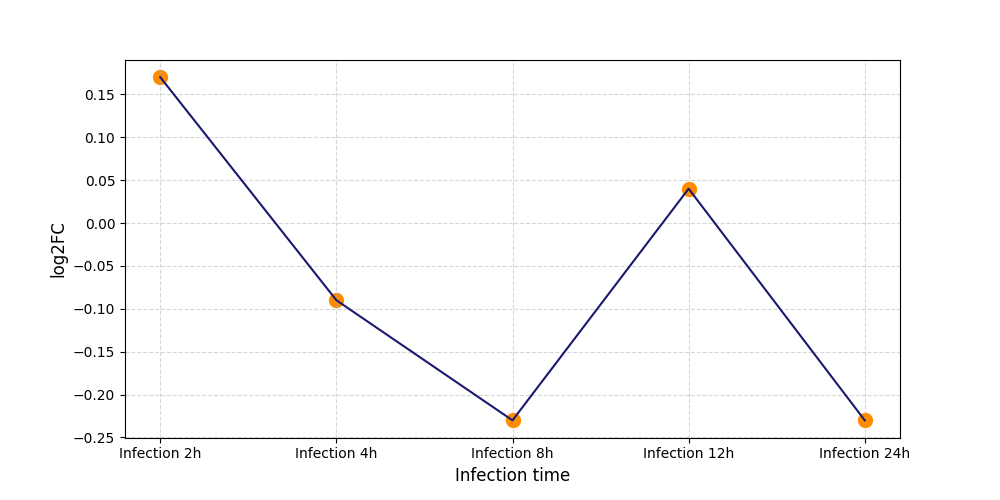
S1793
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
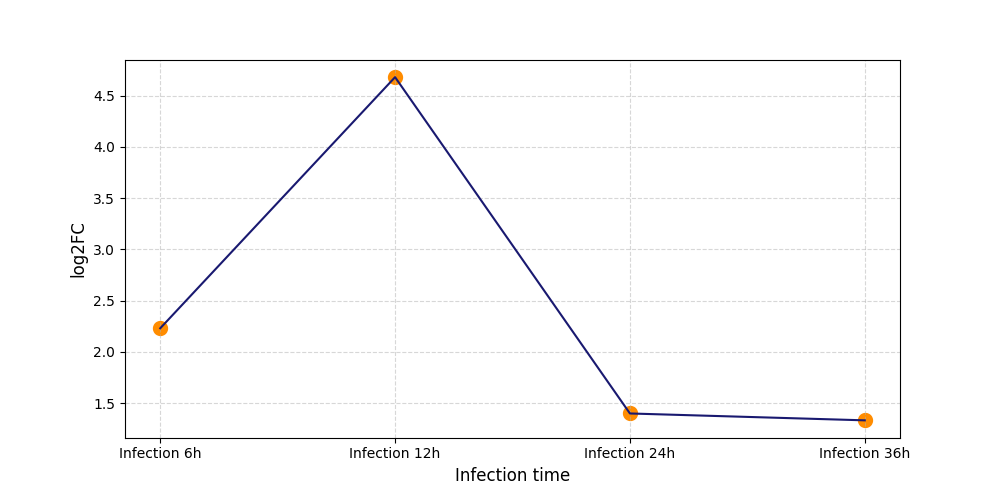
S1793
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1797
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
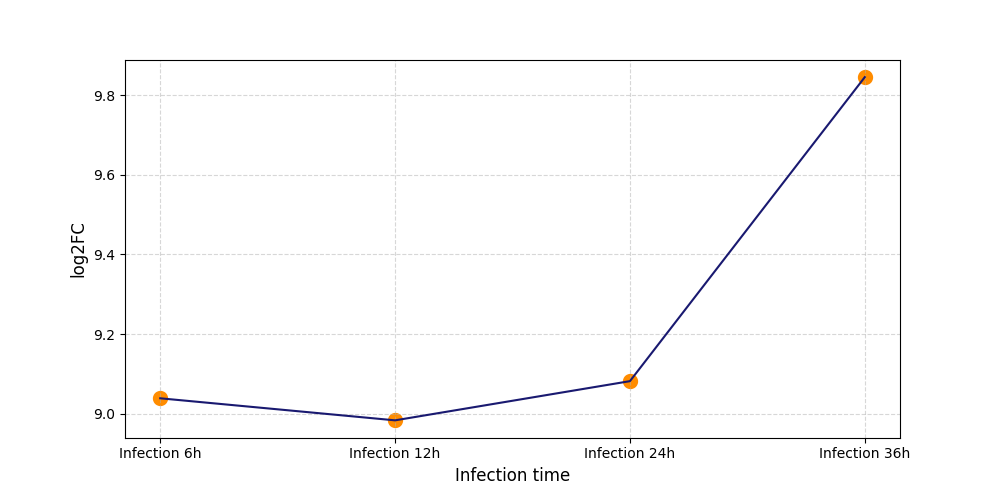
S1816
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
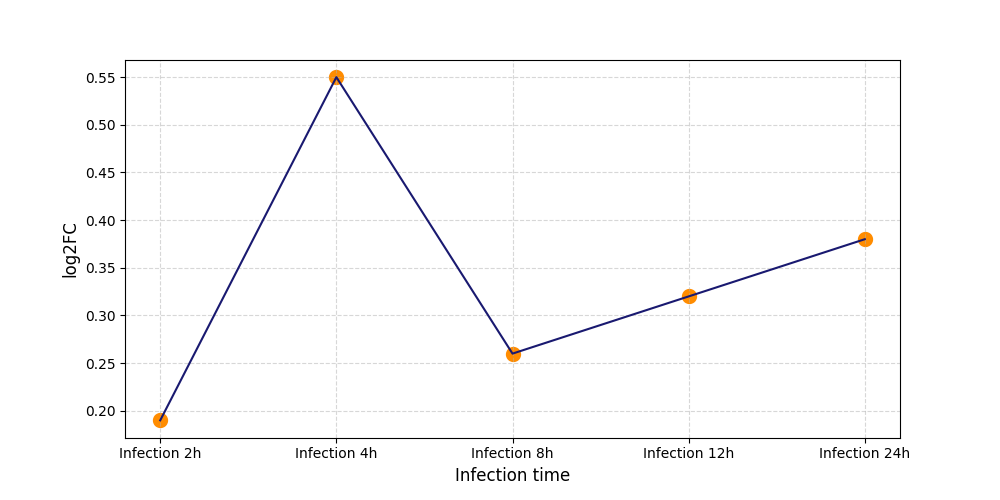
|
S1819
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
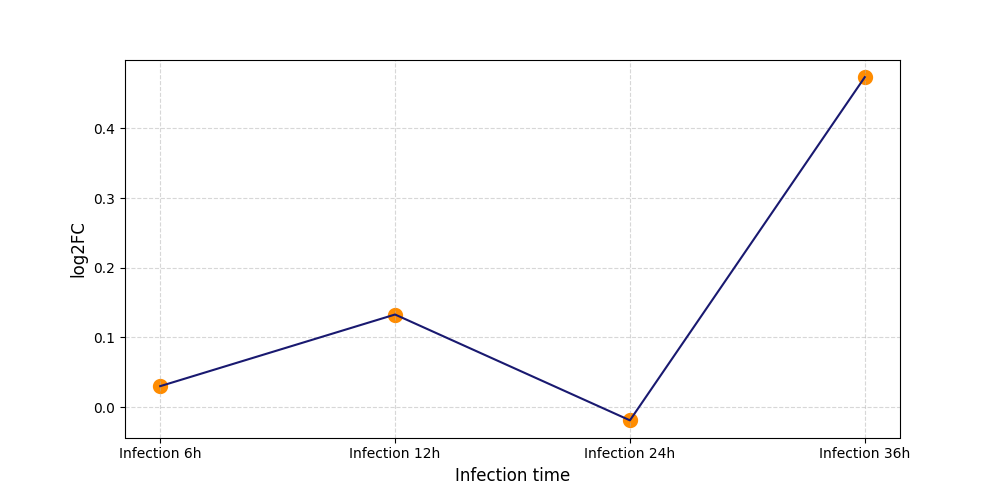
|
S1819
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
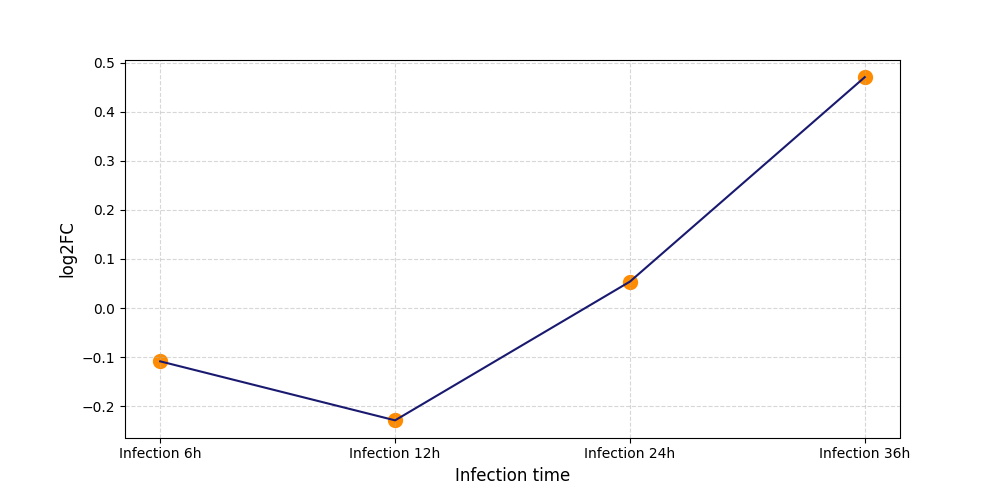
|
S1835
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
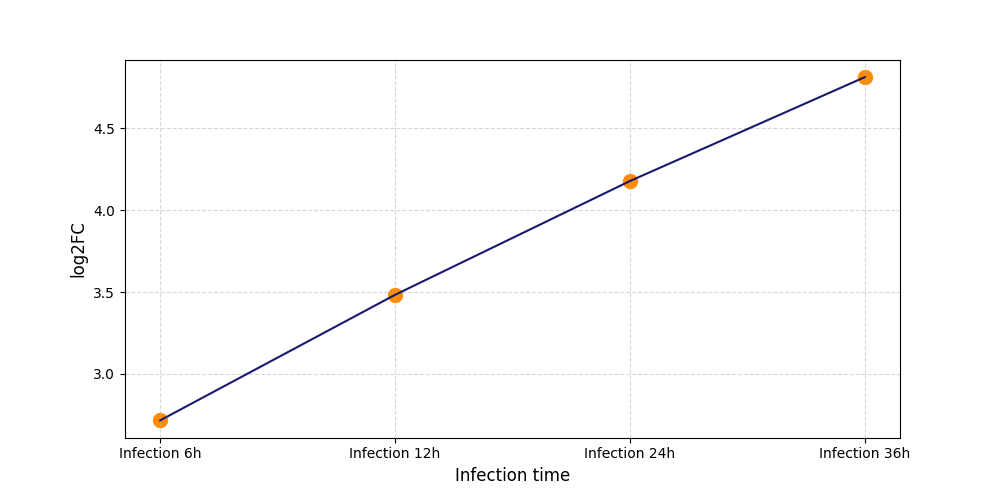
|
S1852
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1881
[5] |
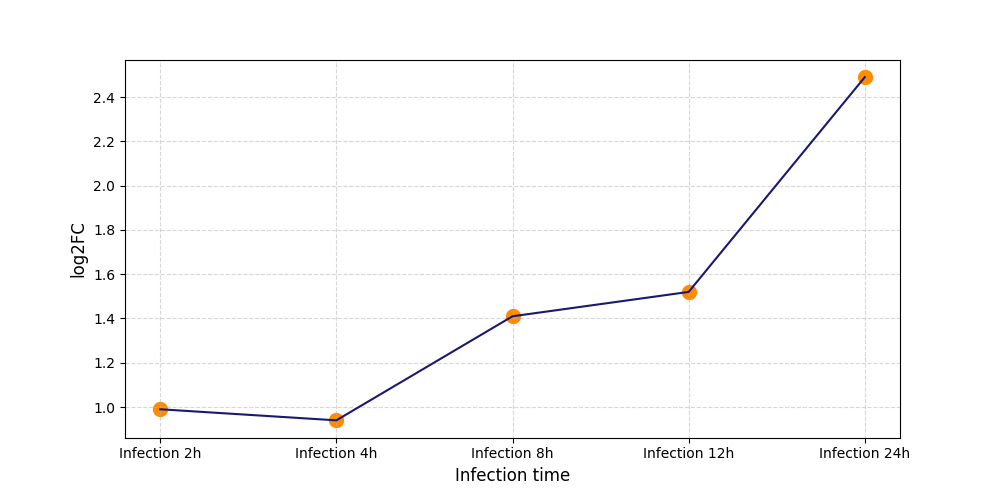 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1881
[4] |
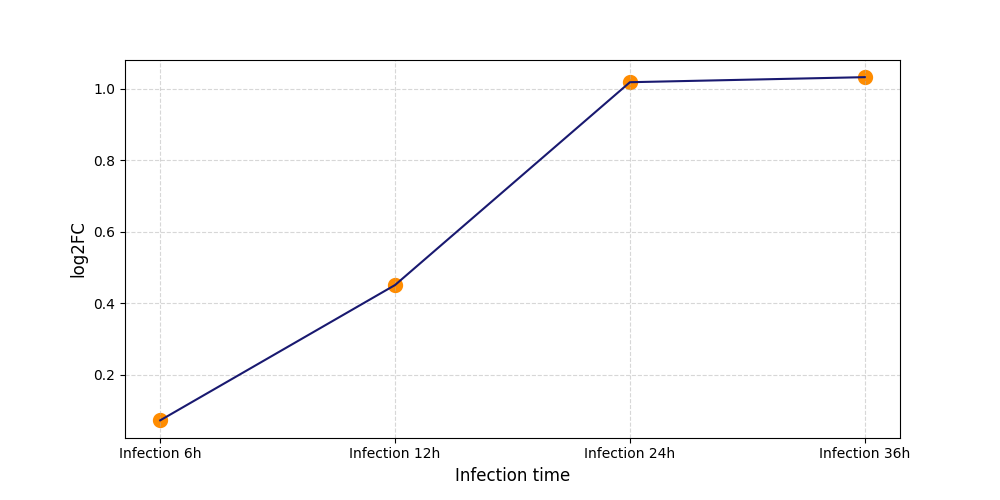 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1899
[4] |
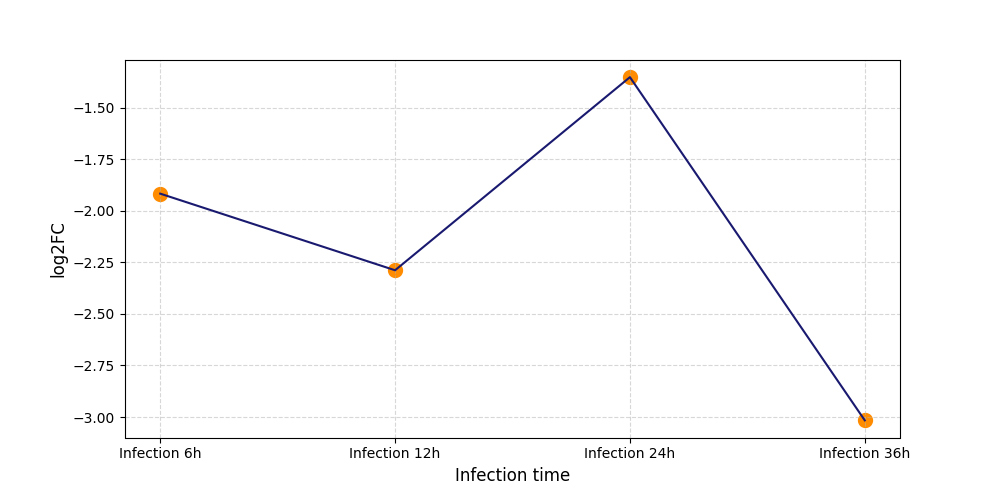 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1915
[5] |
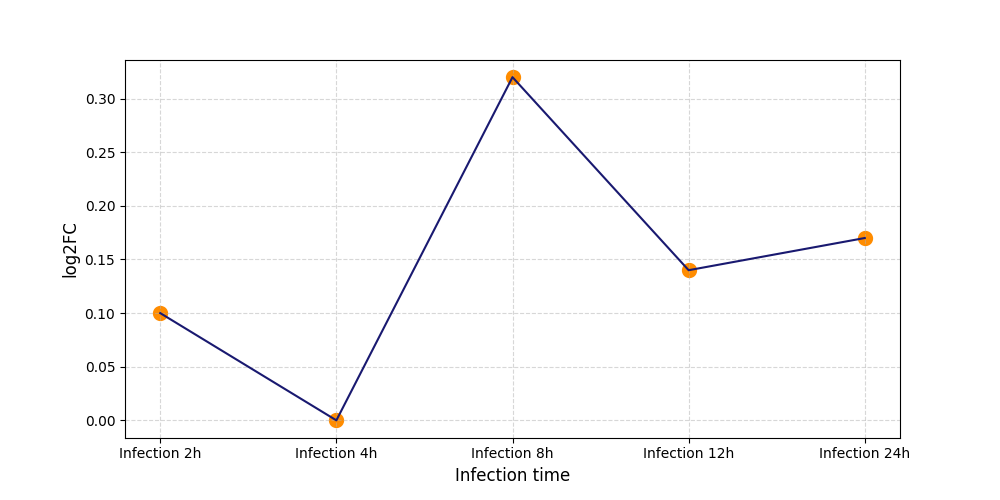 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
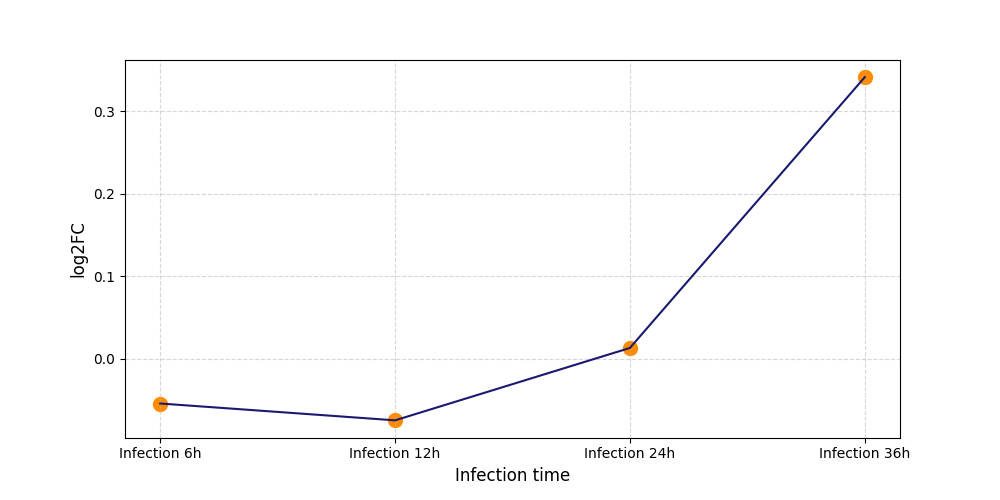
S1915
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
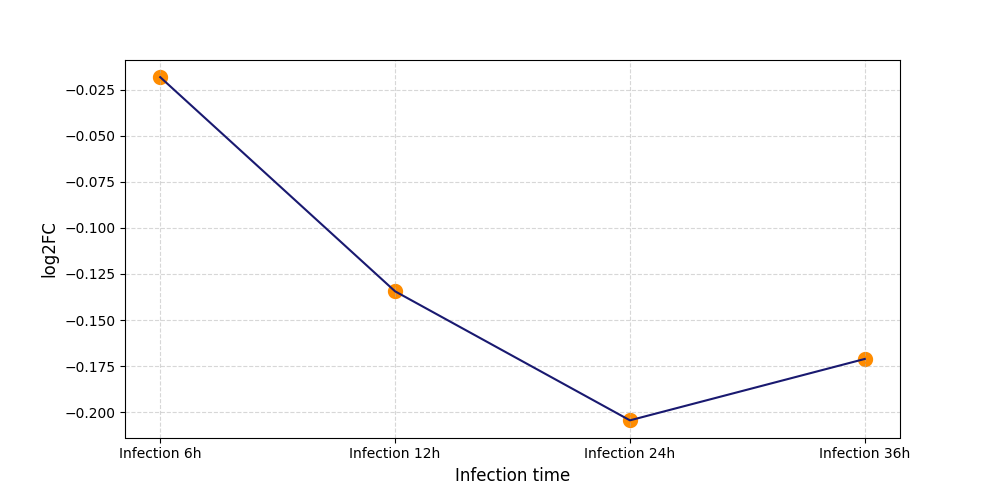
S1965
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
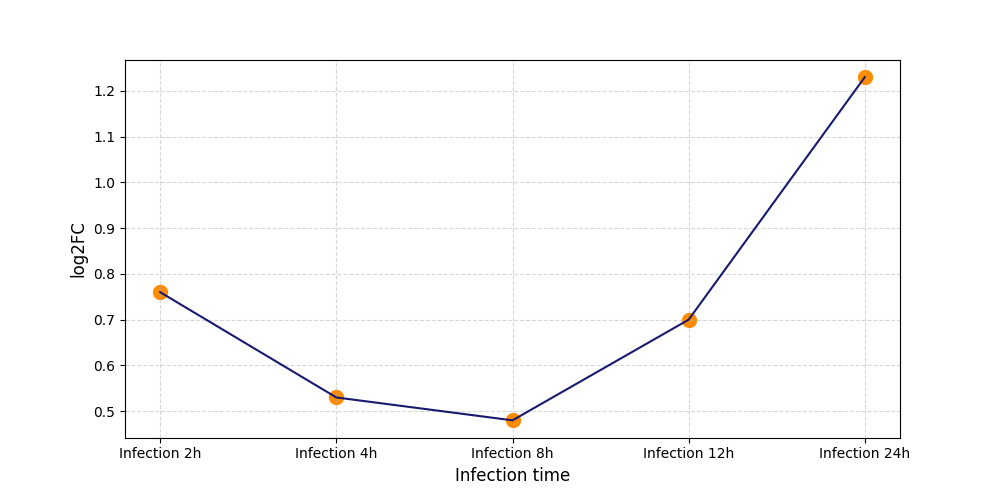
S2072
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
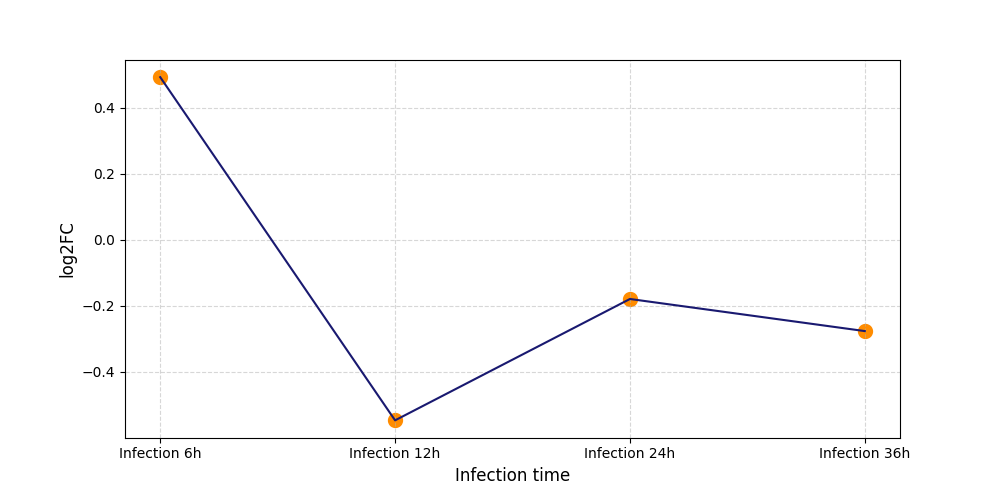
S2098
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2126
[4] |
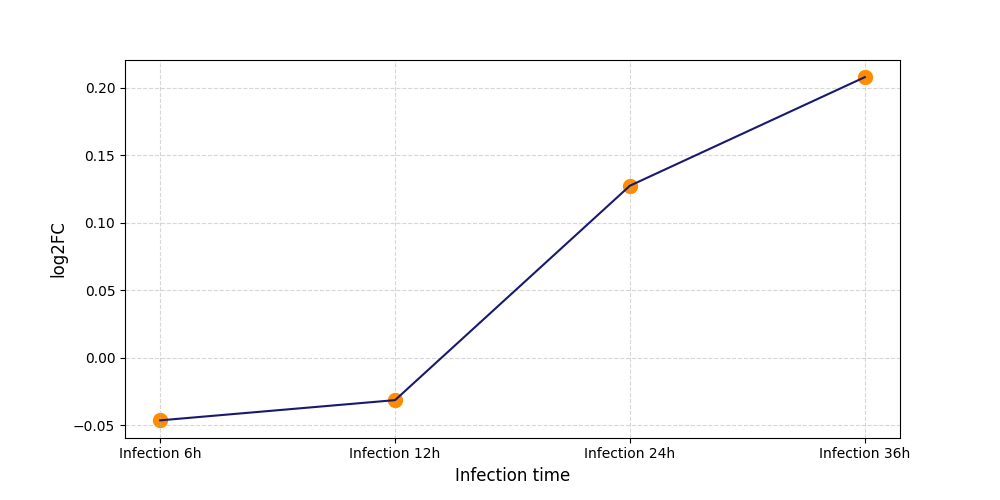 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2209
[4] |
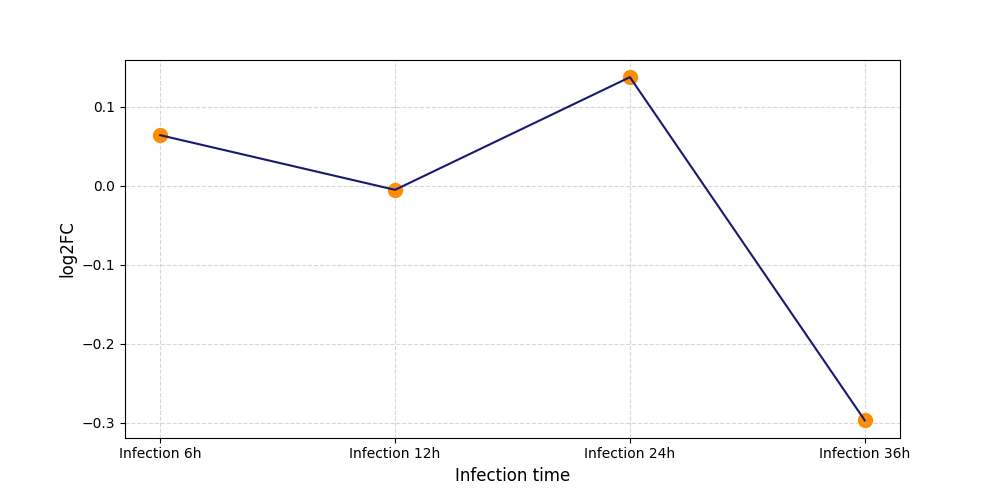 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2211
[4] |
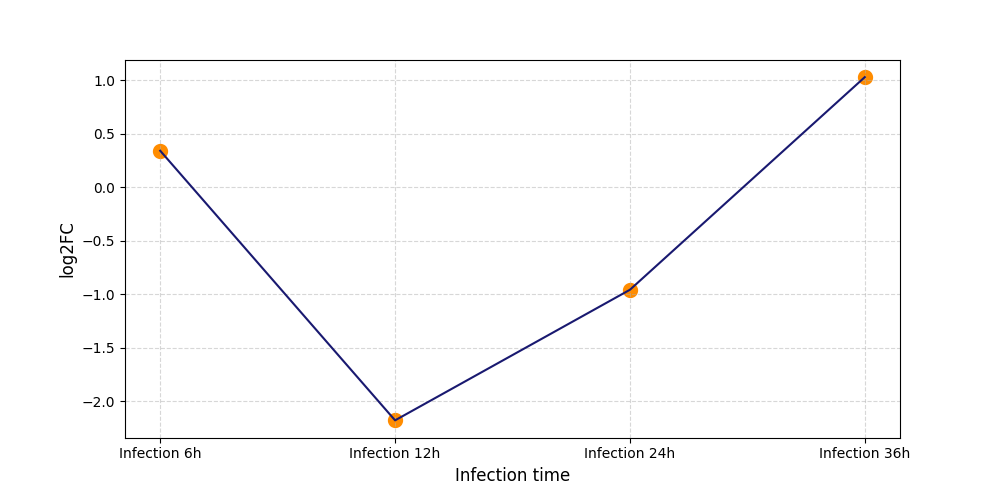 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2271
[4] |
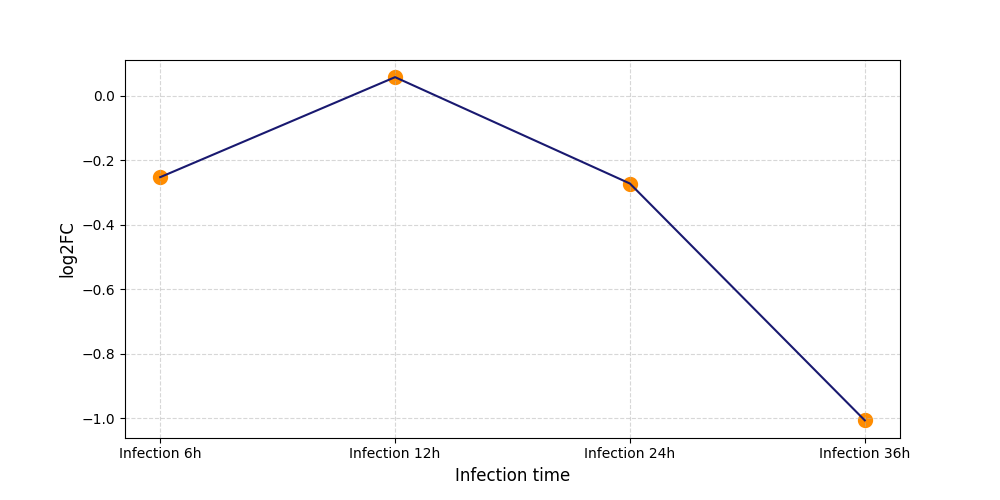 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
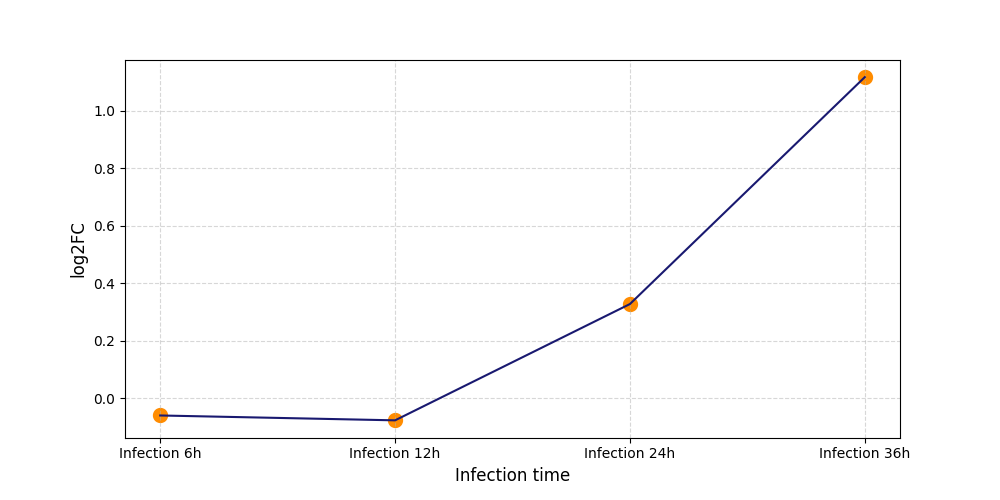
S25
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S339
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
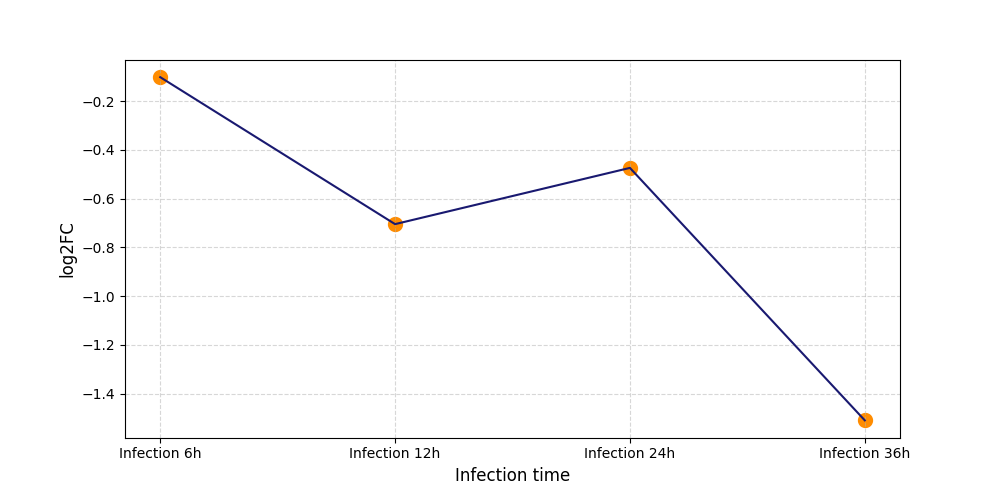
|
S541
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
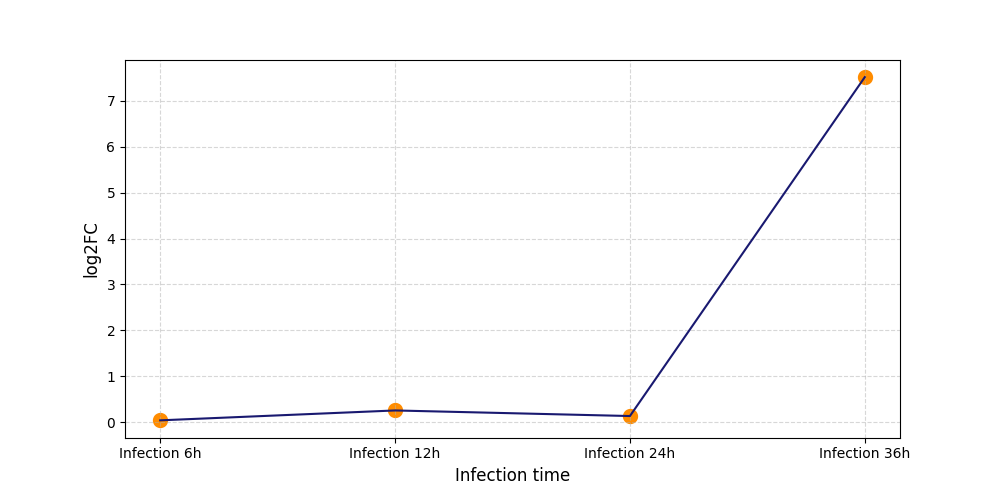
|
S544
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
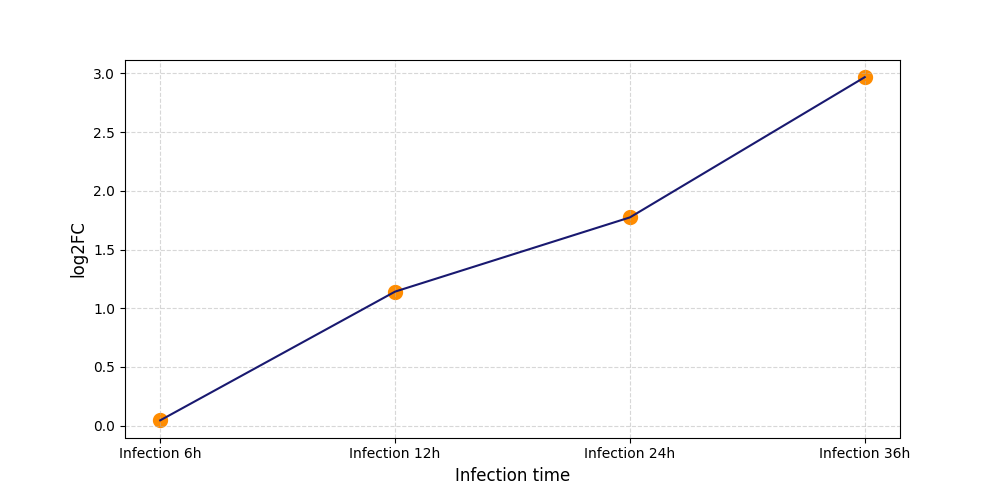
|
S601
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S614
[4] |
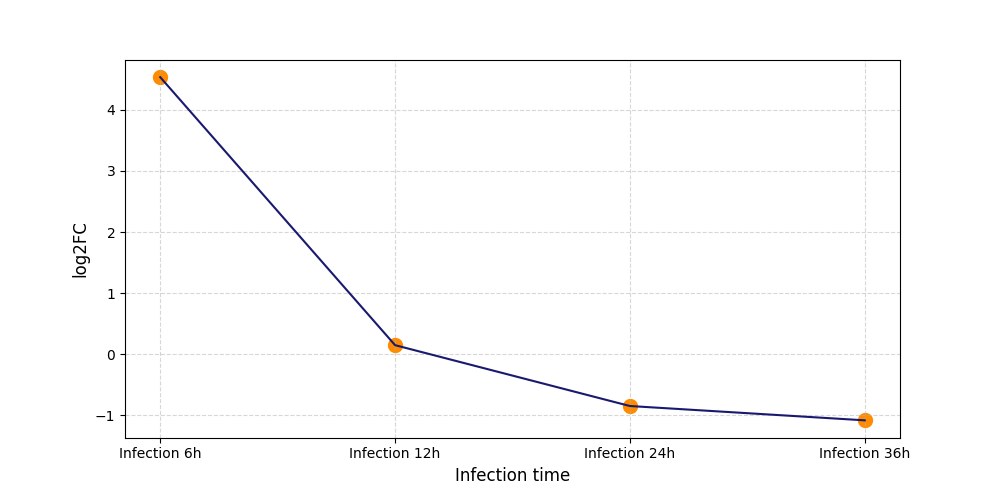 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S831
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S832
[4] |
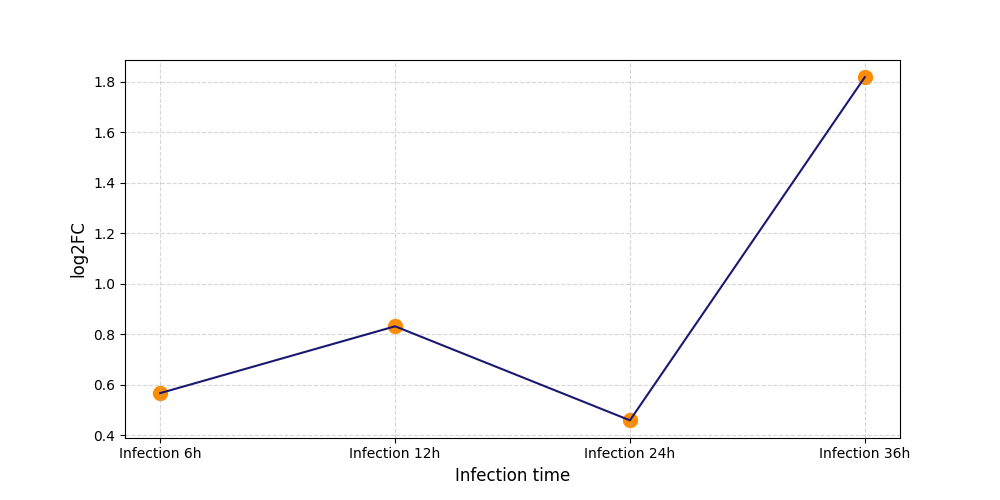 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S936
[4] |
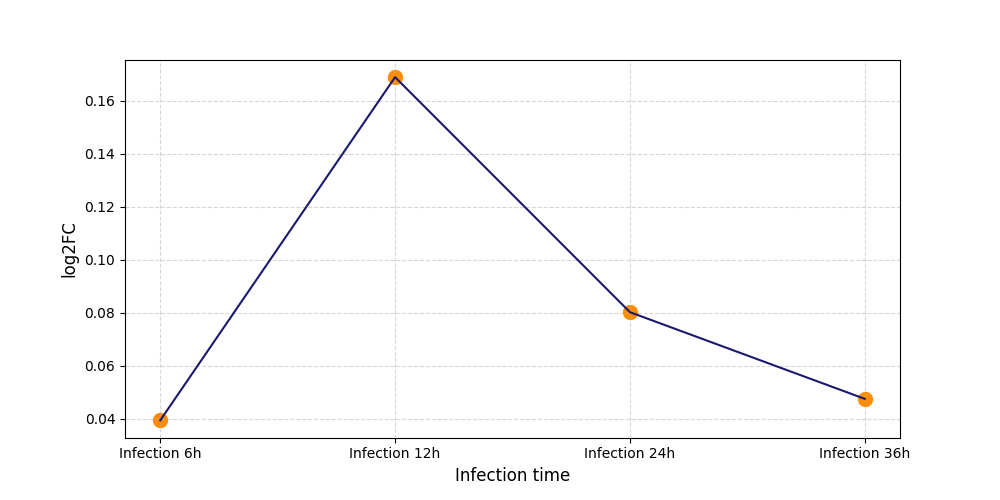 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S937
[4] |
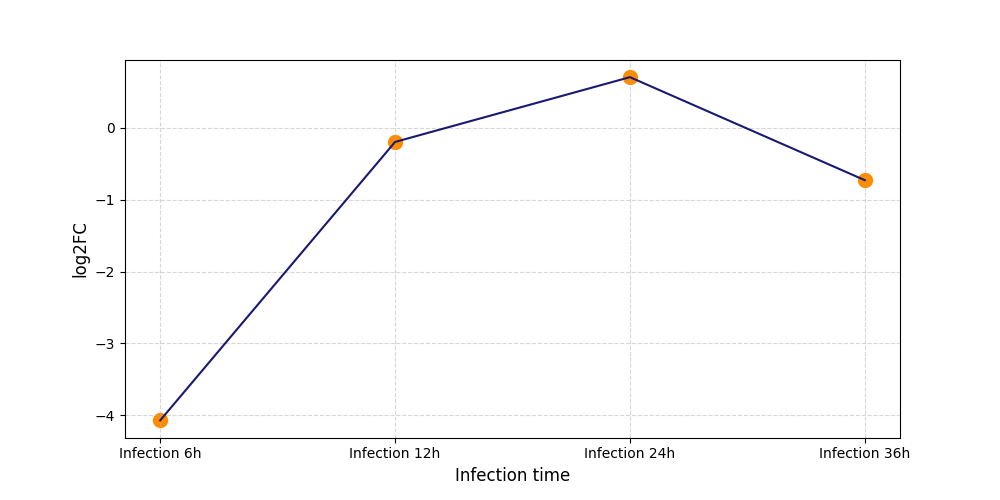 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S967
[4] |
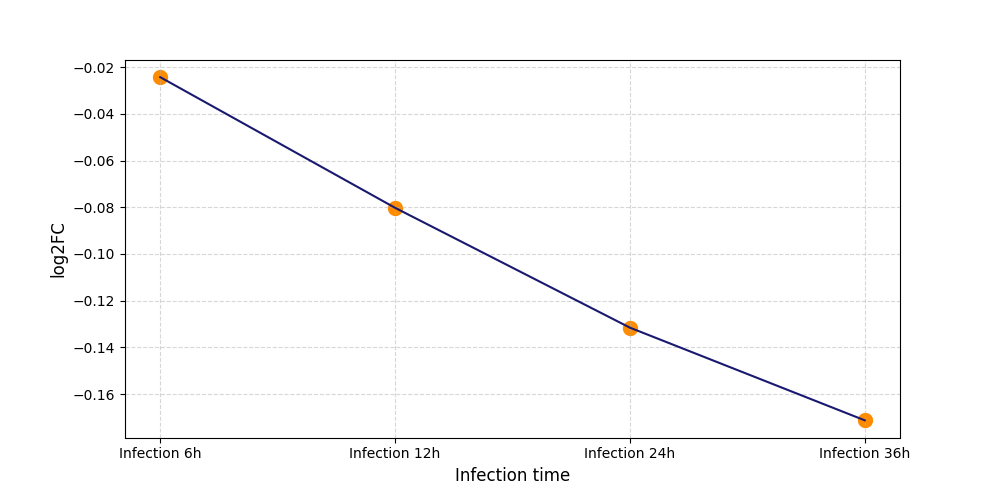 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S970
[4] |
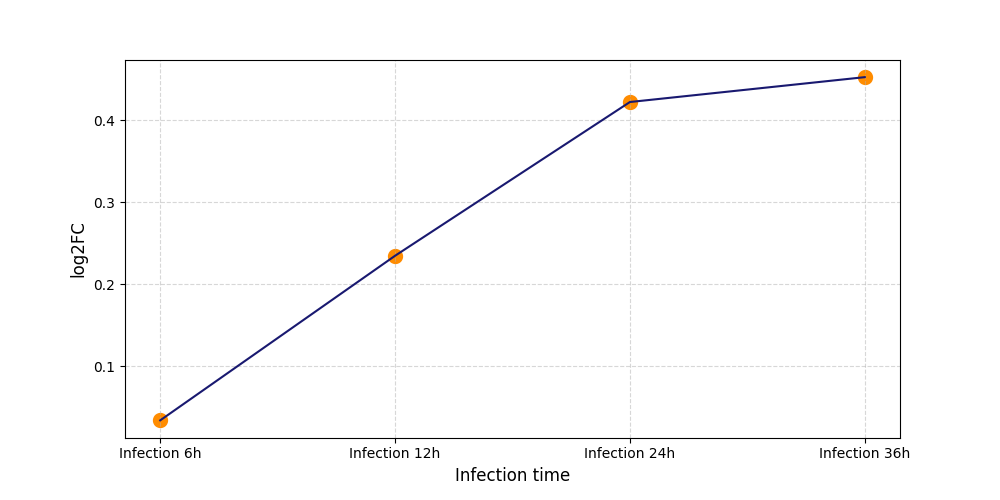 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S992
[4] |
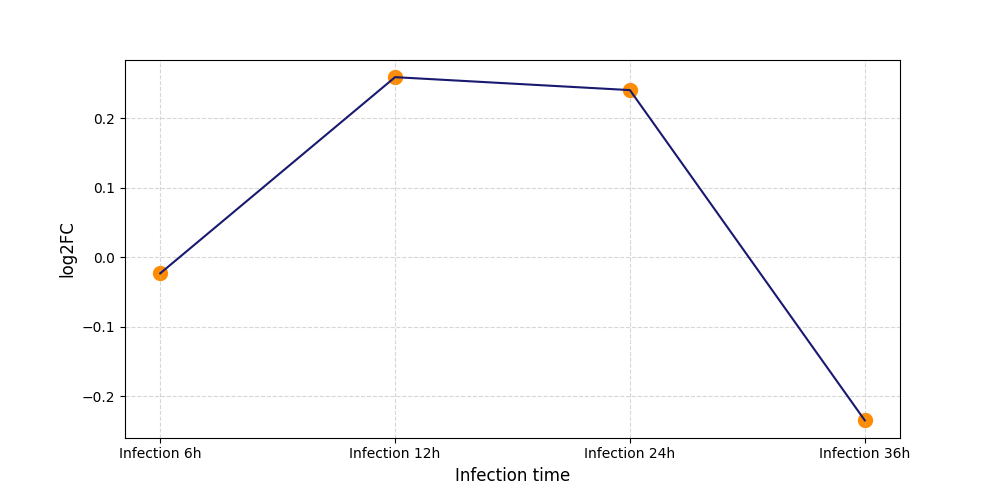 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S995
[4] |
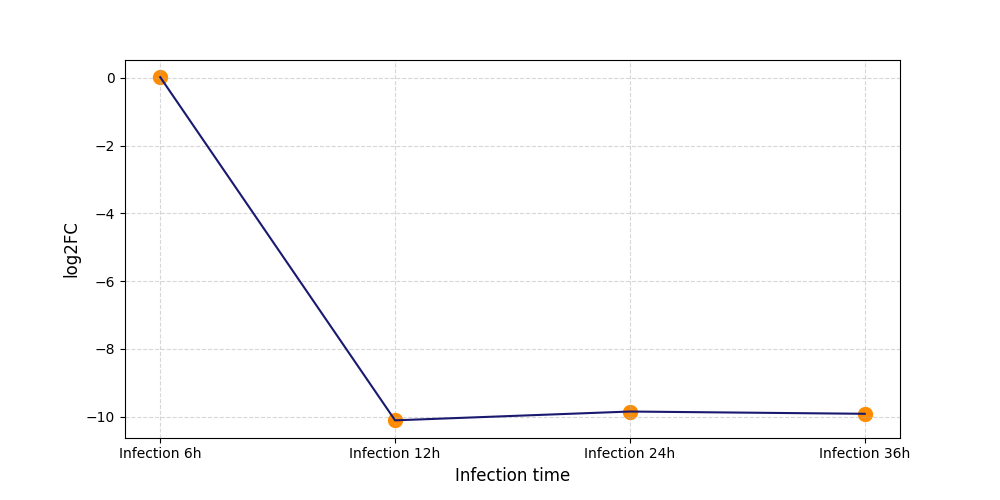 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
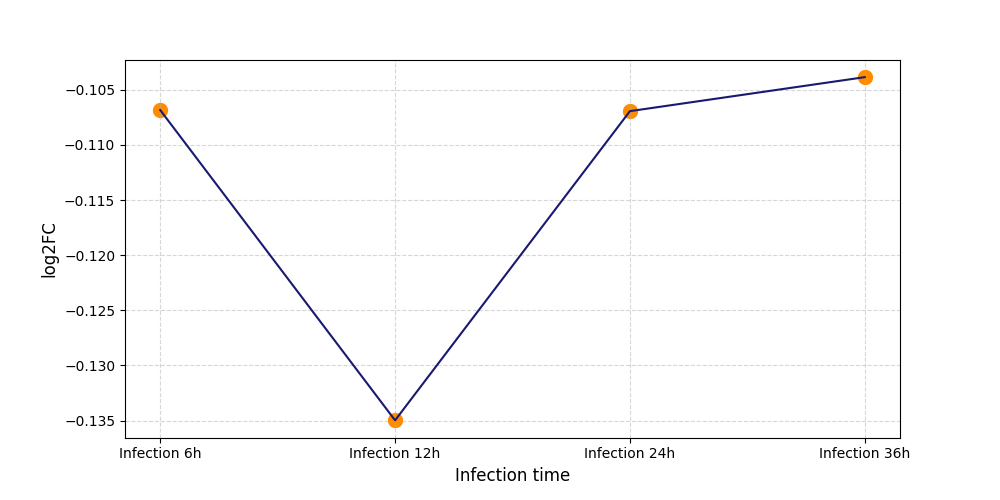
T1067
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
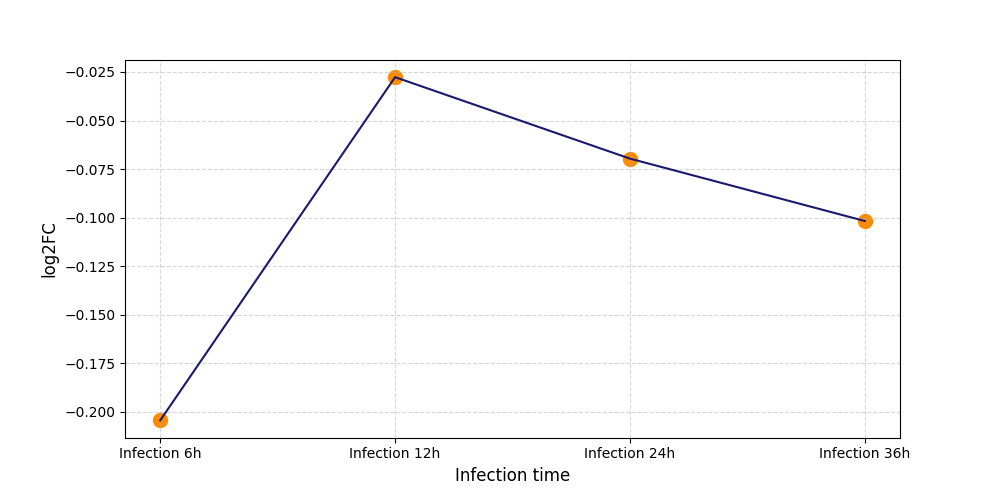
T1069
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
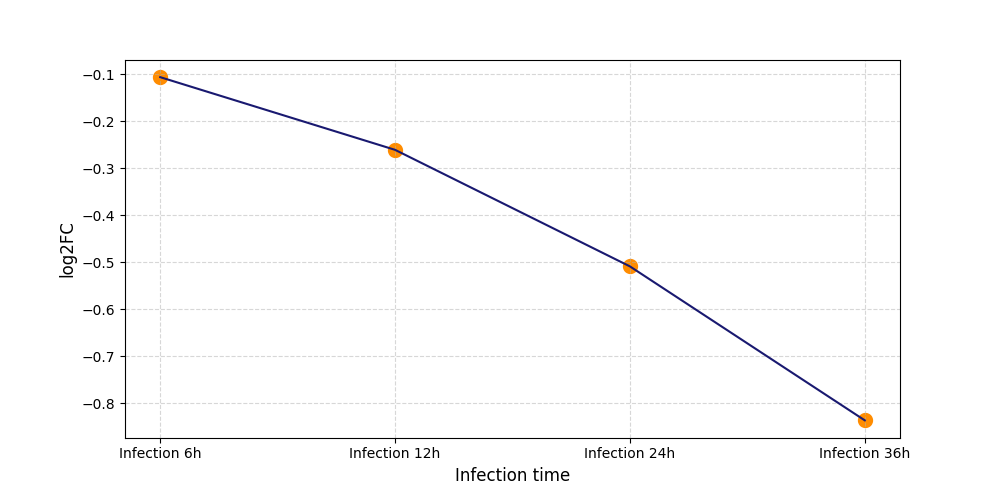
T1282
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
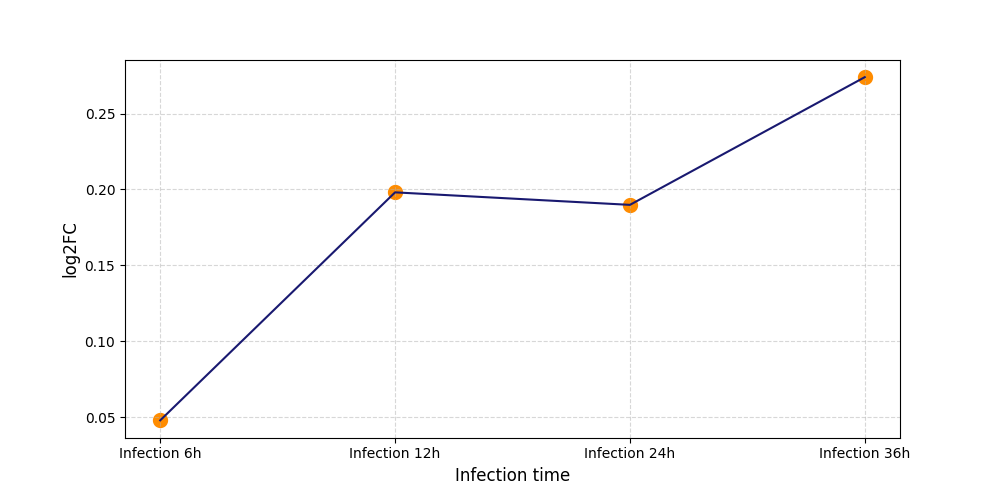
T1302
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1503
[4] |
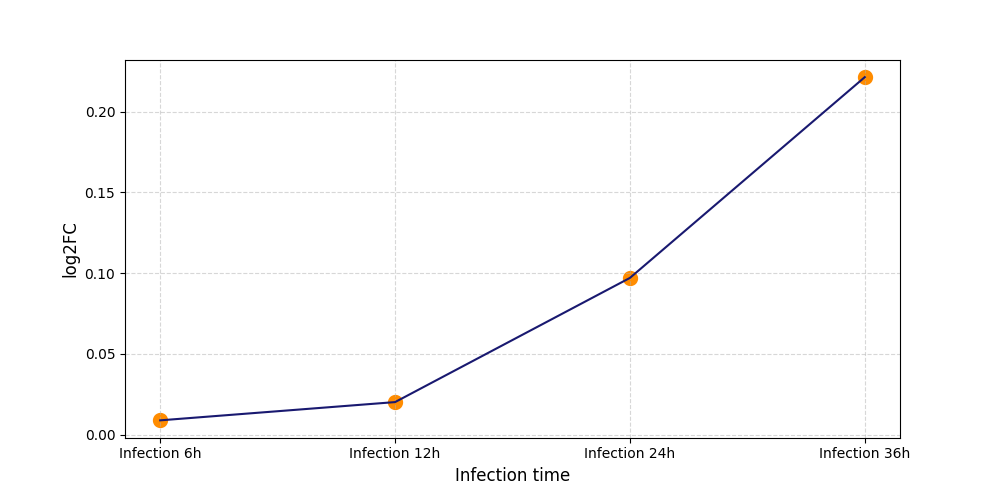 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1633
[4] |
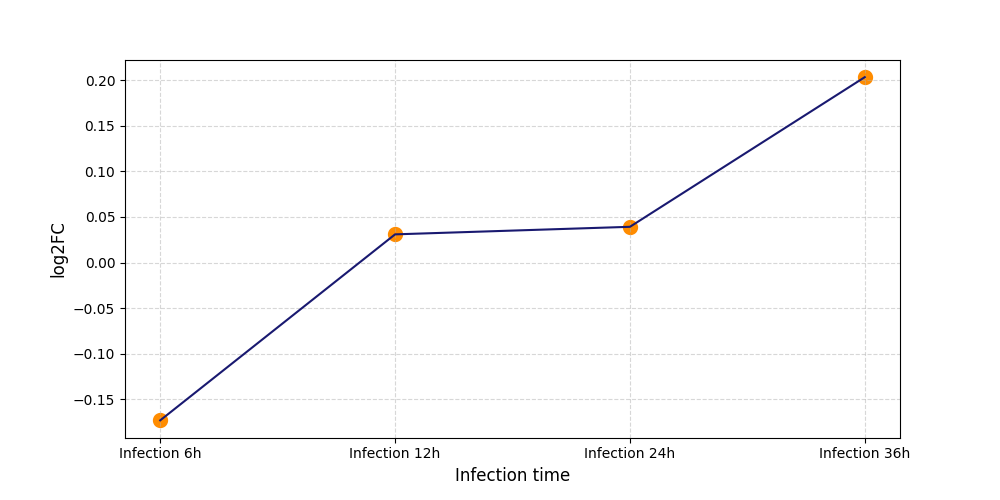 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1788
[4] |
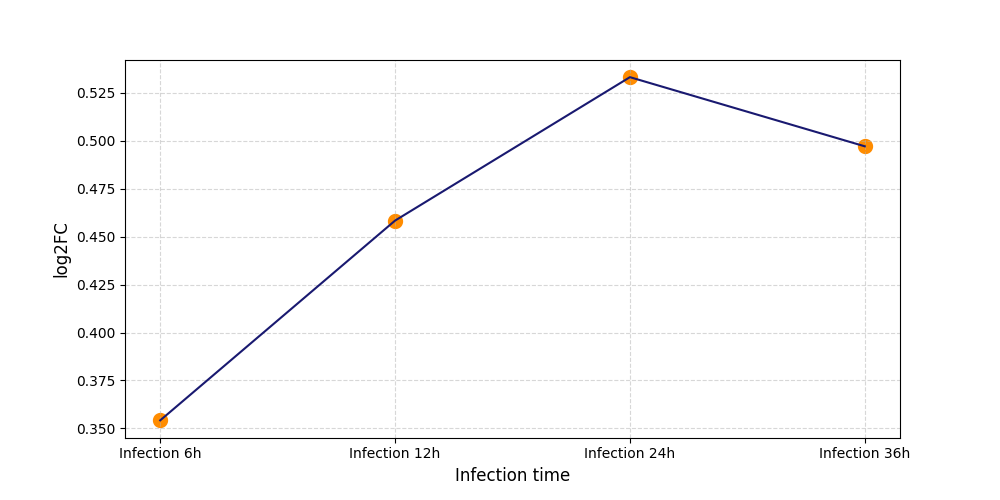 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1853
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1864
[4] |
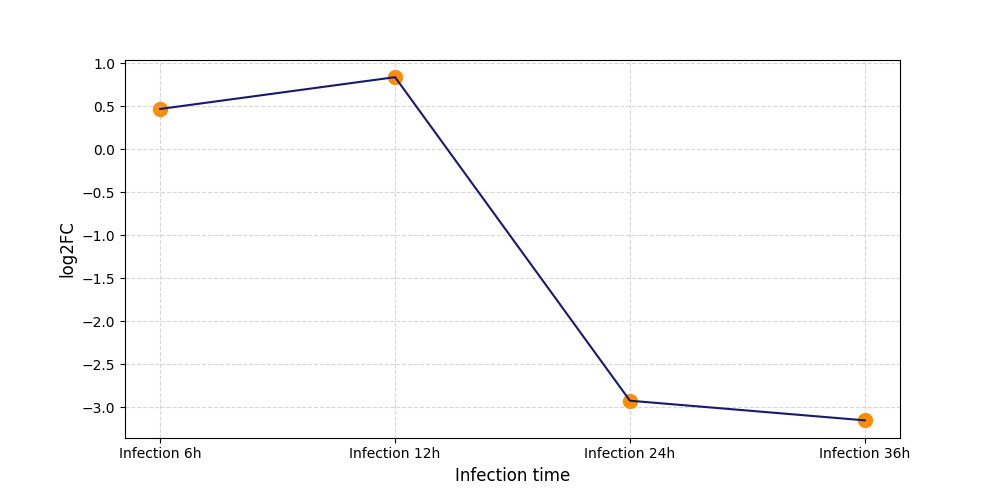 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1898
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1929
[4] |
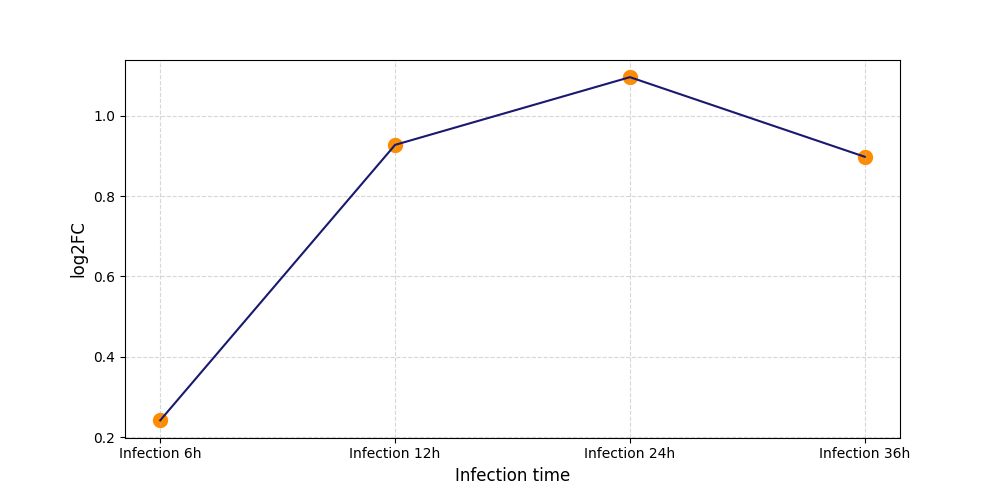 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1932
[4] |
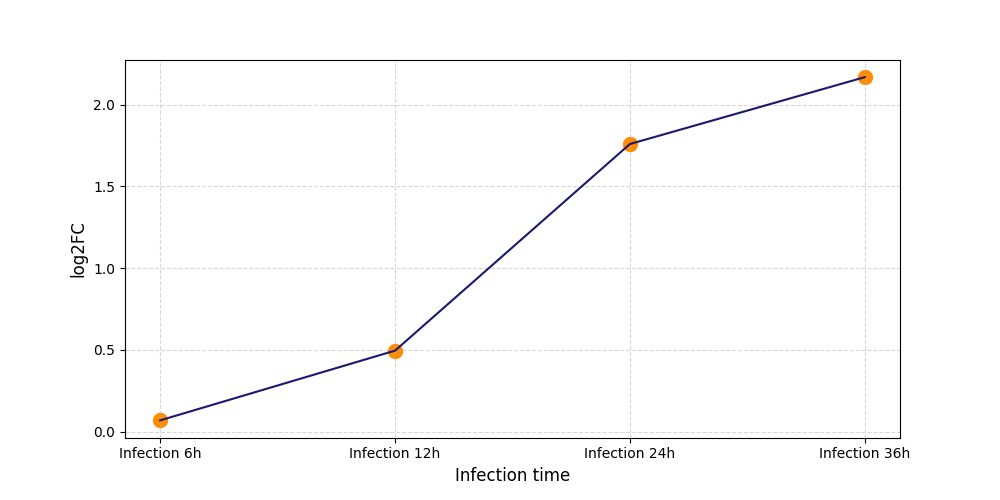 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1949
[5] |
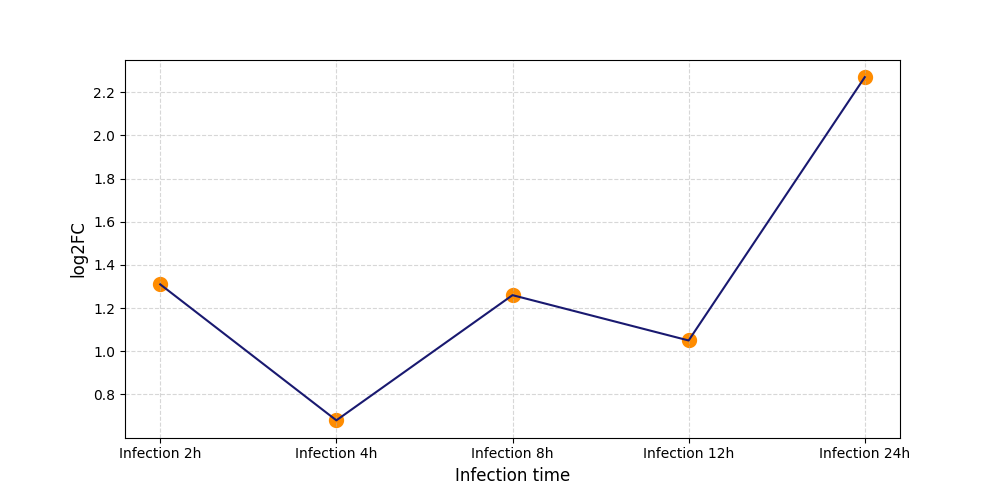 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1949
[4] |
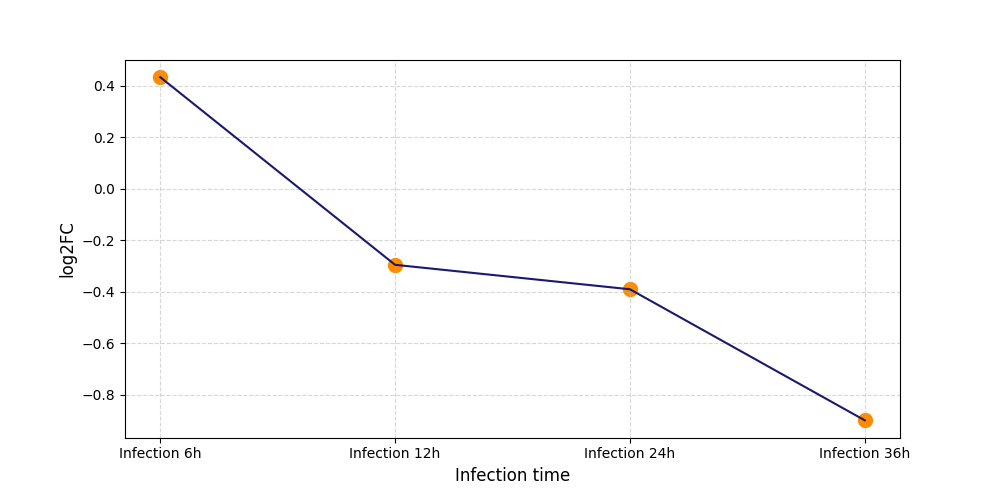 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
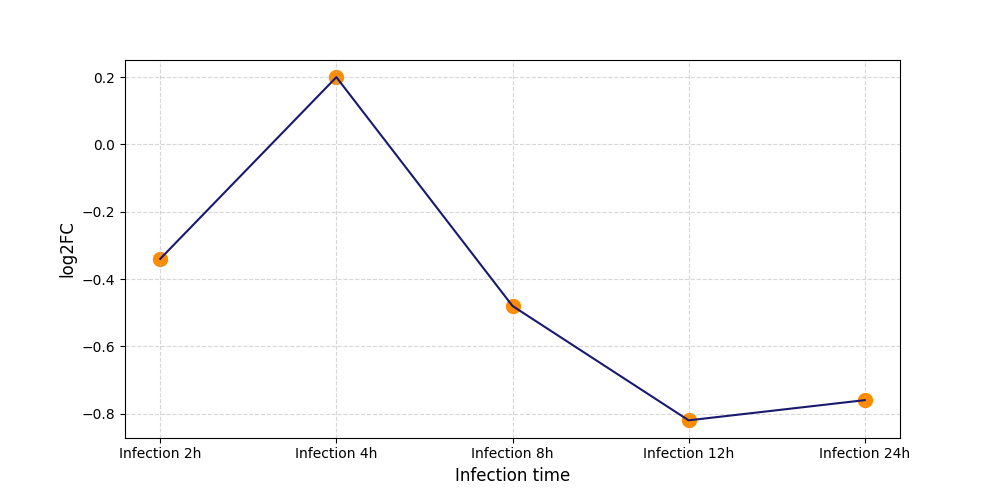
T2034
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
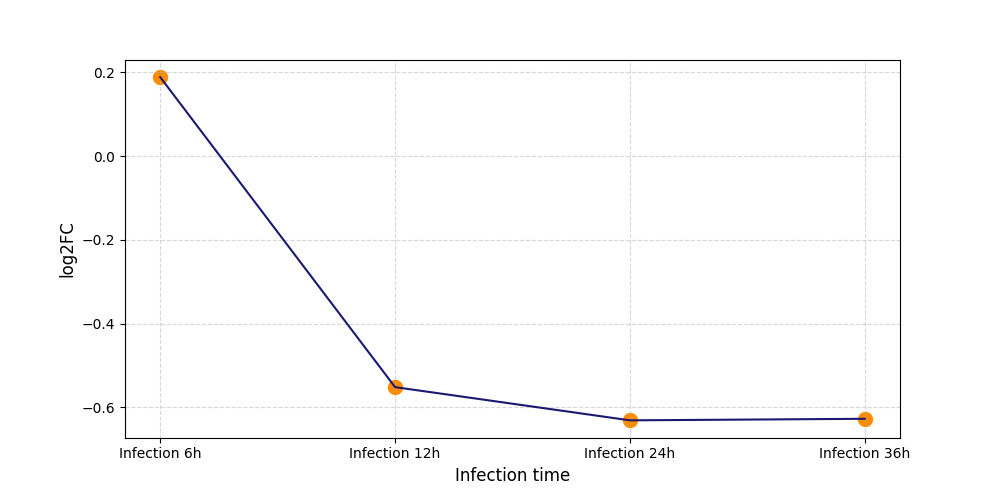
T2034
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
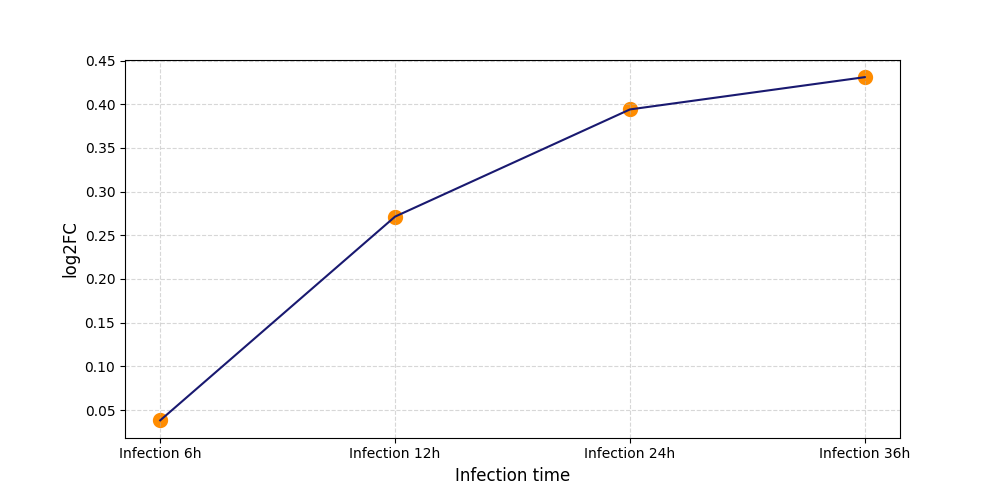
T2124
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
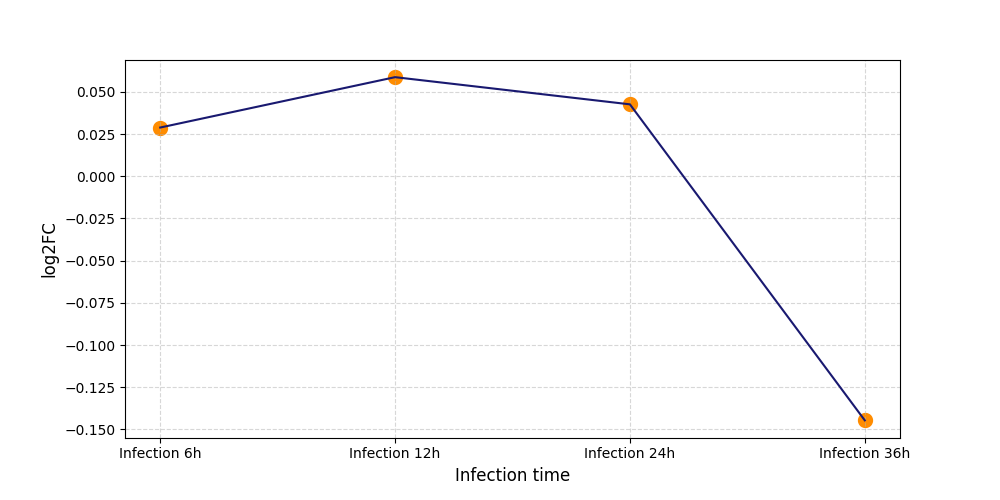
T2304
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
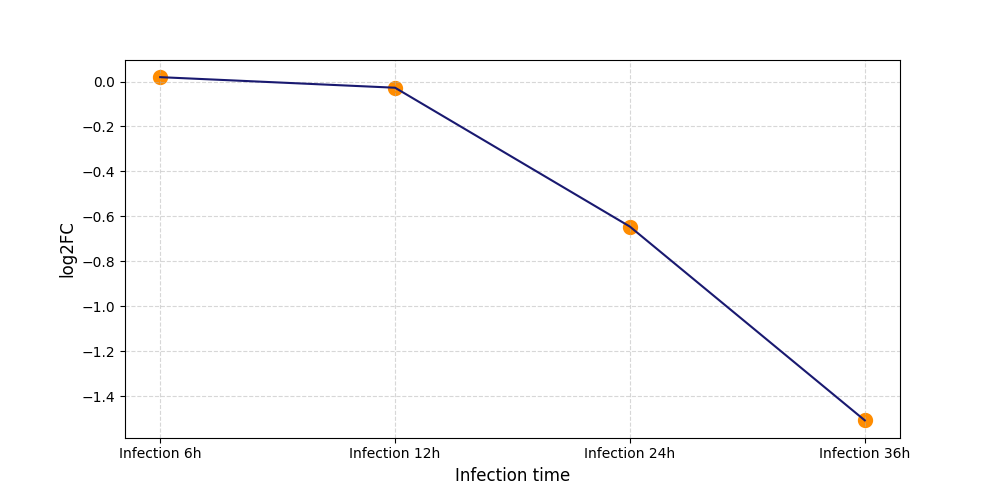
T2305
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
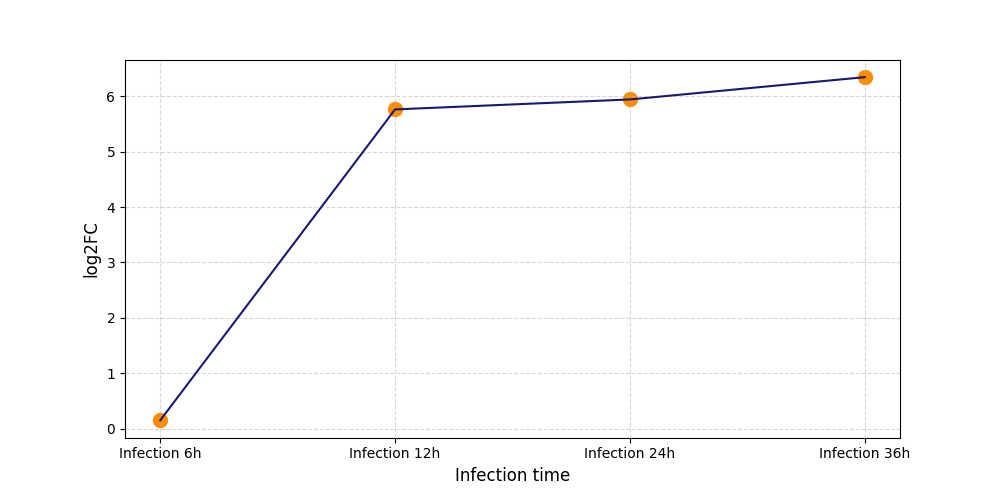
T3
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
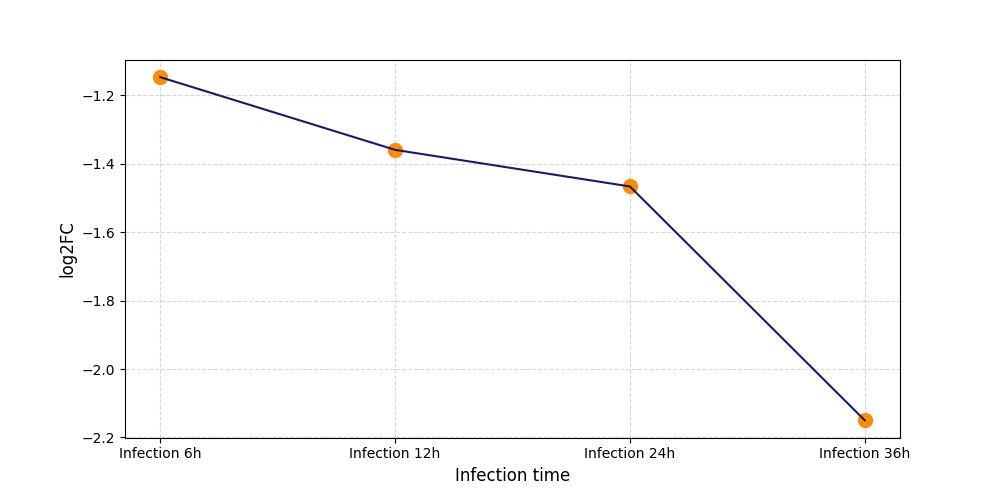
T527
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
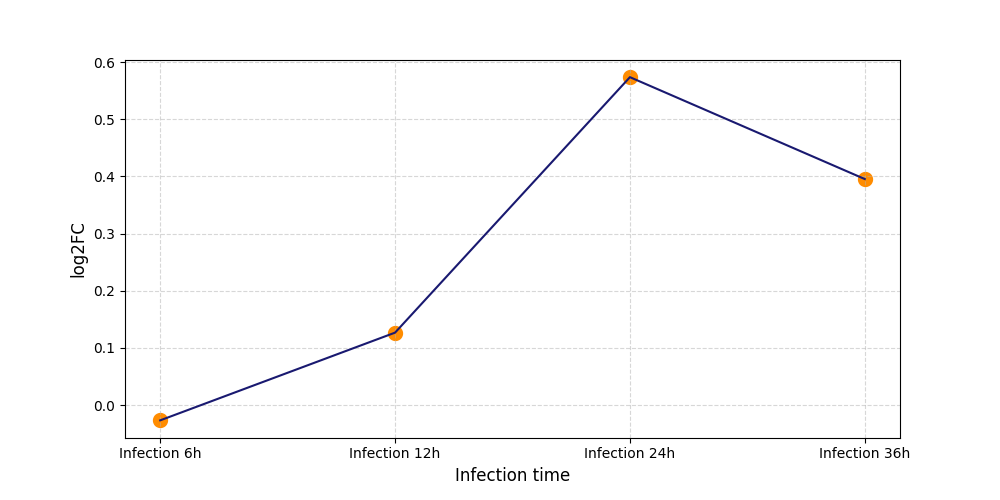
T603
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T744
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T837
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T908
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T948
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
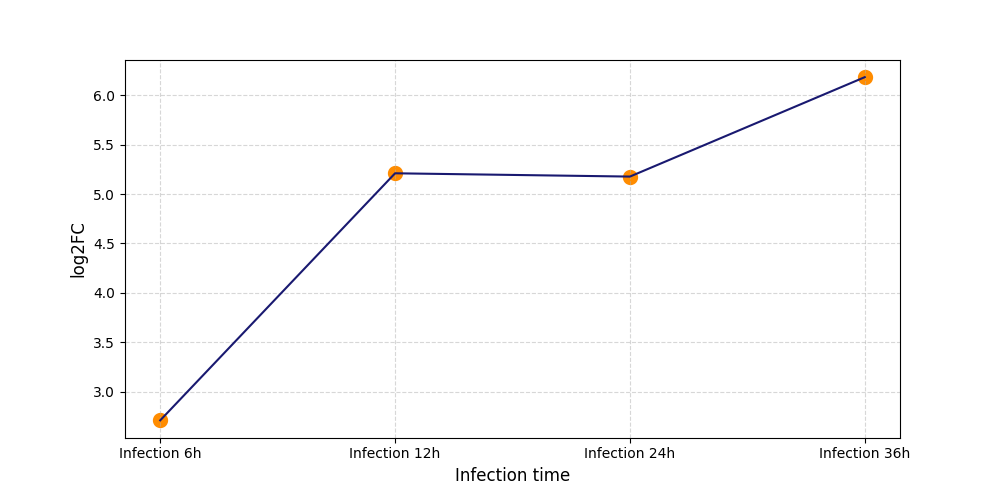
Y1689
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
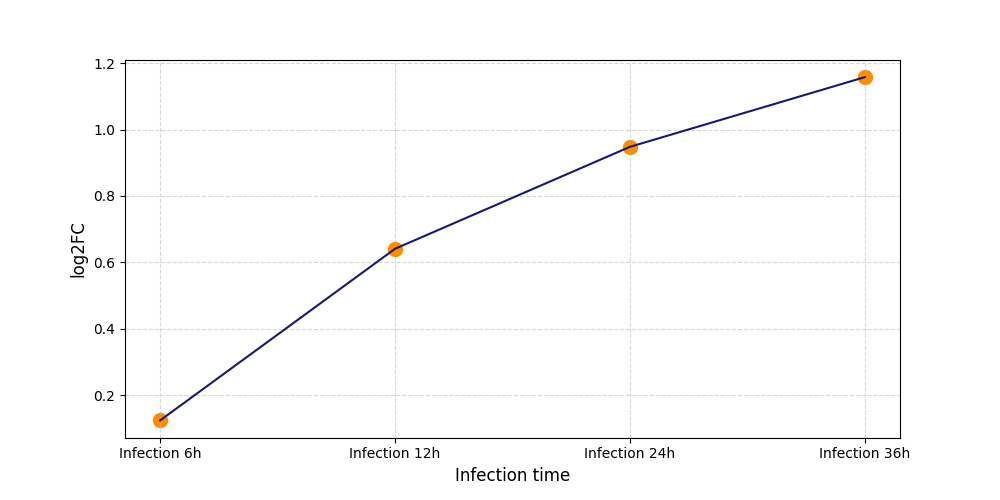
Y1796
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
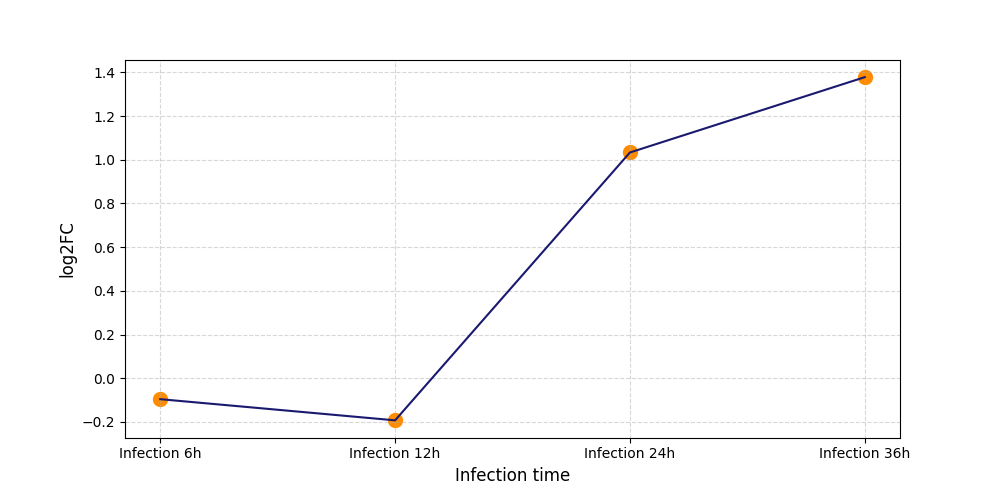
Y1887
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Sequence Information |
MATVVVEATEPEPSGSIANPAASTSPSLSHRFLDSKFYLLVVVGEIVTEEHLRRAIGNIELGIRSWDTNLIECNLDQELKLFVSRHSARFSPEVPGQKILHHRSDVLETVVLINPSDEAVSTEVRLMITDAARHKLLVLTGQCFENTGELILQSGSFSFQNFIEIFTDQEIGELLSTTHPANKASLTLFCPEEGDWKNSNLDRHNLQDFINIKLNSASILPEMEGLSEFTEYLSESVEVPSPFDILEPPTSGGFLKLSKPCCYIFPGGRGDSALFAVNGFNMLINGGSERKSCFWKLIRHLDRVDSILLTHIGDDNLPGINSMLQRKIAELEEEQSQGSTTNSDWMKNLISPDLGVVFLNVPENLKNPEPNIKMKRSIEEACFTLQYLNKLSMKPEPLFRSVGNTIDPVILFQKMGVGKLEMYVLNPVKSSKEMQYFMQQWTGTNKDKAEFILPNGQEVDLPISYLTSVSSLIVWHPANPAEKIIRVLFPGNSTQYNILEGLEKLKHLDFLKQPLATQKDLTGQVPTPVVKQTKLKQRADSRESLKPAAKPLPSKSVRKESKEETPEVTKVNHVEKPPKVESKEKVMVKKDKPIKTETKPSVTEKEVPSKEEPSPVKAEVAEKQATDVKPKAAKEKTVKKETKVKPEDKKEEKEKPKKEVAKKEDKTPIKKEEKPKKEEVKKEVKKEIKKEEKKEPKKEVKKETPPKEVKKEVKKEEKKEVKKEEKEPKKEIKKLPKDAKKSSTPLSEAKKPAALKPKVPKKEESVKKDSVAAGKPKEKGKIKVIKKEGKAAEAVAAAVGTGATTAAVMAAAGIAAIGPAKELEAERSLMSSPEDLTKDFEELKAEEVDVTKDIKPQLELIEDEEKLKETEPVEAYVIQKEREVTKGPAESPDEGITTTEGEGECEQTPEELEPVEKQGVDDIEKFEDEGAGFEESSETGDYEEKAETEEAEEPEEDGEEHVCVSASKHSPTEDEESAKAEADAYIREKRESVASGDDRAEEDMDEAIEKGEAEQSEEEADEEDKAEDAREEEYEPEKMEAEDYVMAVVDKAAEAGGAEEQYGFLTTPTKQLGAQSPGREPASSIHDETLPGGSESEATASDEENREDQPEEFTATSGYTQSTIEISSEPTPMDEMSTPRDVMSDETNNEETESPSQEFVNITKYESSLYSQEYSKPADVTPLNGFSEGSKTDATDGKDYNASASTISPPSSMEEDKFSRSALRDAYCSEVKASTTLDIKDSISAVSSEKVSPSKSPSLSPSPPSPLEKTPLGERSVNFSLTPNEIKVSAEAEVAPVSPEVTQEVVEEHCASPEDKTLEVVSPSQSVTGSAGHTPYYQSPTDEKSSHLPTEVIEKPPAVPVSFEFSDAKDENERASVSPMDEPVPDSESPIEKVLSPLRSPPLIGSESAYESFLSADDKASGRGAESPFEEKSGKQGSPDQVSPVSEMTSTSLYQDKQEGKSTDFAPIKEDFGQEKKTDDVEAMSSQPALALDERKLGDVSPTQIDVSQFGSFKEDTKMSISEGTVSDKSATPVDEGVAEDTYSHMEGVASVSTASVATSSFPEPTTDDVSPSLHAEVGSPHSTEVDDSLSVSVVQTPTTFQETEMSPSKEECPRPMSISPPDFSPKTAKSRTPVQDHRSEQSSMSIEFGQESPEQSLAMDFSRQSPDHPTVGAGVLHITENGPTEVDYSPSDMQDSSLSHKIPPMEEPSYTQDNDLSELISVSQVEASPSTSSAHTPSQIASPLQEDTLSDVAPPRDMSLYASLTSEKVQSLEGEKLSPKSDISPLTPRESSPLYSPTFSDSTSAVKEKTATCHSSSSPPIDAASAEPYGFRASVLFDTMQHHLALNRDLSTPGLEKDSGGKTPGDFSYAYQKPEETTRSPDEEDYDYESYEKTTRTSDVGGYYYEKIERTTKSPSDSGYSYETIGKTTKTPEDGDYSYEIIEKTTRTPEEGGYSYDISEKTTSPPEVSGYSYEKTERSRRLLDDISNGYDDSEDGGHTLGDPSYSYETTEKITSFPESEGYSYETSTKTTRTPDTSTYCYETAEKITRTPQASTYSYETSDLCYTAEKKSPSEARQDVDLCLVSSCEYKHPKTELSPSFINPNPLEWFASEEPTEESEKPLTQSGGAPPPPGGKQQGRQCDETPPTSVSESAPSQTDSDVPPETEECPSITADANIDSEDESETIPTDKTVTYKHMDPPPAPVQDRSPSPRHPDVSMVDPEALAIEQNLGKALKKDLKEKTKTKKPGTKTKSSSPVKKSDGKSKPLAASPKPAGLKESSDKVSRVASPKKKESVEKAAKPTTTPEVKAARGEEKDKETKNAANASASKSAKTATAGPGTTKTTKSSAVPPGLPVYLDLCYIPNHSNSKNVDVEFFKRVRSSYYVVSGNDPAAEEPSRAVLDALLEGKAQWGSNMQVTLIPTHDSEVMREWYQETHEKQQDLNIMVLASSSTVVMQDESFPACKIEL
Click to Show/Hide
|
|---|