Details of Host Protein
| Host Protein General Information (ID: PT0658) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name |
Msx2-interacting protein (SPEN)
|
Gene Name |
SPEN
|
||||||
| Host Species |
Homo sapiens
|
Uniprot Entry Name |
MINT_HUMAN
|
||||||
| Protein Families |
RRM Spen family
|
||||||||
| Subcellular Location |
Nucleus
|
||||||||
| External Link | |||||||||
| NCBI Gene ID | |||||||||
| Uniprot ID | |||||||||
| Ensembl ID | |||||||||
| HGNC ID | |||||||||
| Function in Host |
May serve as a nuclear matrix platform that organizes andintegrates transcriptional responses. In osteoblasts, supportstranscription activation: synergizes with RUNX2 to enhance FGFR2-mediated activation of the osteocalcin FGF-responsive element (OCFRE). Has also been shown to be an essential corepressorprotein, which probably regulates different key pathways such as theNotch pathway. Negative regulator of the Notch pathway via itsinteraction with RBPSUH, which prevents the association between NOTCH1and RBPSUH, and therefore suppresses the transactivation activity ofNotch signaling. Blocks the differentiation of precursor B-cells intomarginal zone B-cells. Probably represses transcription via therecruitment of large complexes containing histone deacetylase proteins. May bind both to DNA and RNA.
[1-2]
Click to Show/Hide
|
||||||||
| 3D Structure |
|
||||||||
| Function of This Protein During Virus Infection | |||||||||
|---|---|---|---|---|---|---|---|---|---|
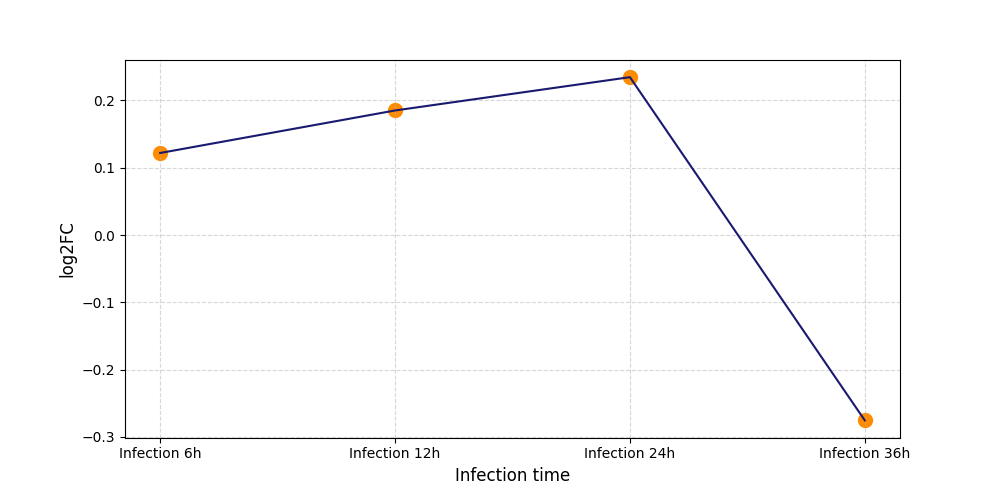
| Virus Name | SARS-COV-2 | Protein Function | Anti-viral | [3] | |||||
| Infected Tissue | Lung | Infection Time | 36 h | ||||||
| Infected Cell | A549 Cells (Adenocarcinomic Human alveolar basal epithelial cells) | Cellosaurus ID | CVCL_H249 | ||||||
| Method Description | To detect the role of host protein SPEN in viral infection, SPEN protein knockout A549 Cells were infected with SARS-COV-2 for 36 h , and the effects on infection was detected through WB. | ||||||||
| Results | It is reported that Knockdown of SPEN increases infection compared with control group. | ||||||||
| Full List of Virus RNA Interacting with This Protien | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| RNA Region: Not Specified Virus Region (hCoV-19/England/02/2020 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[4] | |||||||
| Strains Name |
hCoV-19/England/02/2020
|
||||||||
| Strains Family |
Beta (B.1.351)
|
||||||||
| RNA Binding Region |
Not Specified Virus Region
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Infection Cells | Calu-3 cells (Human lung cancer cell) (CVCL_0609 ) | ||||||||
| Cell Originated Tissue | Lung | ||||||||
| Infection Time | 24 h | ||||||||
| Interaction Score | P-adjust = 0.006 | ||||||||
| Method Description | UV protein-RNA crosslinking; RNA interactome capture (cRIC); RNA antisense purification coupled with mass spectrometry (RAP-MS) | ||||||||
| Differential Gene Expression During SARS-COV-2 Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Phosphorylation after Virus Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
S1006
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1062
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
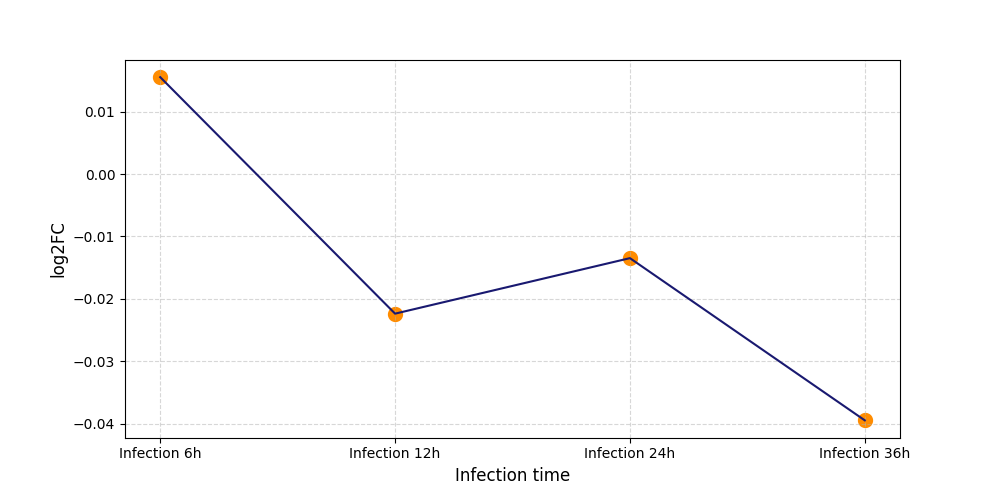
|
S1222
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
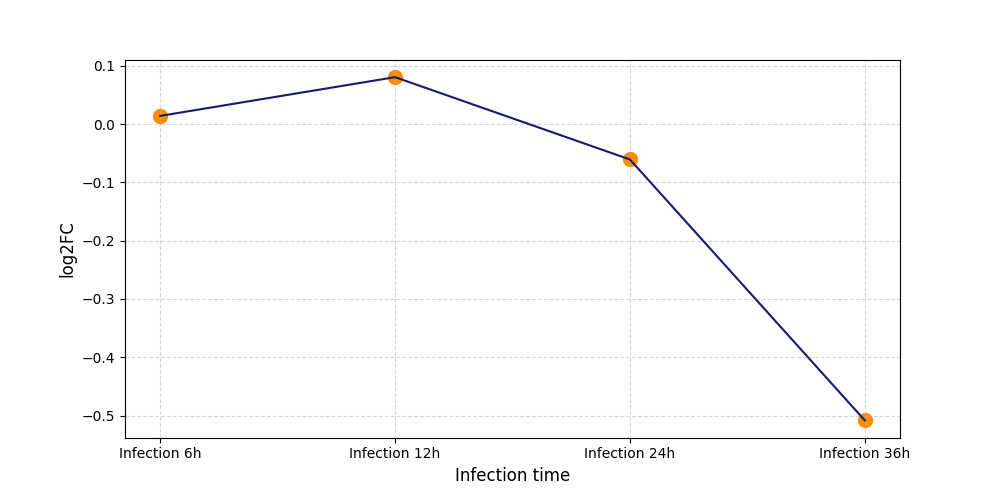
|
S1261
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
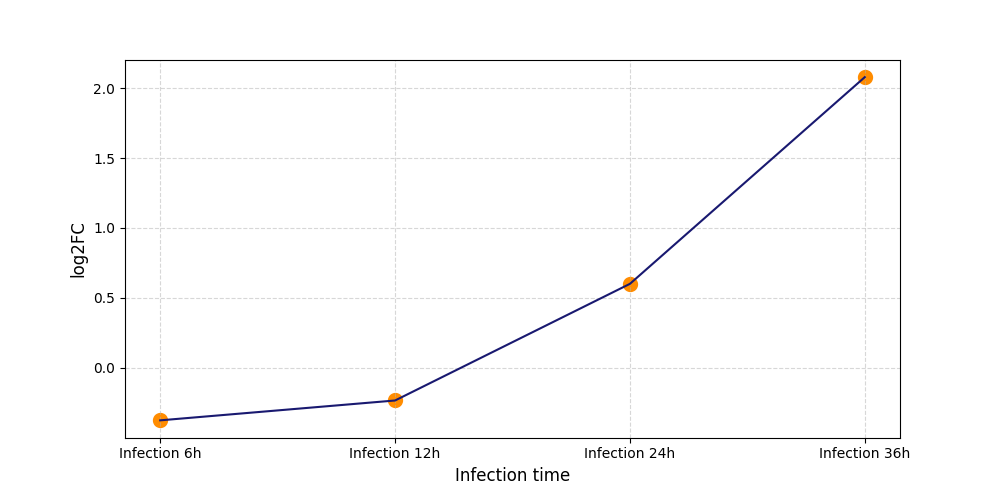
|
S1268
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1278
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
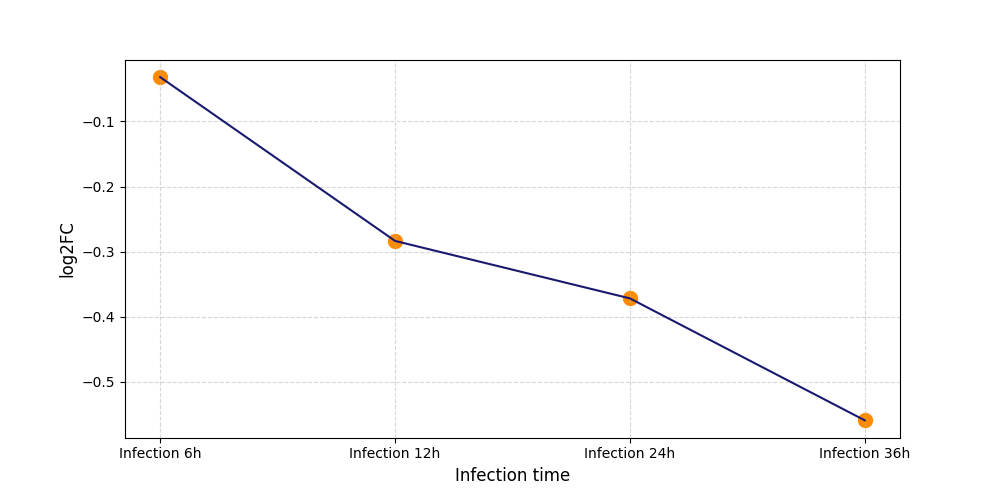
|
S1278
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
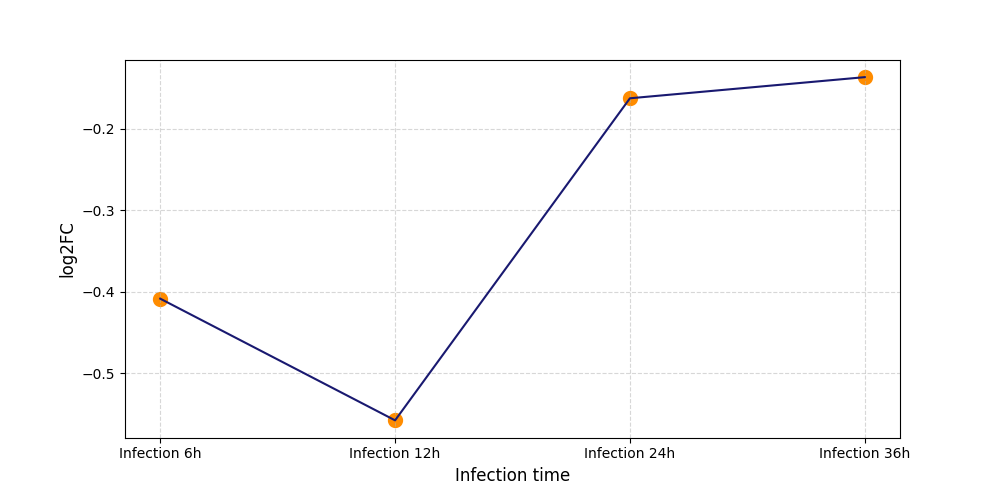
|
S188
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
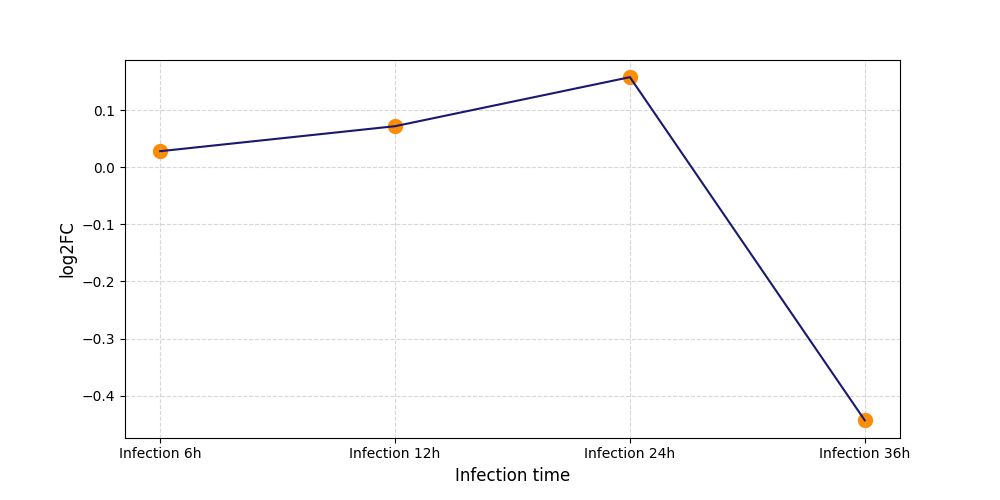
|
S190
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
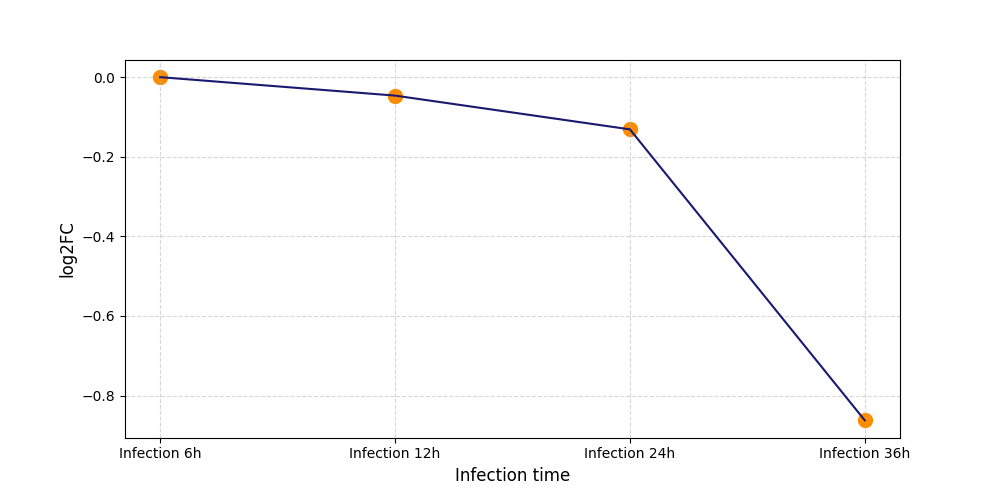
|
S1918
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
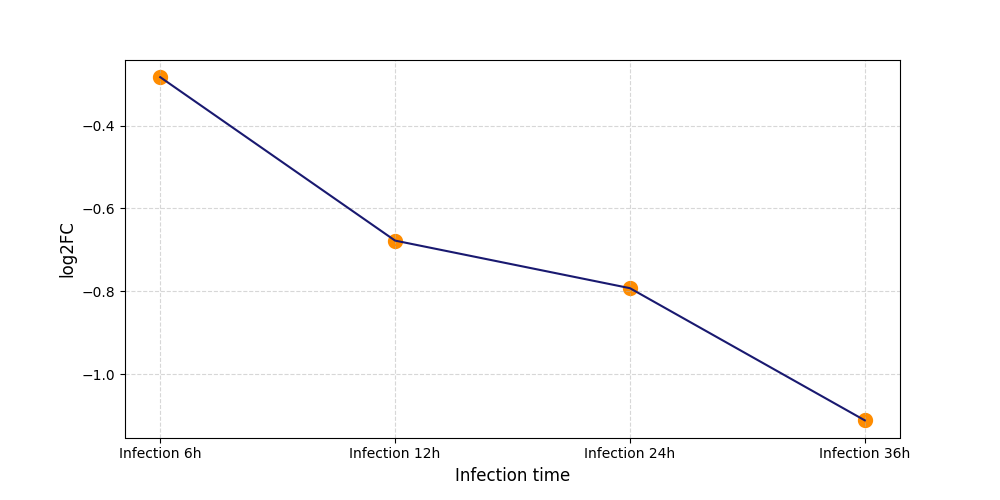
|
S1983
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2101
[5] |
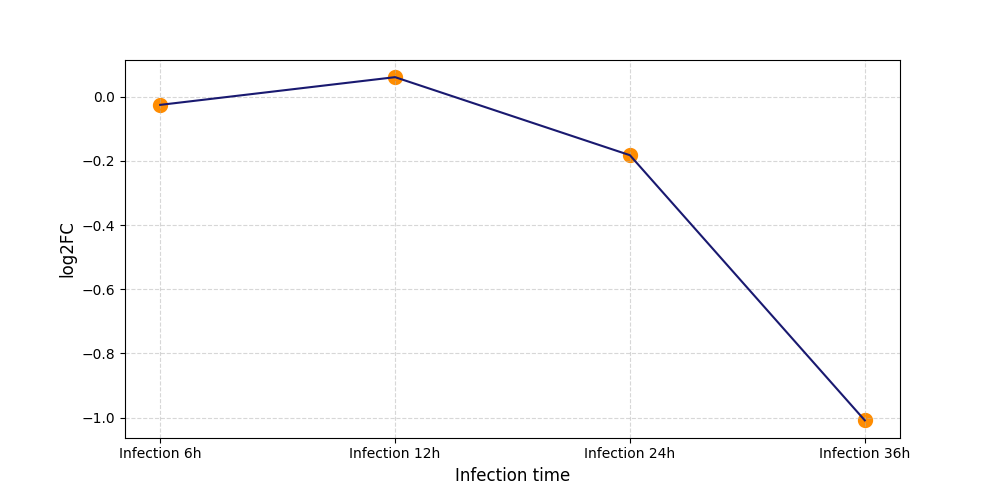 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2120
[5] |
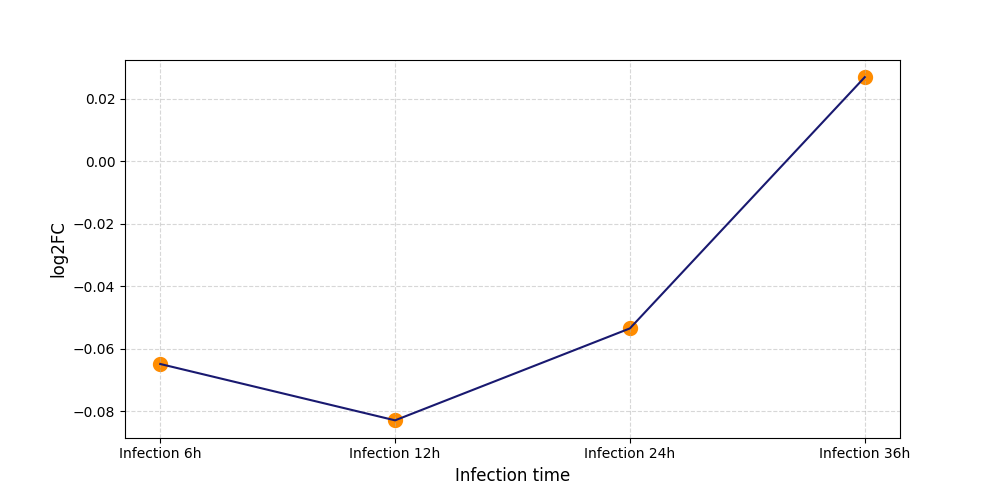 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2159
[5] |
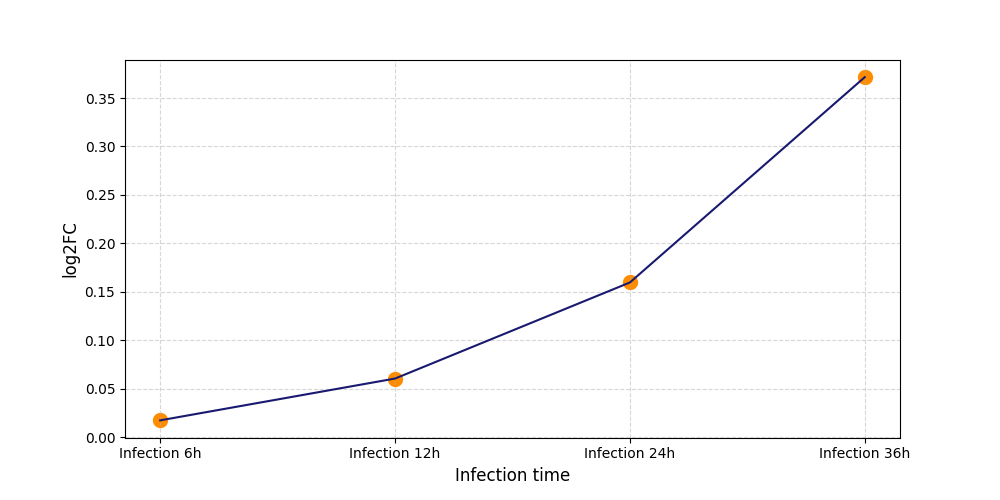 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2392
[5] |
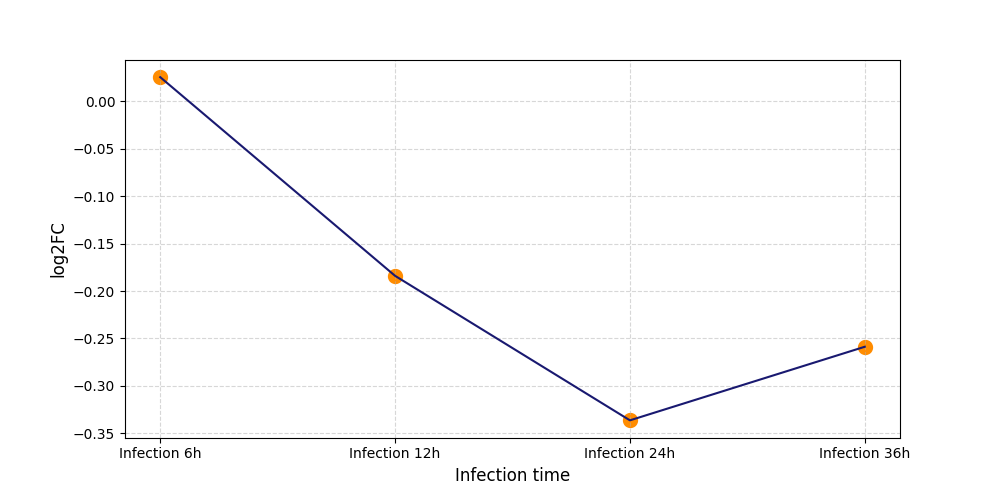 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
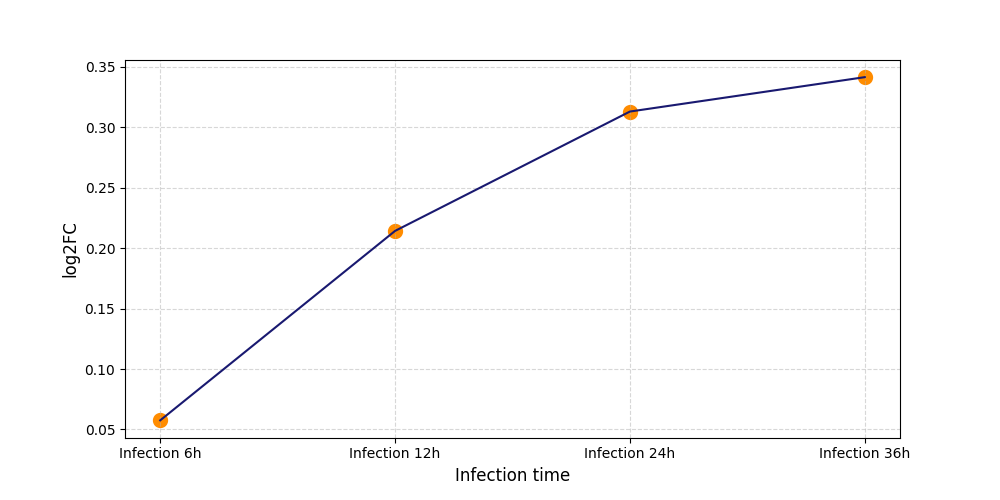
|
S2463
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
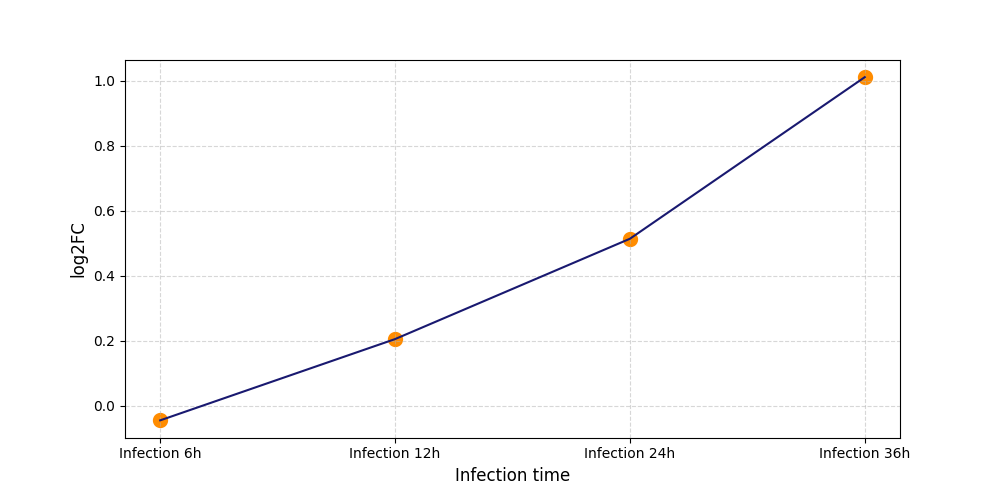
|
S2481
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
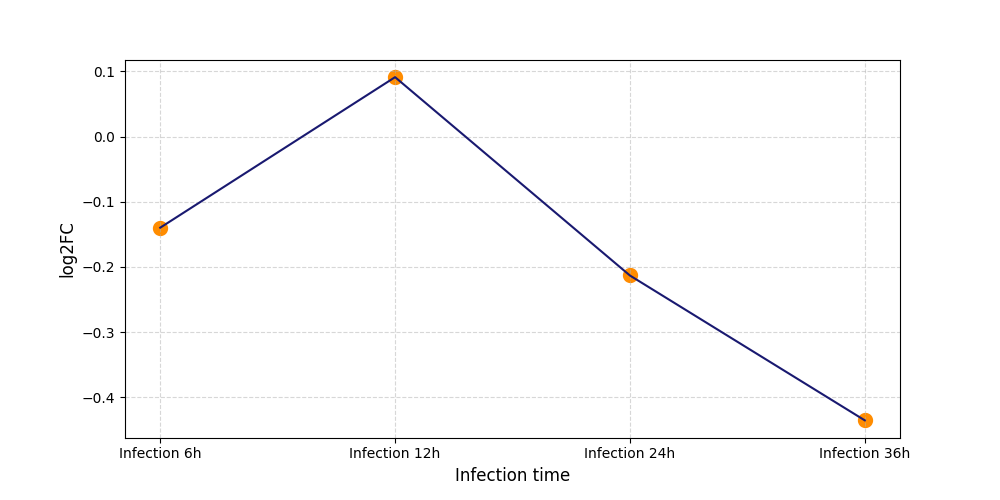
|
S2493
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
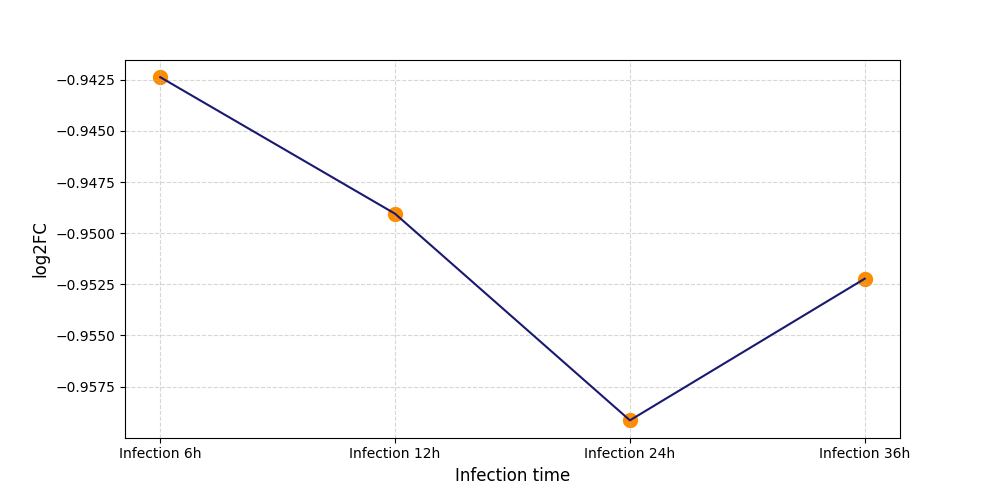
|
S3025
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S727
[6] |
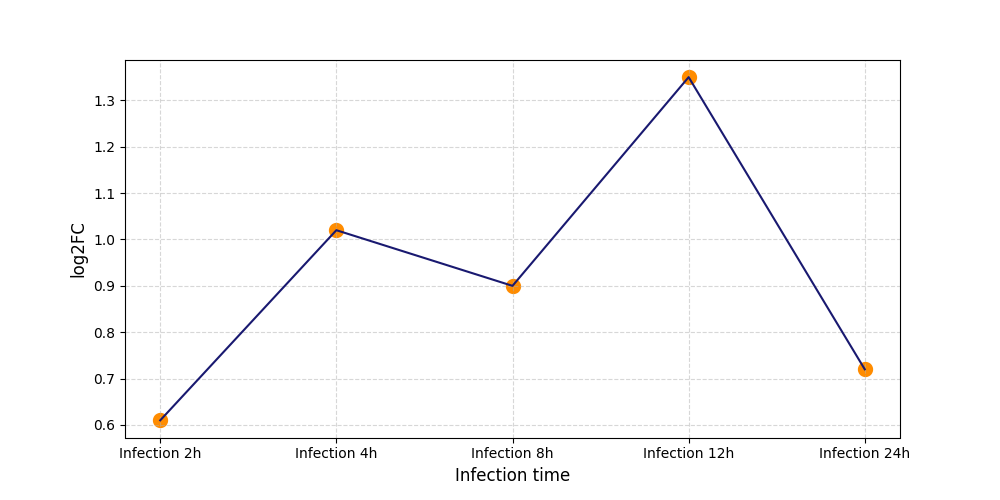 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S727
[5] |
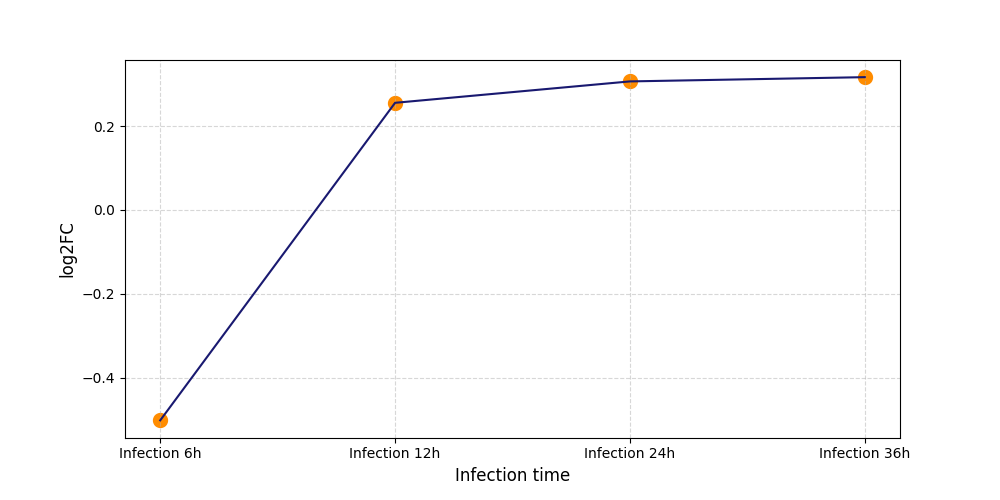 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S736
[5] |
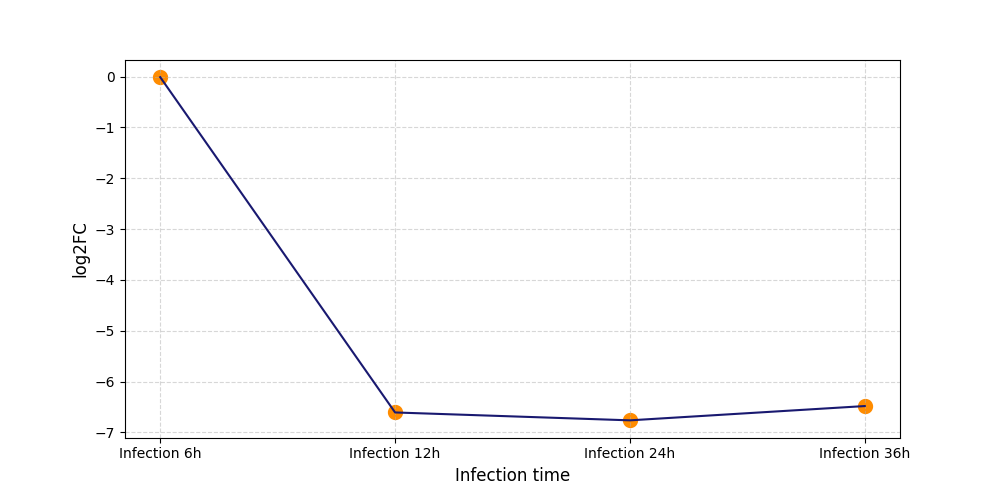 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S740
[5] |
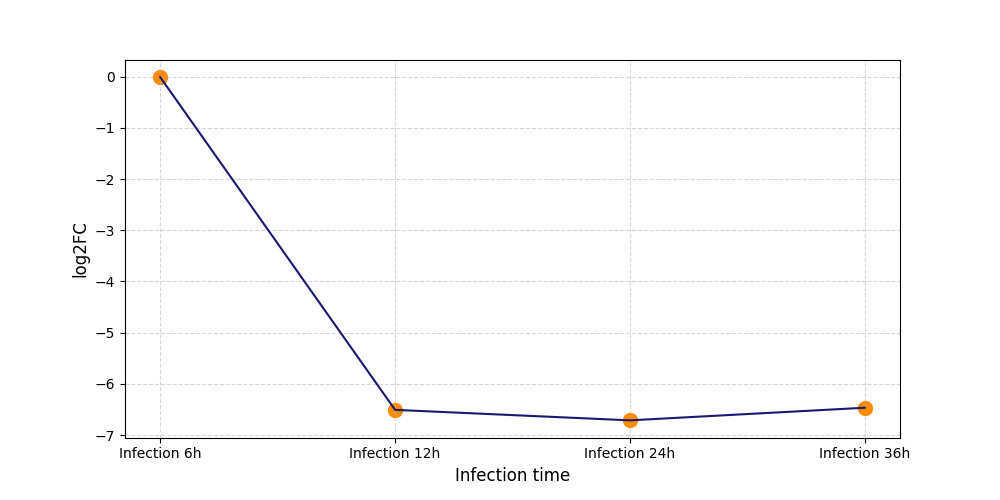 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
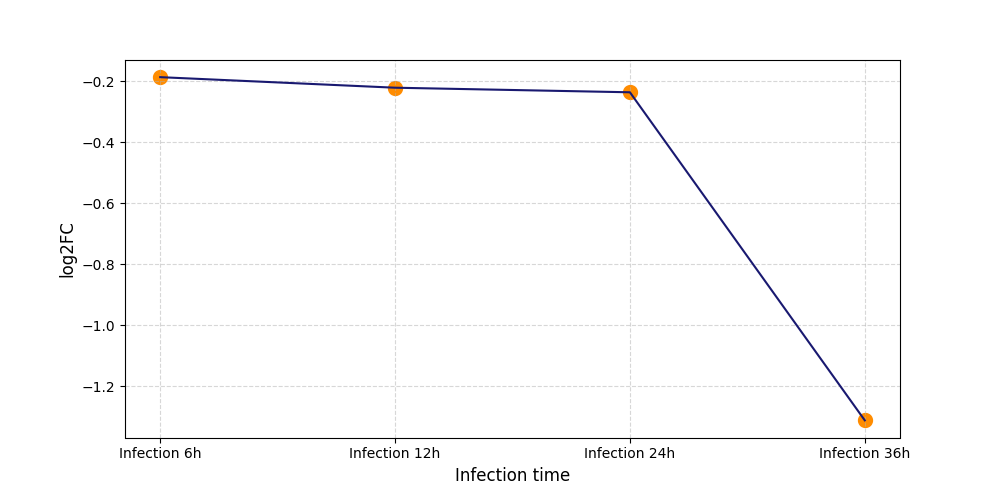
|
S749
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
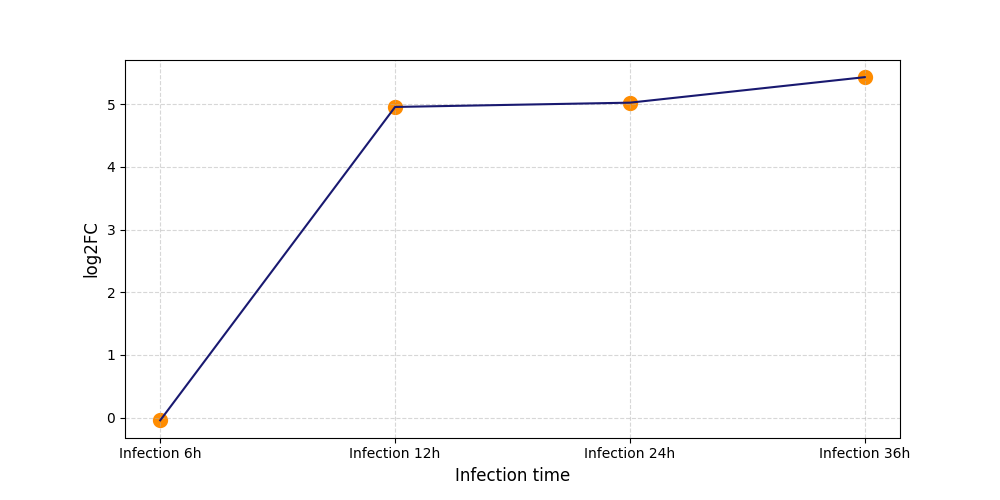
|
S751
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
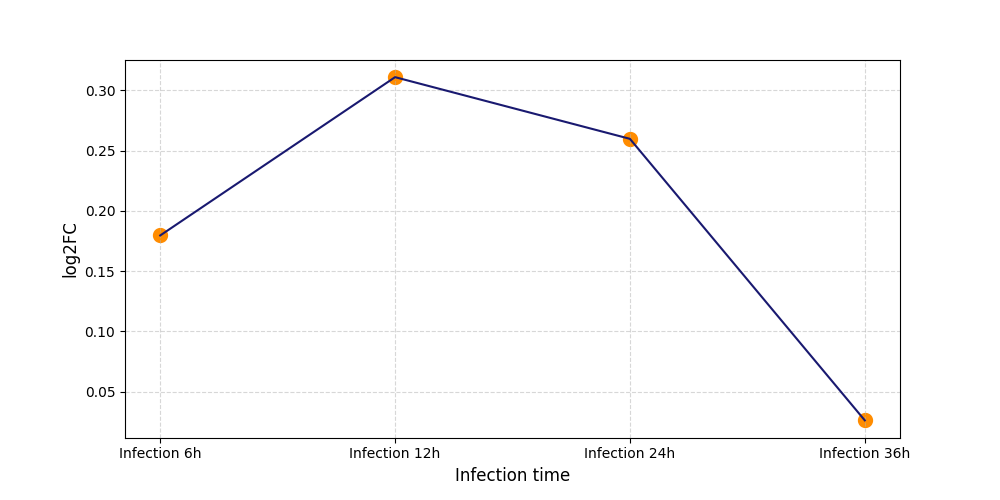
|
T1219
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
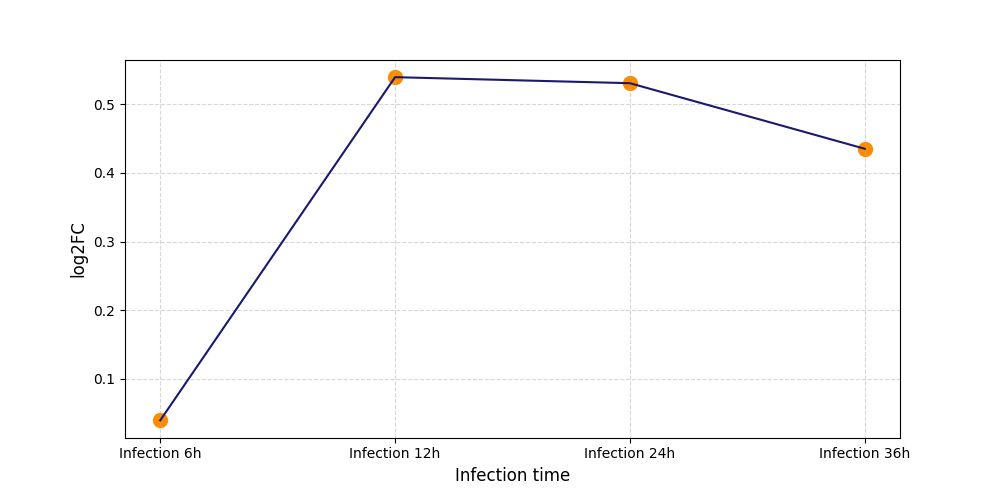
|
T1619
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
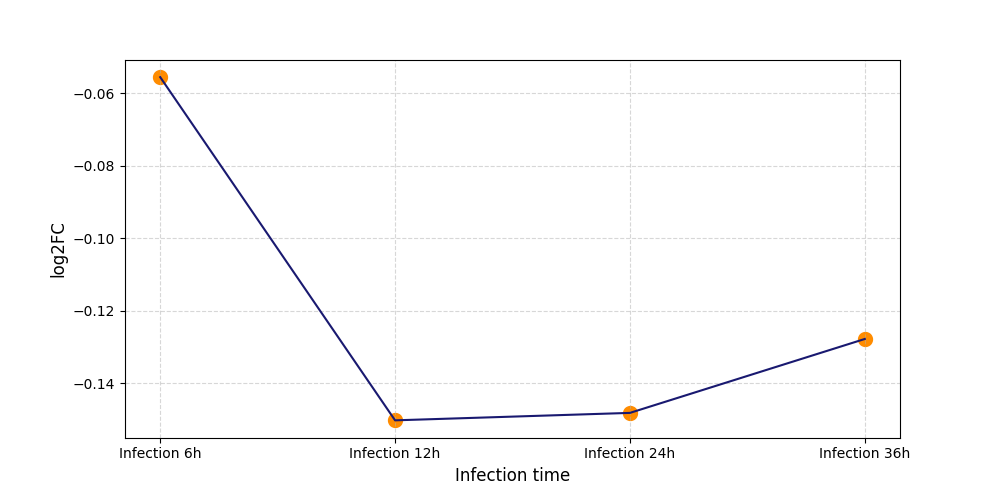
|
T1643
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
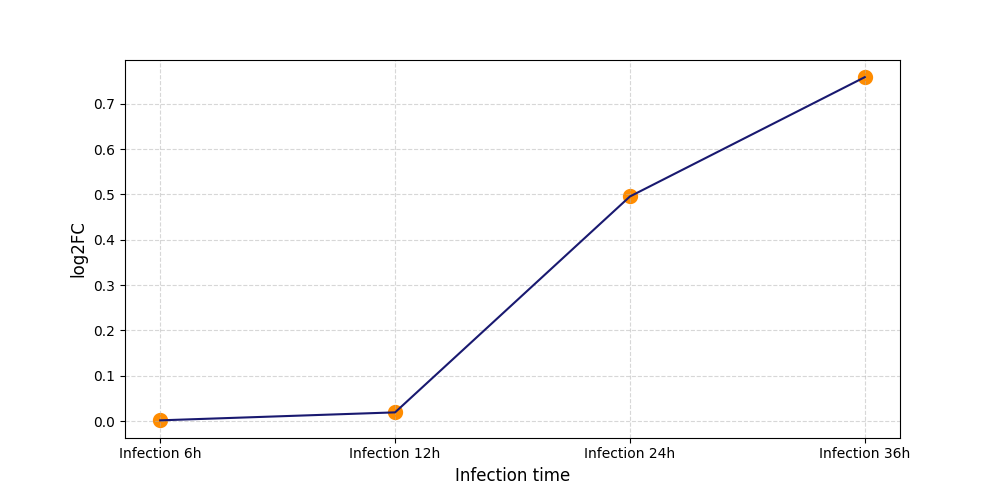
|
T1826
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
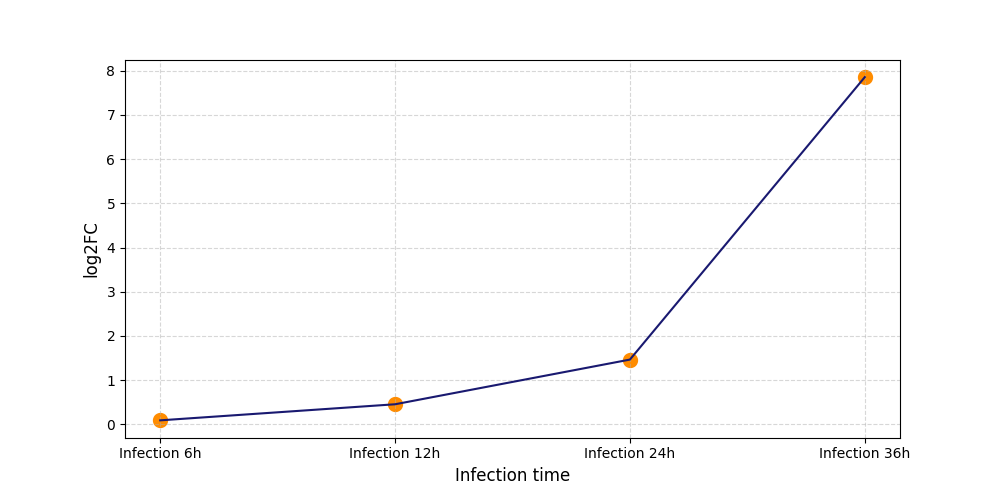
|
T1910
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
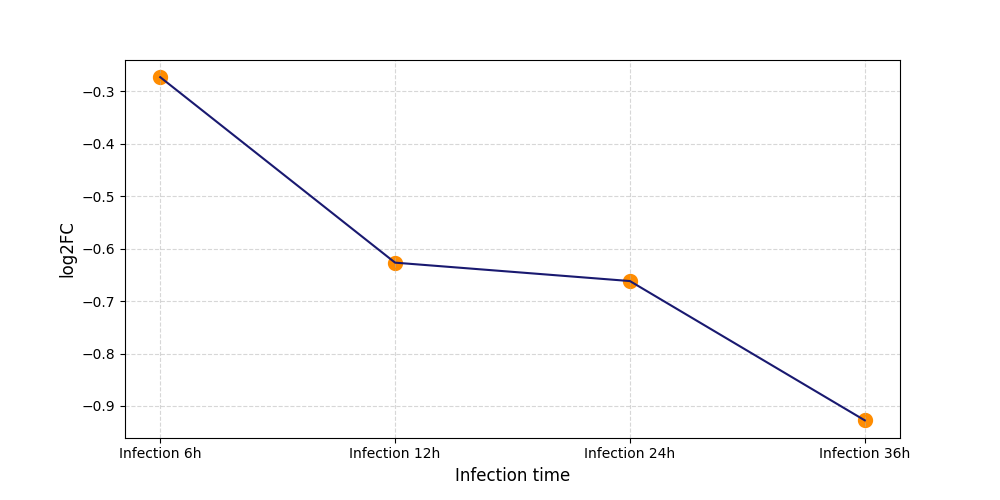
|
T1946
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
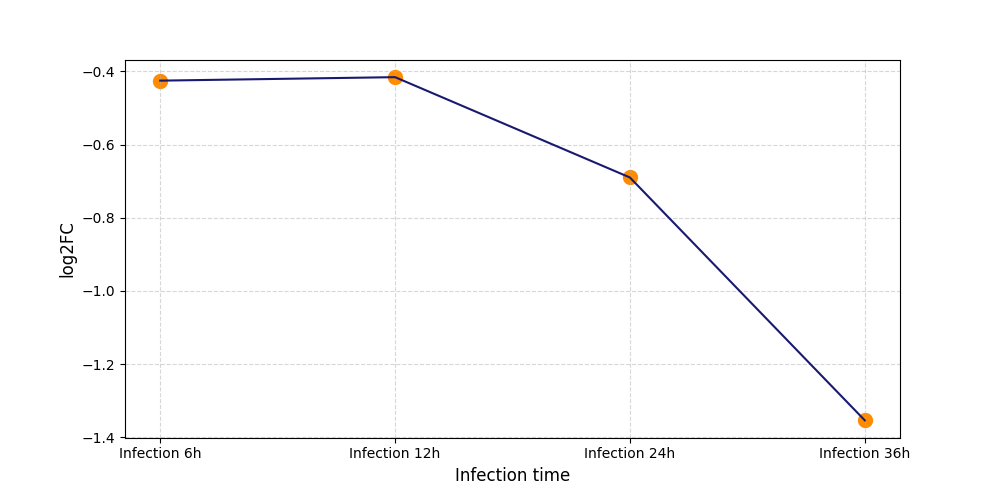
T1947
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
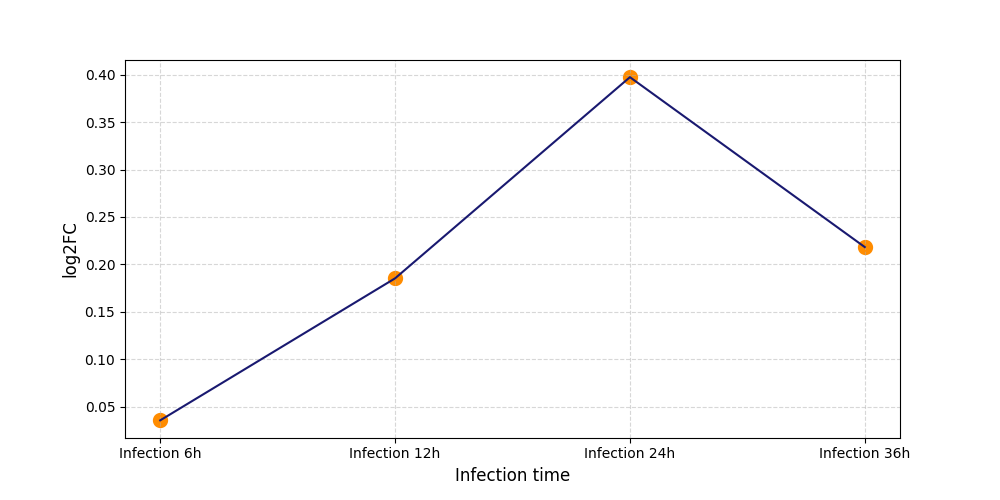
T2163
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
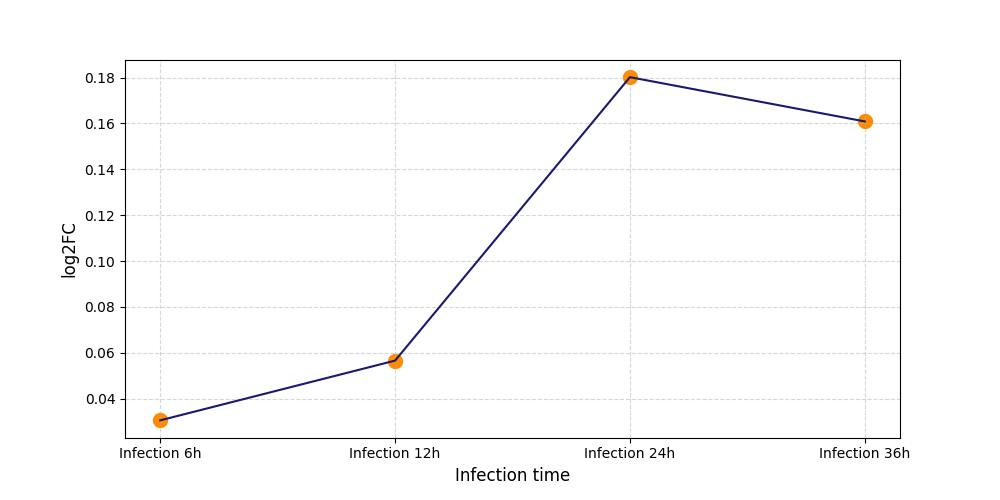
T2393
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
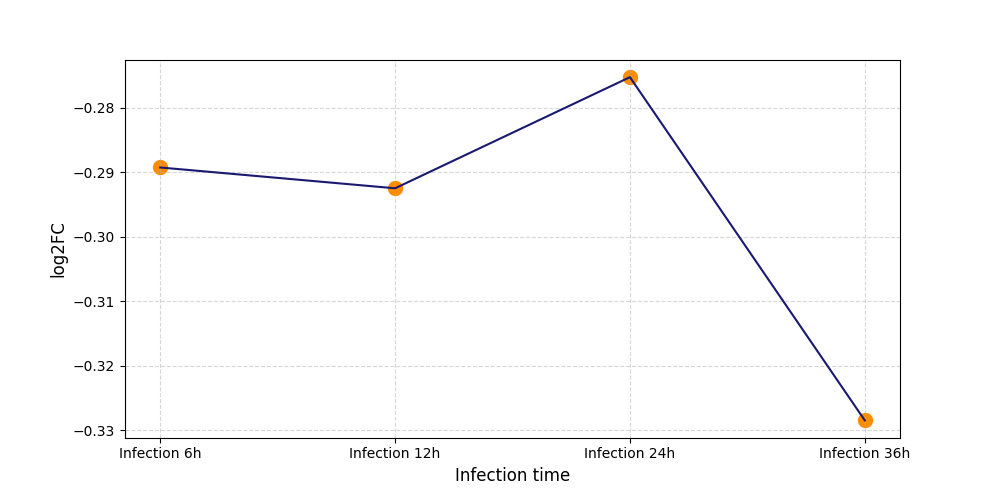
T2421
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2423
[5] |
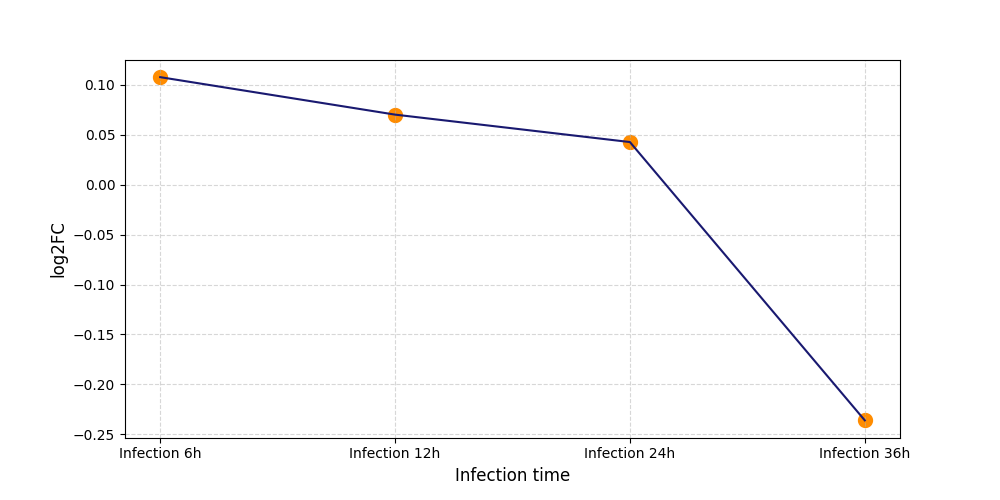 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2460
[5] |
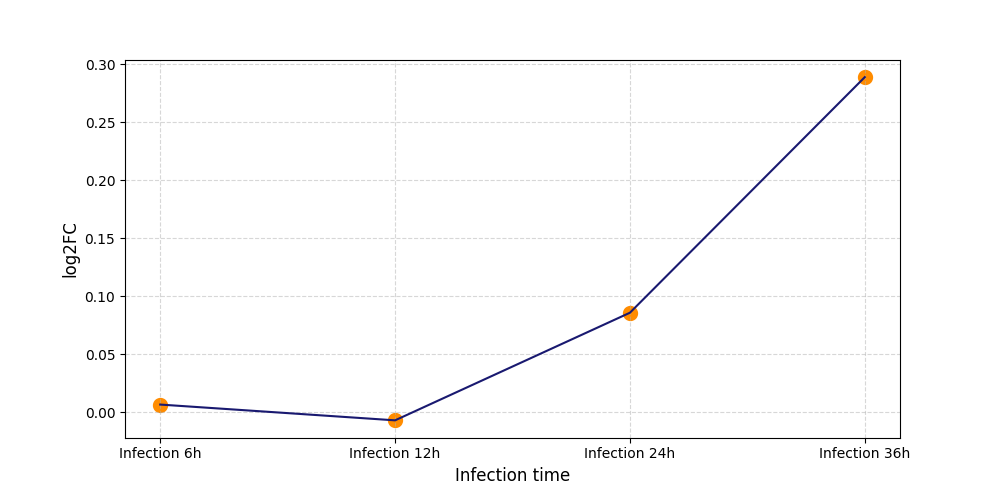 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T3139
[5] |
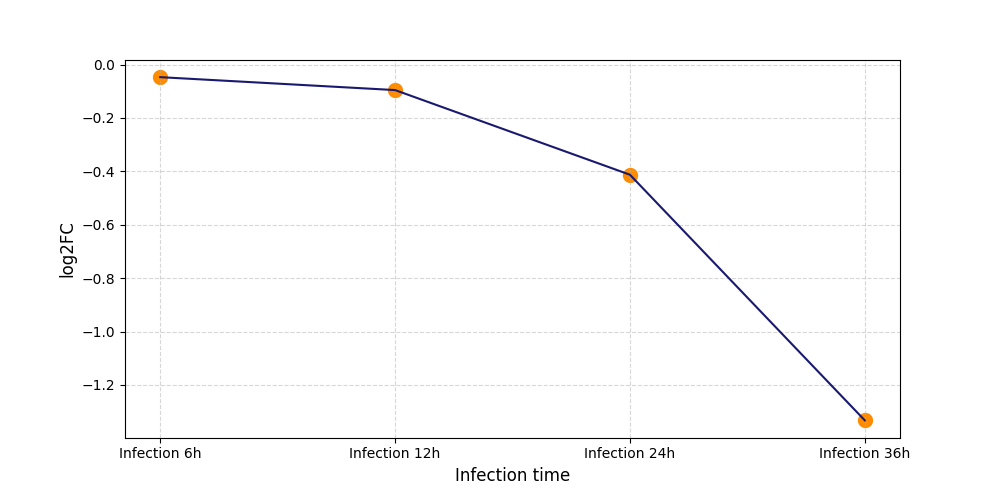 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Sequence Information |
MVRETRHLWVGNLPENVREEKIIEHFKRYGRVESVKILPKRGSEGGVAAFVDFVDIKSAQKAHNSVNKMGDRDLRTDYNEPGTIPSAARGLDDTVSIASRSREVSGFRGGGGGPAYGPPPSLHAREGRYERRLDGASDNRERAYEHSAYGHHERGTGGFDRTRHYDQDYYRDPRERTLQHGLYYASRSRSPNRFDAHDPRYEPRAREQFTLPSVVHRDIYRDDITREVRGRRPERNYQHSRSRSPHSSQSRNQSPQRLASQASRPTRSPSGSGSRSRSSSSDSISSSSSTSSDSSDSSSSSSDDSPARSVQSAAVPAPTSQLLSSLEKDEPRKSFGIKVQNLPVRSTDTSLKDGLFHEFKKFGKVTSVQIHGTSEERYGLVFFRQQEDQEKALTASKGKLFFGMQIEVTAWIGPETESENEFRPLDERIDEFHPKATRTLFIGNLEKTTTYHDLRNIFQRFGEIVDIDIKKVNGVPQYAFLQYCDIASVCKAIKKMDGEYLGNNRLKLGFGKSMPTNCVWLDGLSSNVSDQYLTRHFCRYGPVVKVVFDRLKGMALVLYNEIEYAQAAVKETKGRKIGGNKIKVDFANRESQLAFYHCMEKSGQDIRDFYEMLAERREERRASYDYNQDRTYYESVRTPGTYPEDSRRDYPARGREFYSEWETYQGDYYESRYYDDPREYRDYRNDPYEQDIREYSYRQRERERERERFESDRDRDHERRPIERSQSPVHLRRPQSPGASPSQAERLPSDSERRLYSRSSDRSGSCSSLSPPRYEKLDKSRLERYTKNEKTDKERTFDPERVERERRLIRKEKVEKDKTDKQKRKGKVHSPSSQSSETDQENEREQSPEKPRSCNKLSREKADKEGIAKNRLELMPCVVLTRVKEKEGKVIDHTPVEKLKAKLDNDTVKSSALDQKLQVSQTEPAKSDLSKLESVRMKVPKEKGLSSHVEVVEKEGRLKARKHLKPEQPADGVSAVDLEKLEARKRRFADSNLKAEKQKPEVKKSSPEMEDARVLSKKQPDVSSREVILLREGEAERKPVRKEILKRESKKIKLDRLNTVASPKDCQELASISVGSGSRPSSDLQARLGELAGESVENQEVQSKKPIPSKPQLKQLQVLDDQGPEREDVRKNYCSLRDETPERKSGQEKSHSVNTEEKIGIDIDHTQSYRKQMEQSRRKQQMEMEIAKSEKFGSPKKDVDEYERRSLVHEVGKPPQDVTDDSPPSKKKRMDHVDFDICTKRERNYRSSRQISEDSERTGGSPSVRHGSFHEDEDPIGSPRLLSVKGSPKVDEKVLPYSNITVREESLKFNPYDSSRREQMADMAKIKLSVLNSEDELNRWDSQMKQDAGRFDVSFPNSIIKRDSLRKRSVRDLEPGEVPSDSDEDGEHKSHSPRASALYESSRLSFLLRDREDKLRERDERLSSSLERNKFYSFALDKTITPDTKALLERAKSLSSSREENWSFLDWDSRFANFRNNKDKEKVDSAPRPIPSWYMKKKKIRTDSEGKMDDKKEDHKEEEQERQELFASRFLHSSIFEQDSKRLQHLERKEEDSDFISGRIYGKQTSEGANSTTDSIQEPVVLFHSRFMELTRMQQKEKEKDQKPKEVEKQEDTENHPKTPESAPENKDSELKTPPSVGPPSVTVVTLESAPSALEKTTGDKTVEAPLVTEEKTVEPATVSEEAKPASEPAPAPVEQLEQVDLPPGADPDKEAAMMPAGVEEGSSGDQPPYLDAKPPTPGASFSQAESNVDPEPDSTQPLSKPAQKSEEANEPKAEKPDATADAEPDANQKAEAAPESQPPASEDLEVDPPVAAKDKKPNKSKRSKTPVQAAAVSIVEKPVTRKSERIDREKLKRSNSPRGEAQKLLELKMEAEKITRTASKNSAADLEHPEPSLPLSRTRRRNVRSVYATMGDHENRSPVKEPVEQPRVTRKRLERELQEAAAVPTTPRRGRPPKTRRRADEEEENEAKEPAETLKPPEGWRSPRSQKTAAGGGPQGKKGKNEPKVDATRPEATTEVGPQIGVKESSMEPKAAEEEAGSEQKRDRKDAGTDKNPPETAPVEVVEKKPAPEKNSKSKRGRSRNSRLAVDKSASLKNVDAAVSPRGAAAQAGERESGVVAVSPEKSESPQKEDGLSSQLKSDPVDPDKEPEKEDVSASGPSPEATQLAKQMELEQAVEHIAKLAEASASAAYKADAPEGLAPEDRDKPAHQASETELAAAIGSIINDISGEPENFPAPPPYPGESQTDLQPPAGAQALQPSEEGMETDEAVSGILETEAATESSRPPVNAPDPSAGPTDTKEARGNSSETSHSVPEAKGSKEVEVTLVRKDKGRQKTTRSRRKRNTNKKVVAPVESHVPESNQAQGESPAANEGTTVQHPEAPQEEKQSEKPHSTPPQSCTSDLSKIPSTENSSQEISVEERTPTKASVPPDLPPPPQPAPVDEEPQARFRVHSIIESDPVTPPSDPSIPIPTLPSVTAAKLSPPVASGGIPHQSPPTKVTEWITRQEEPRAQSTPSPALPPDTKASDVDTSSSTLRKILMDPKYVSATSVTSTSVTTAIAEPVSAAPCLHEAPPPPVDSKKPLEEKTAPPVTNNSEIQASEVLVAADKEKVAPVIAPKITSVISRMPVSIDLENSQKITLAKPAPQTLTGLVSALTGLVNVSLVPVNALKGPVKGSVTTLKSLVSTPAGPVNVLKGPVNVLTGPVNVLTTPVNATVGTVNAAPGTVNAAASAVNATASAVTVTAGAVTAASGGVTATTGTVTMAGAVIAPSTKCKQRASANENSRFHPGSMPVIDDRPADAGSGAGLRVNTSEGVVLLSYSGQKTEGPQRISAKISQIPPASAMDIEFQQSVSKSQVKPDSVTASQPPSKGPQAPAGYANVATHSTLVLTAQTYNASPVISSVKADRPSLEKPEPIHLSVSTPVTQGGTVKVLTQGINTPPVLVHNQLVLTPSIVTTNKKLADPVTLKIETKVLQPANLGSTLTPHHPPALPSKLPTEVNHVPSGPSIPADRTVSHLAAAKLDAHSPRPSGPGPSSFPRASHPSSTASTALSTNATVMLAAGIPVPQFISSIHPEQSVIMPPHSITQTVSLSHLSQGEVRMNTPTLPSITYSIRPEALHSPRAPLQPQQIEVRAPQRASTPQPAPAGVPALASQHPPEEEVHYHLPVARATAPVQSEVLVMQSEYRLHPYTVPRDVRIMVHPHVTAVSEQPRAADGVVKVPPASKAPQQPGKEAAKTPDAKAAPTPTPAPVPVPVPLPAPAPAPHGEARILTVTPSNQLQGLPLTPPVVVTHGVQIVHSSGELFQEYRYGDIRTYHPPAQLTHTQFPAASSVGLPSRTKTAAQGPPPEGEPLQPPQPVQSTQPAQPAPPCPPSQLGQPGQPPSSKMPQVSQEAKGTQTGVEQPRLPAGPANRPPEPHTQVQRAQAETGPTSFPSPVSVSMKPDLPVSLPTQTAPKQPLFVPTTSGPSTPPGLVLPHTEFQPAPKQDSSPHLTSQRPVDMVQLLKKYPIVWQGLLALKNDTAAVQLHFVSGNNVLAHRSLPLSEGGPPLRIAQRMRLEATQLEGVARRMTVETDYCLLLALPCGRDQEDVVSQTESLKAAFITYLQAKQAAGIINVPNPGSNQPAYVLQIFPPCEFSESHLSRLAPDLLASISNISPHLMIVIASV
Click to Show/Hide
|
|---|