Details of Host Protein
| Host Protein General Information (ID: PT0809) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name |
Antigen KI-67 messenger RNA (MKI67)
|
Gene Name |
MKI67
|
||||||
| Host Species |
Homo sapiens
|
Uniprot Entry Name |
KI67_HUMAN
|
||||||
| Subcellular Location |
Chromosome Nucleus; nucleolus
|
||||||||
| External Link | |||||||||
| NCBI Gene ID | |||||||||
| Uniprot ID | |||||||||
| Ensembl ID | |||||||||
| HGNC ID | |||||||||
| Function in Host |
Required to maintain individual mitotic chromosomes dispersedin the cytoplasm following nuclear envelope disassembly. Associates with the surface of the mitoticchromosome, the perichromosomal layer, and covers a substantialfraction of the chromosome surface. Preventschromosomes from collapsing into a single chromatin mass by forming asteric and electrostatic charge barrier: the protein has a high netelectrical charge and acts as a surfactant, dispersing chromosomes andenabling independent chromosome motility. Binds DNA, with a preference for supercoiled DNA and AT-rich DNA. Does not contribute to the internal structure ofmitotic chromosomes. May play a role in chromatinorganization. It is however unclear whether it playsa direct role in chromatin organization or whether it is an indirectconsequence of its function in maintaining mitotic chromosomesdispersed.
[1-2]
Click to Show/Hide
|
||||||||
| 3D Structure |
|
||||||||
| Function of This Protein During Virus Infection | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Virus Name | SARS-COV-2 | Protein Function | Pro-viral | [3] | |||||
| Infected Tissue | Lung | Infection Time | 7-9 Days | ||||||
| Infected Cell | Calu-3 Cells (Human epithelial cell line) | Cellosaurus ID | CVCL_0609 | ||||||
| Method Description | To detect the role of host protein MKI67 in viral infection, MKI67 protein knockout Calu-3 Cells were infected with SARS-COV-2 for 7 - 9 Days , and the effects on infection was detected through CRISPR-based genome-wide gene-knockout screen. | ||||||||
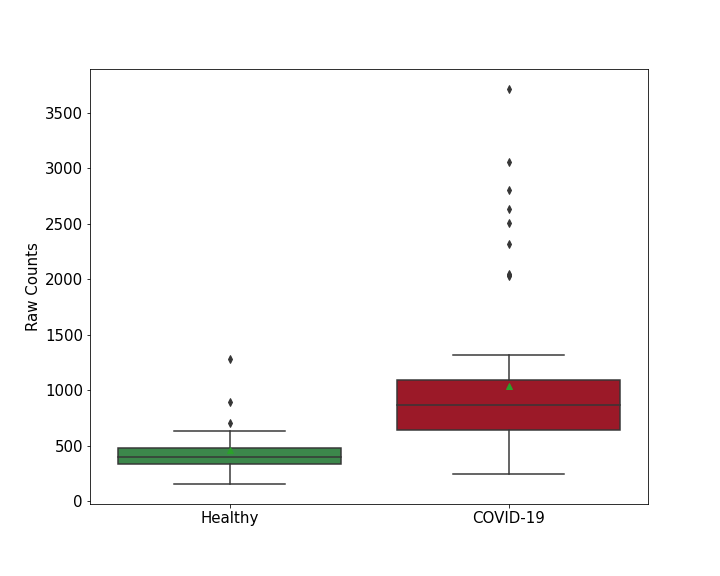
| Results | It is reported that knockout of MKI67 leads to the decreased SARS-CoV-2 RNA levels compared with control group. | ||||||||
| Full List of Virus RNA Interacting with This Protien | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| RNA Region: 5'-UTR (hCoV-19/Wuhan-Hu-1/2019 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[4] | |||||||
| Strains Name |
hCoV-19/Wuhan-Hu-1/2019
|
||||||||
| Strains Family |
Beta (B.1.351)
|
||||||||
| RNA Binding Region |
5'-UTR
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Infection Cells | HEK293 Cells (Human embryonic kidney cell) (CVCL_0045 ) | ||||||||
| Cell Originated Tissue | Liver | ||||||||
| Infection Time | 48 h | ||||||||
| Interaction Score | Prot score = 20 | ||||||||
| Method Description | RNA-protein interaction detection (RaPID) assay; liquid chromatography with tandem mass spectrometry (LC-MS/MS) | ||||||||
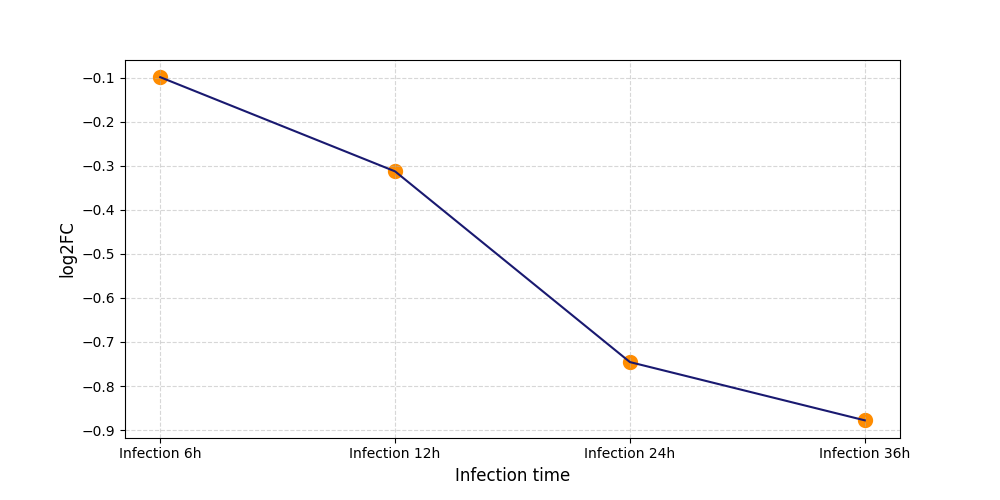
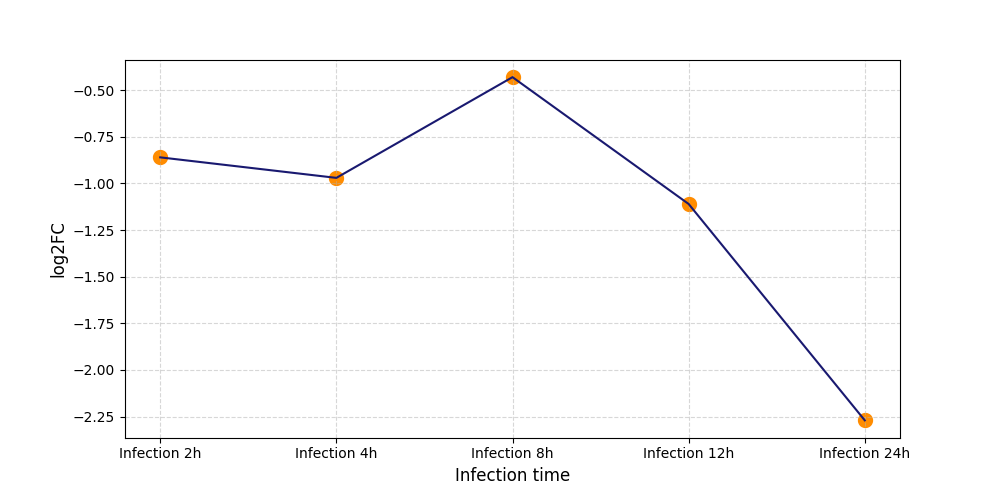
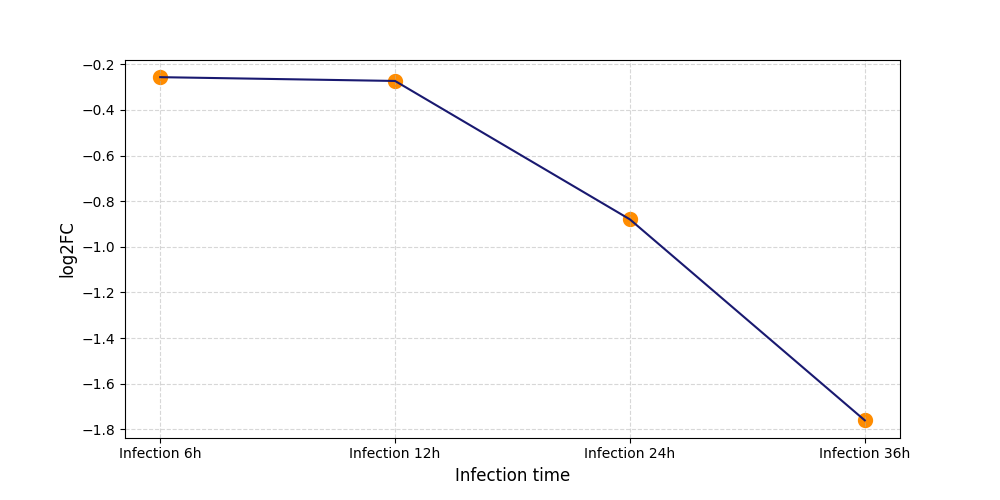
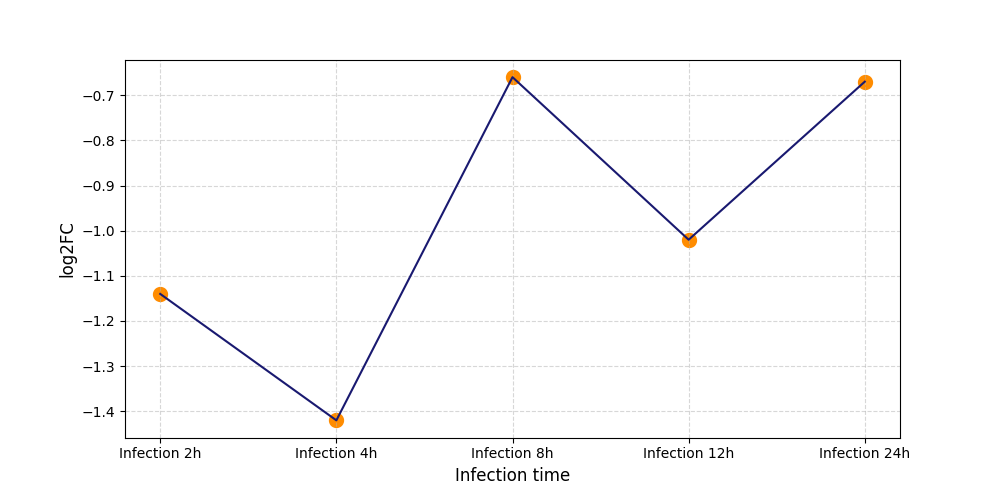
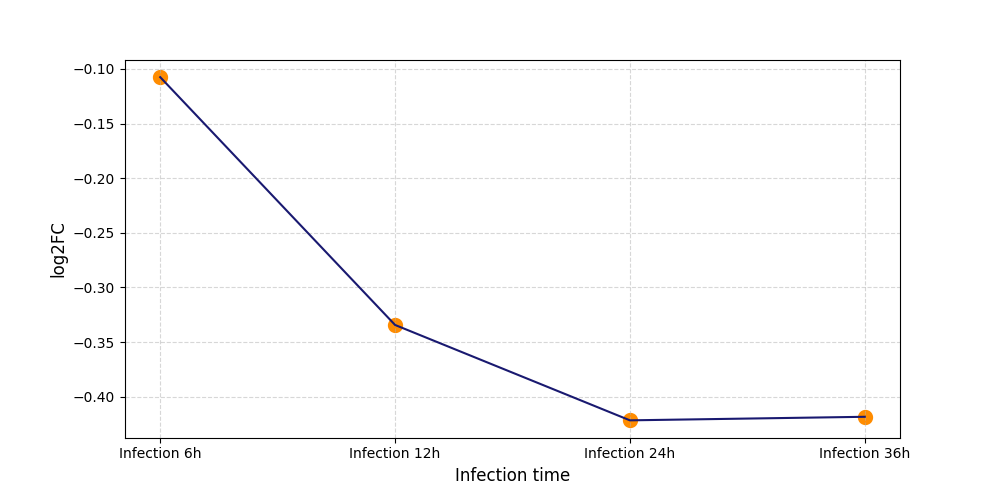
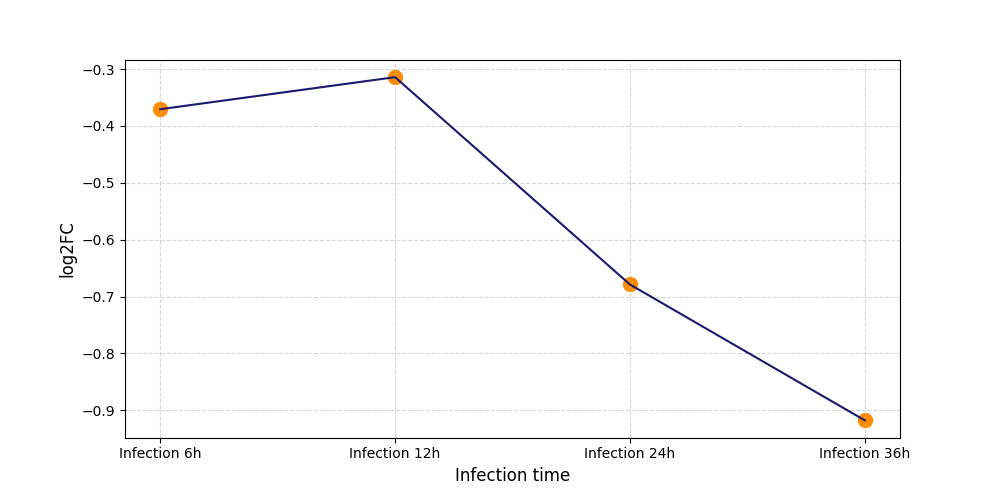
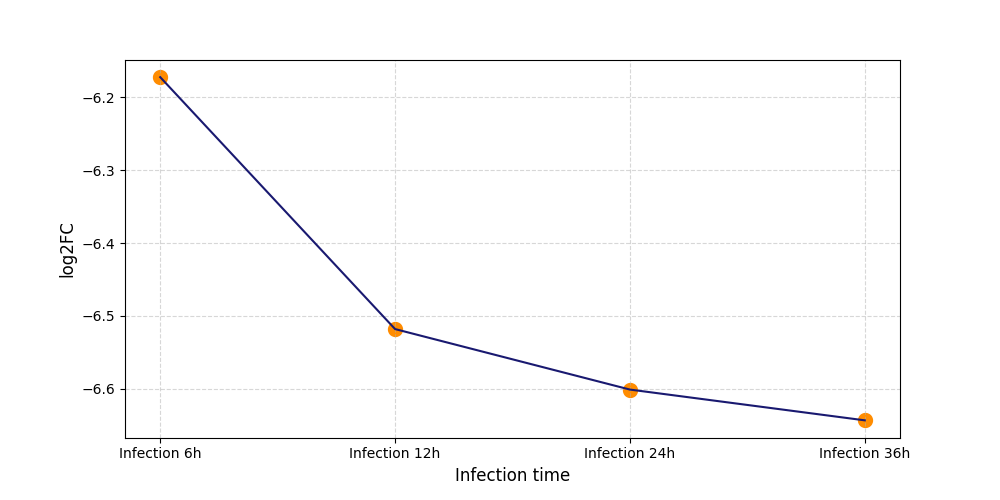
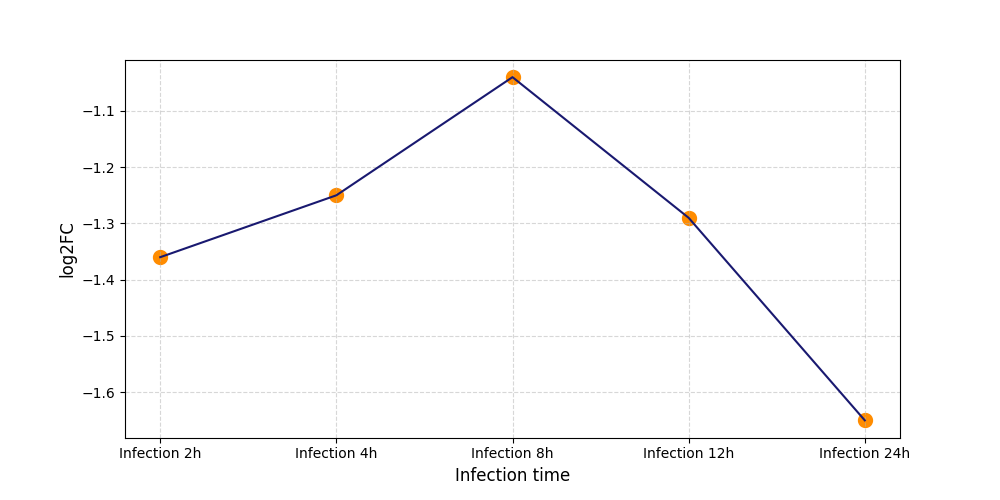
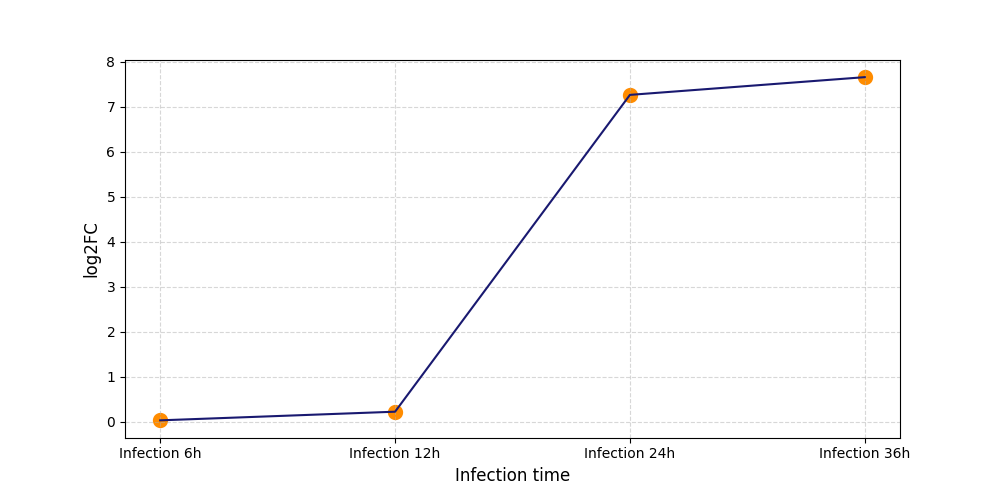
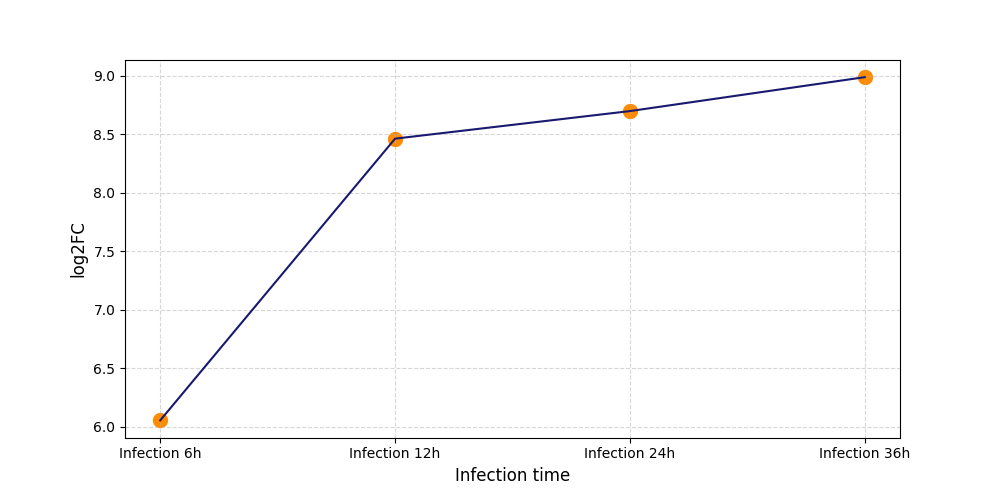
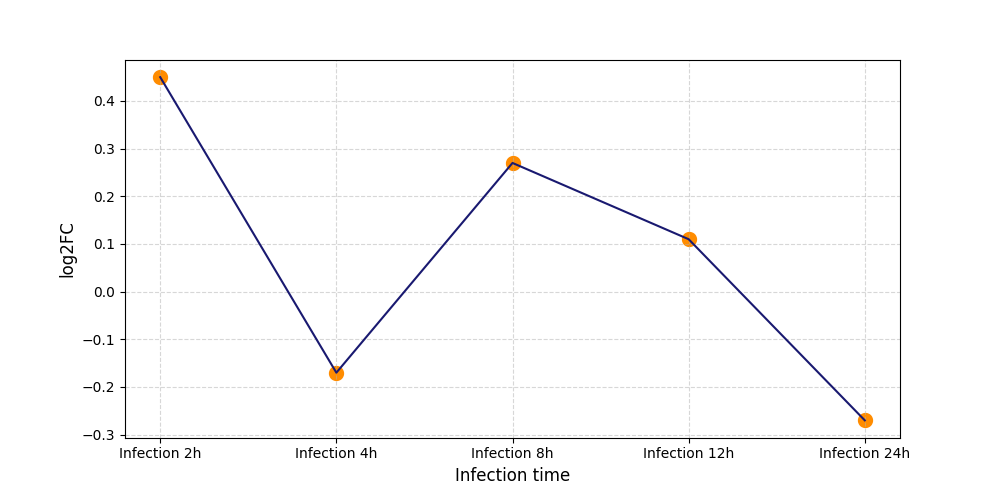
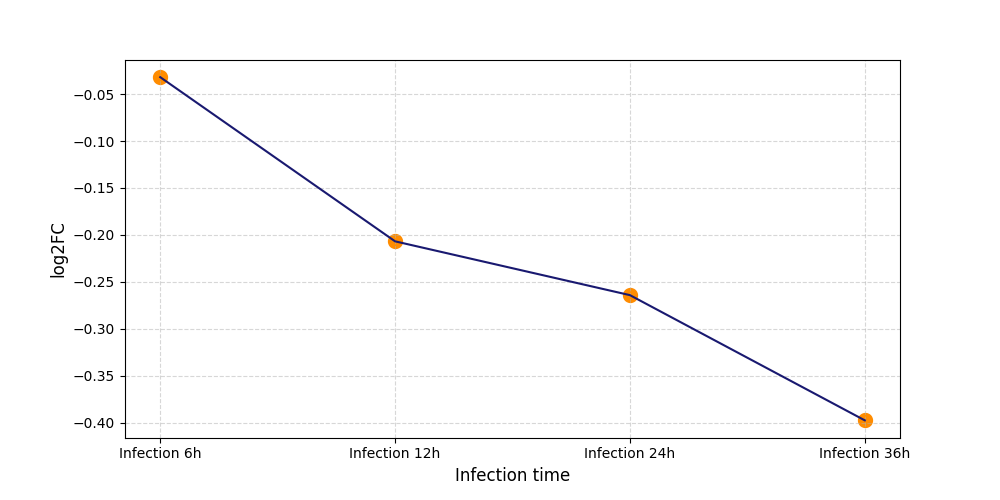
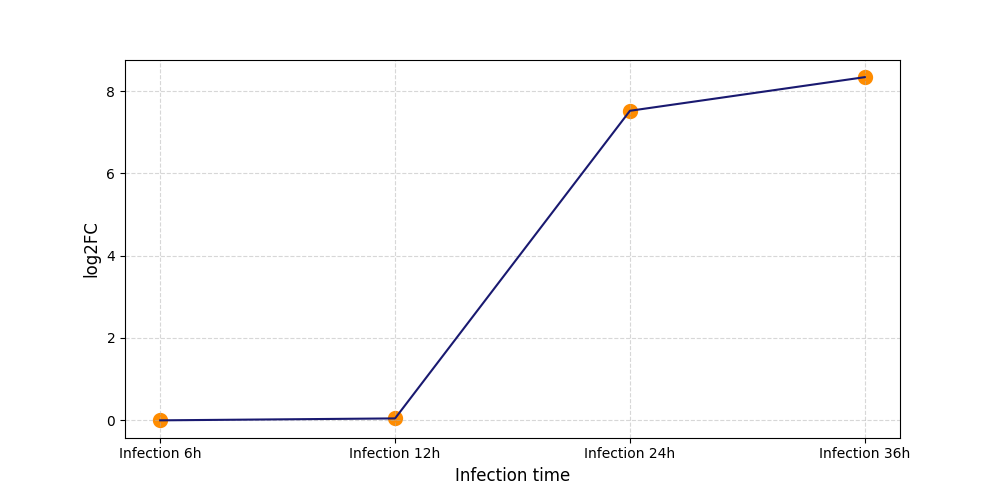
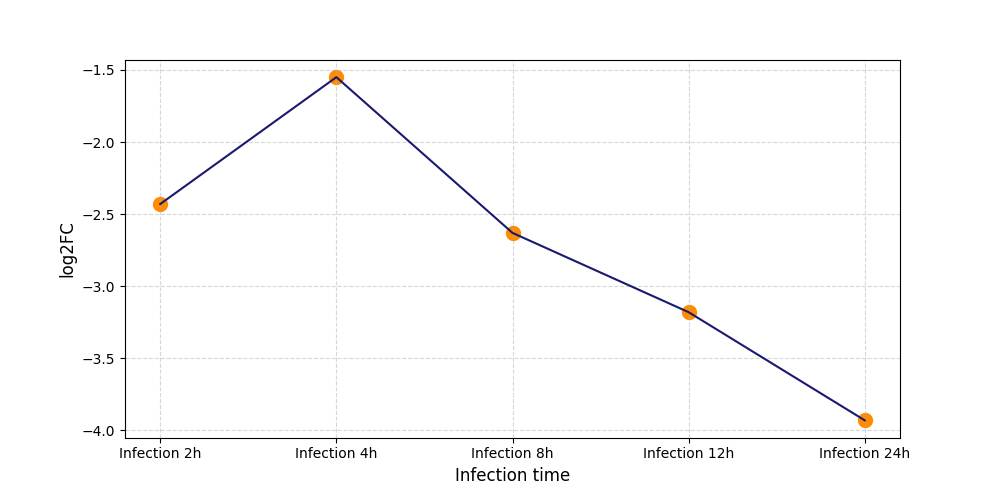
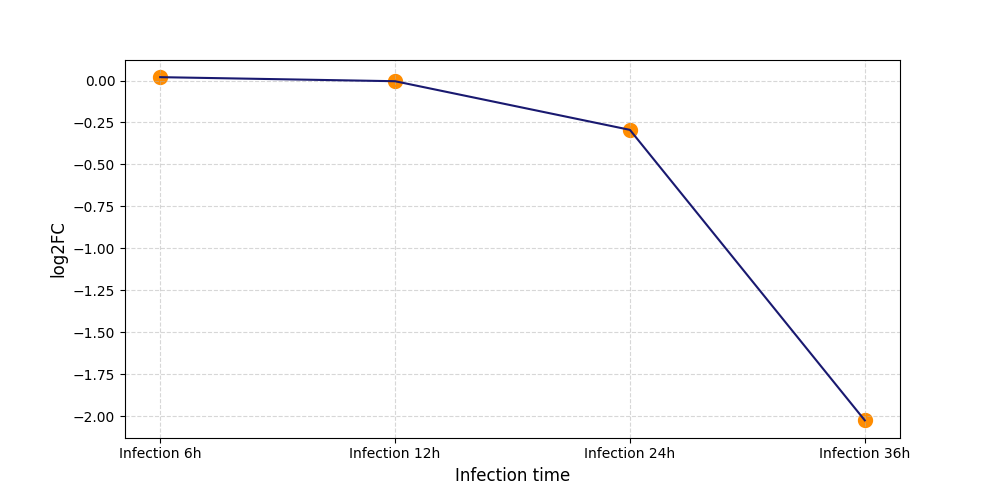
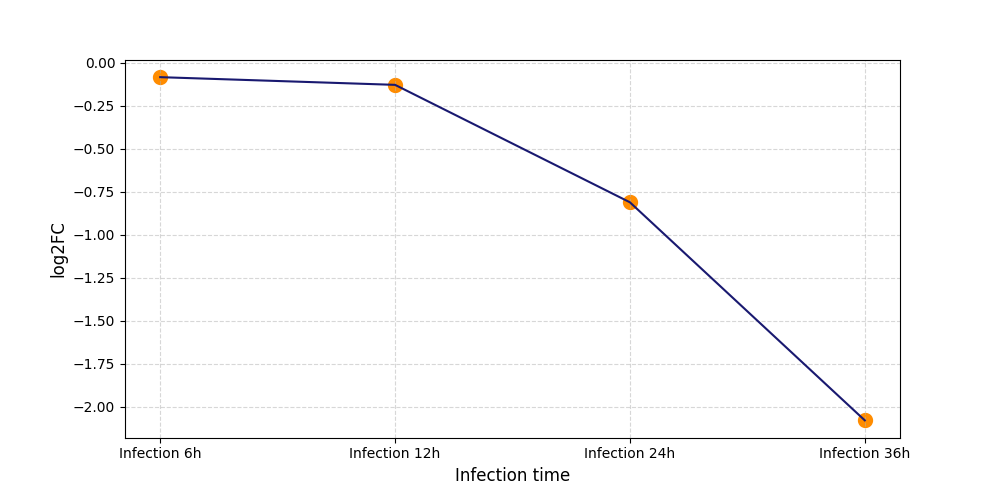
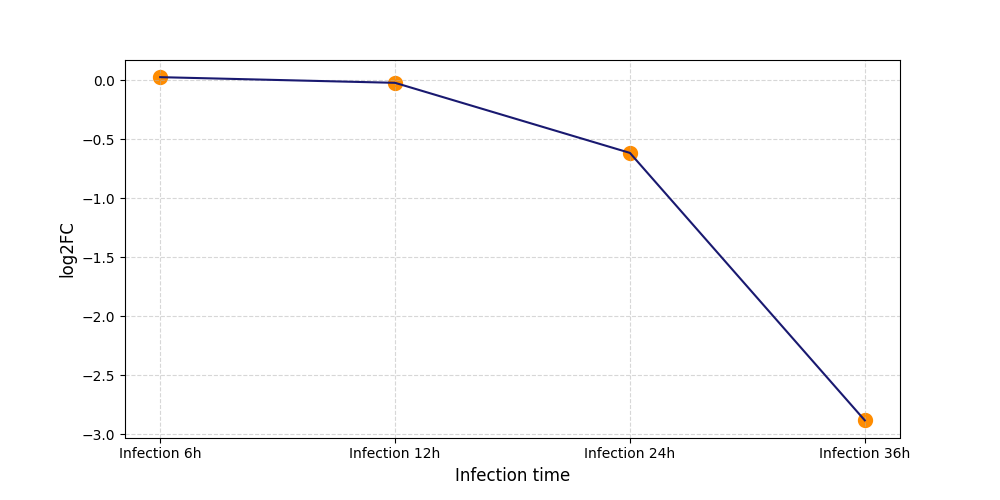
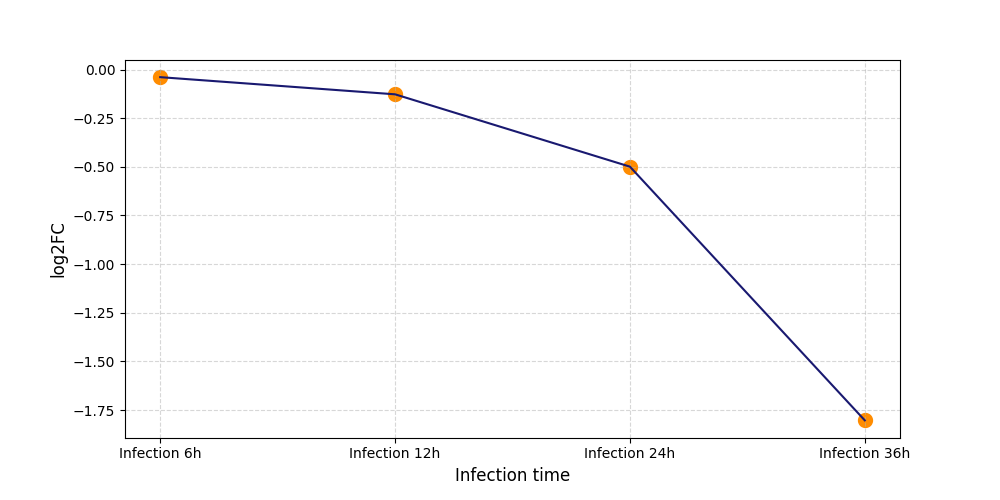
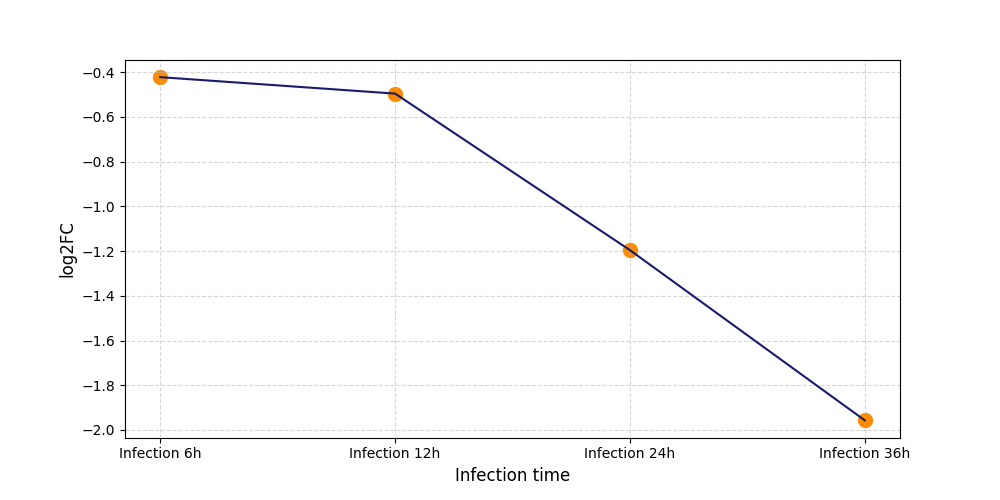
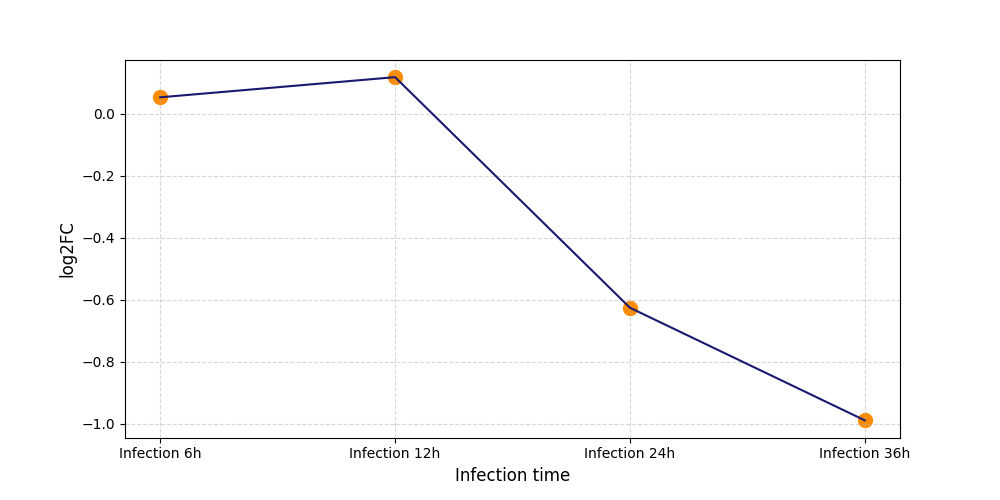
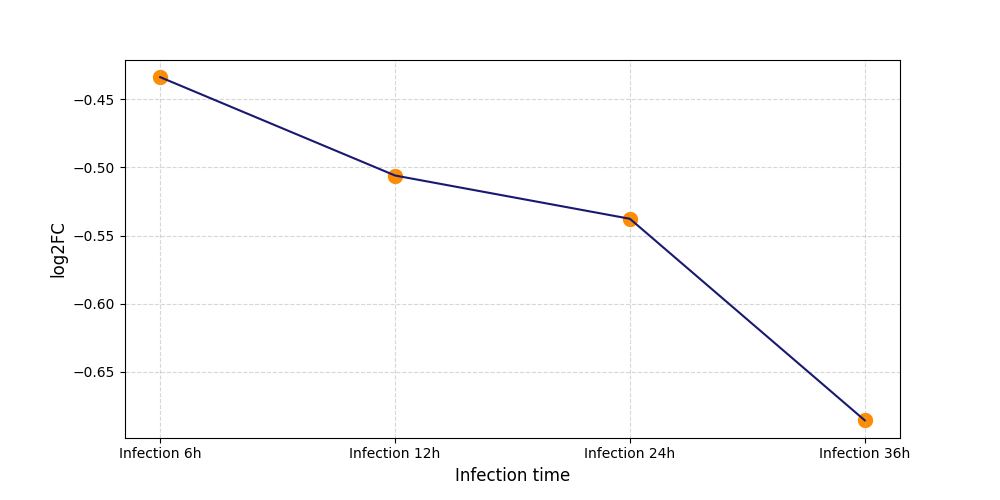
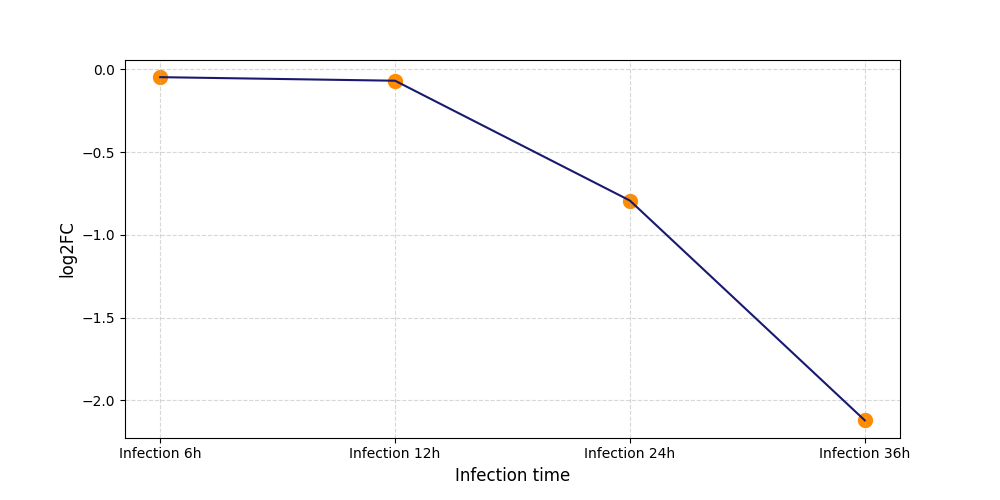
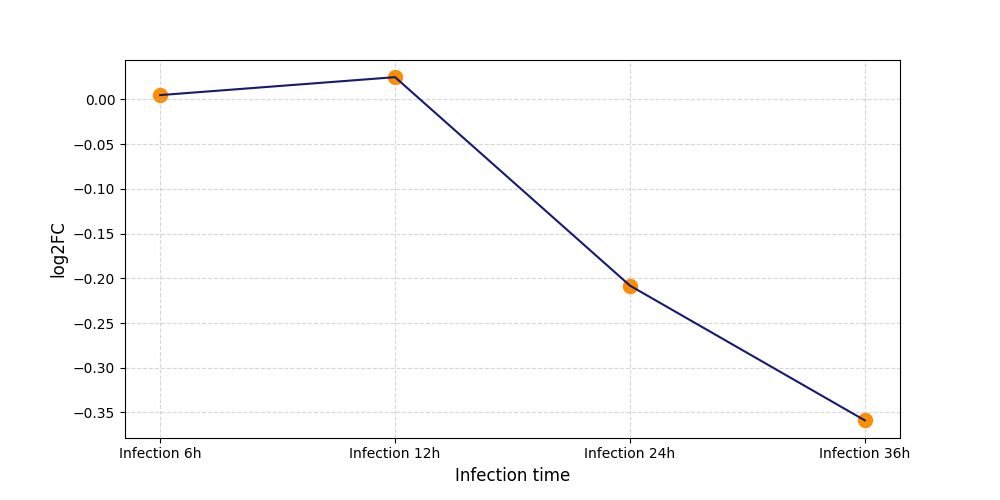
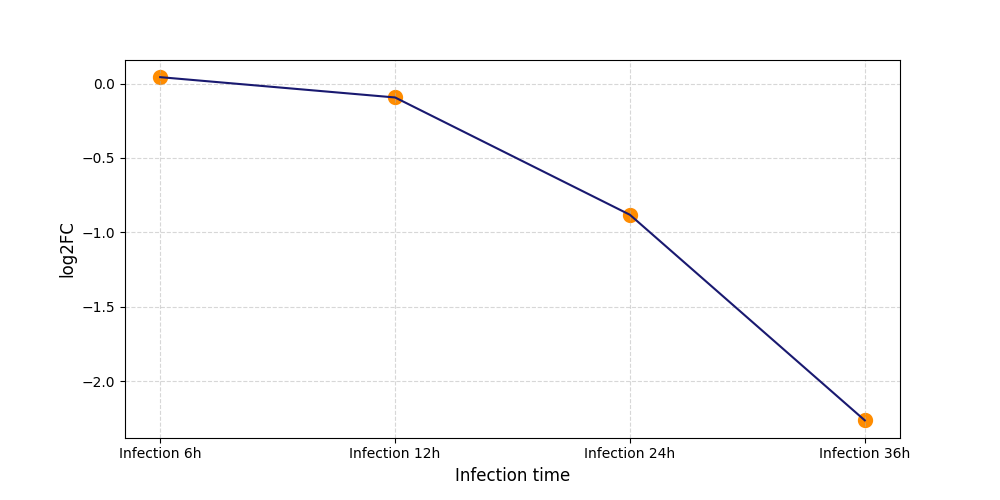
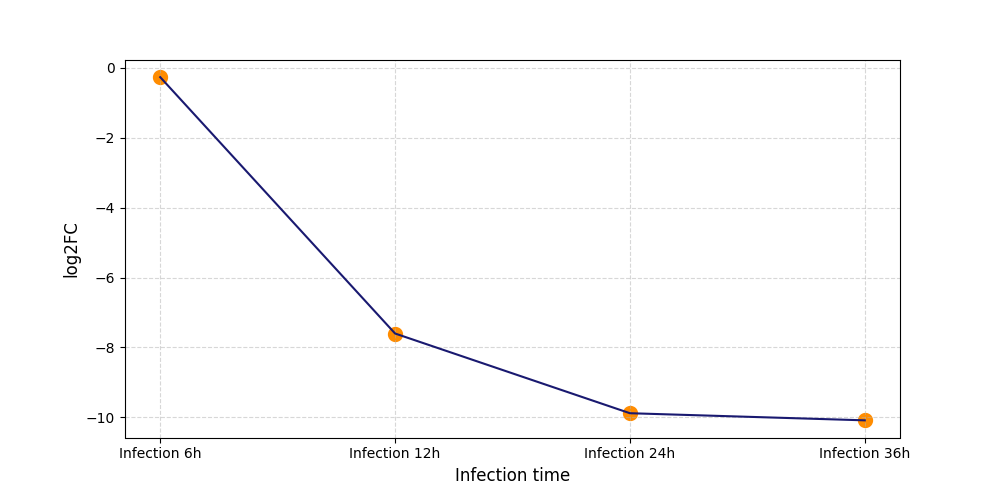
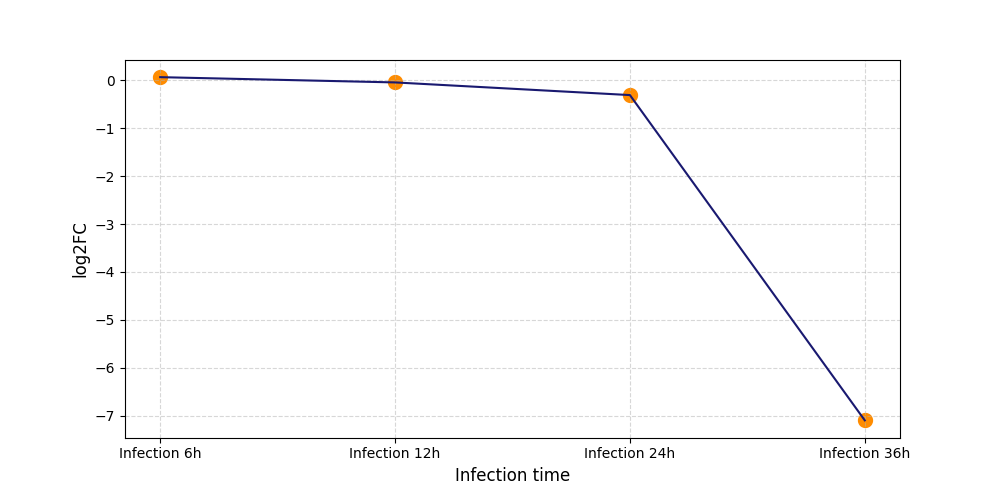
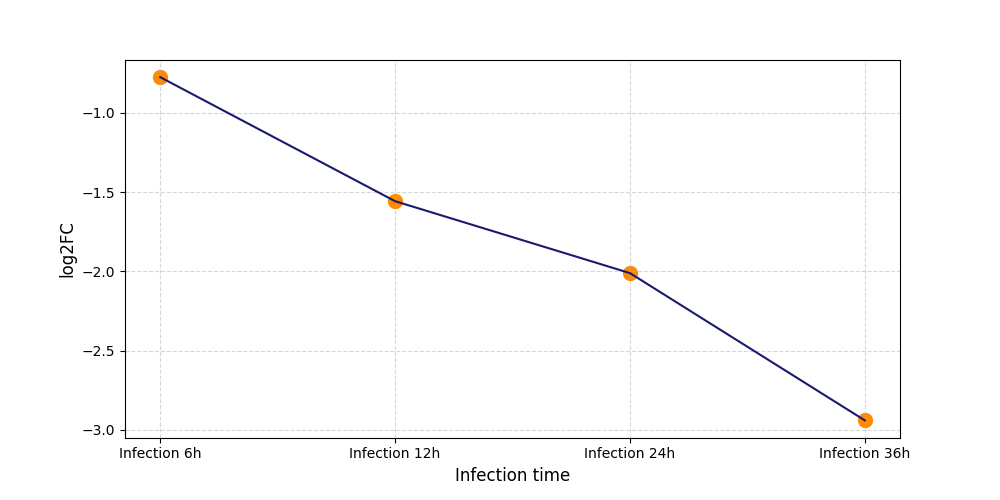
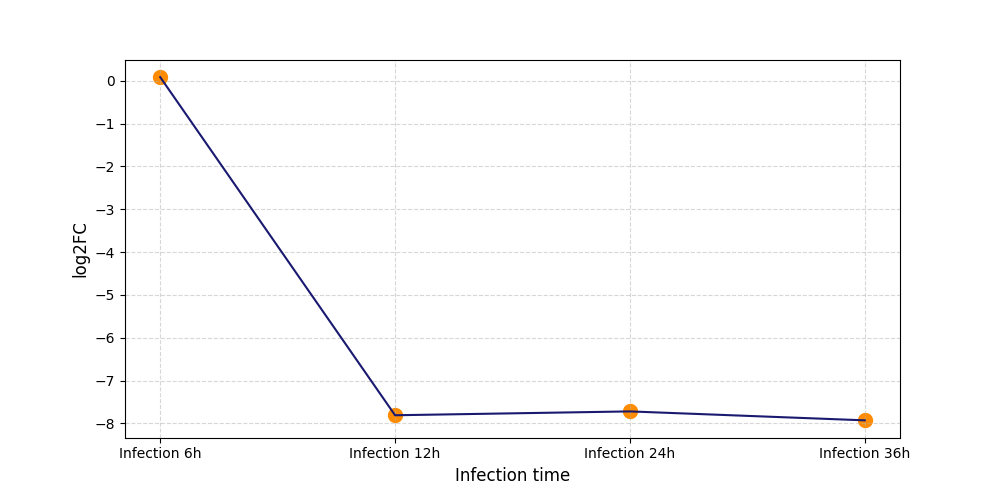
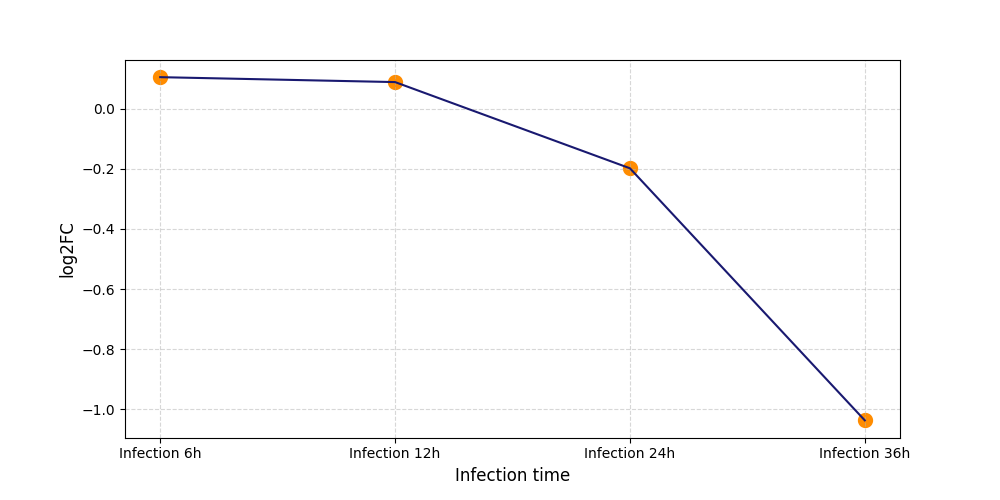
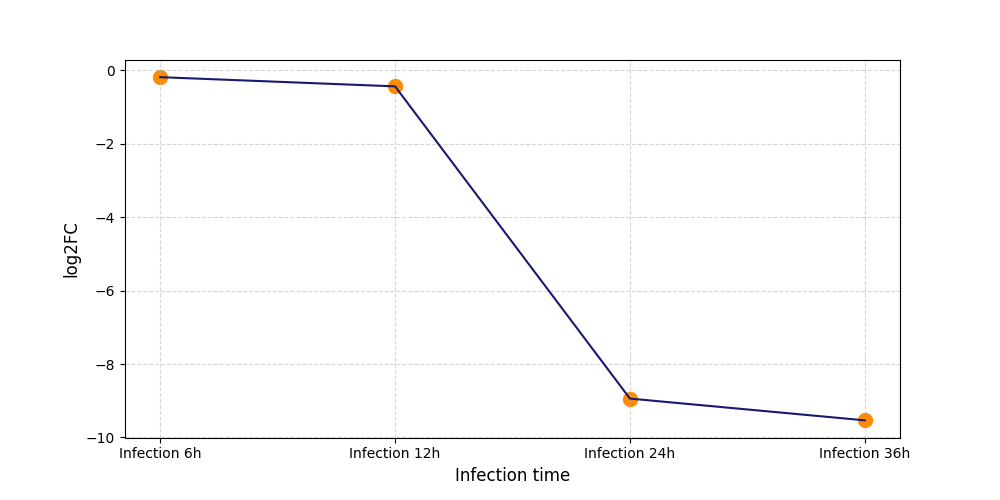
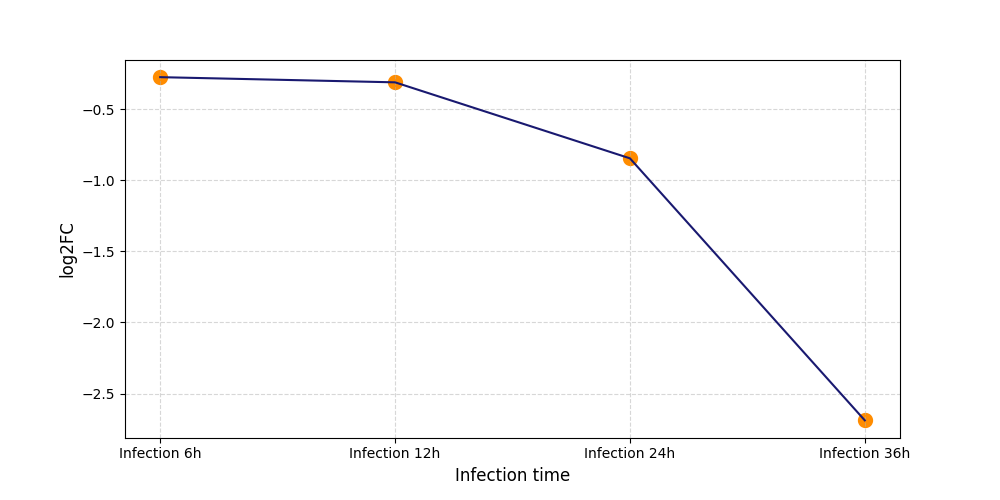
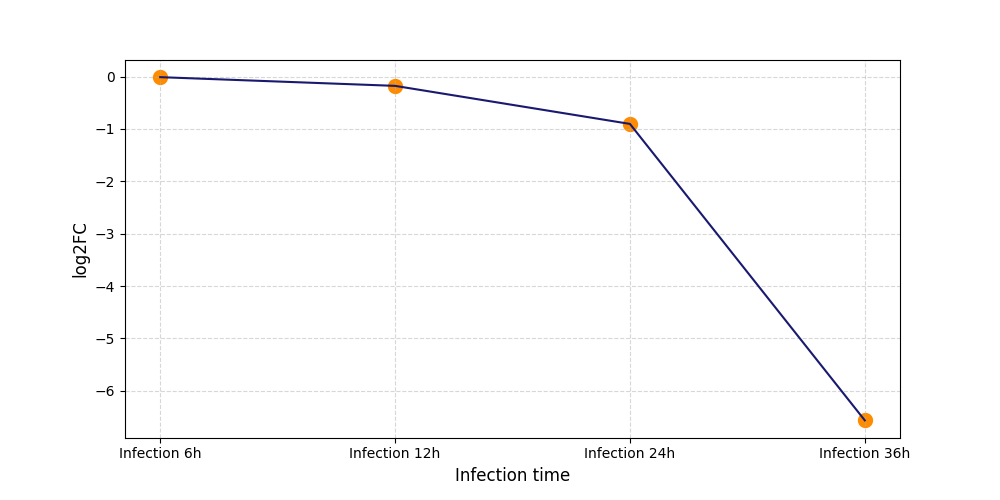
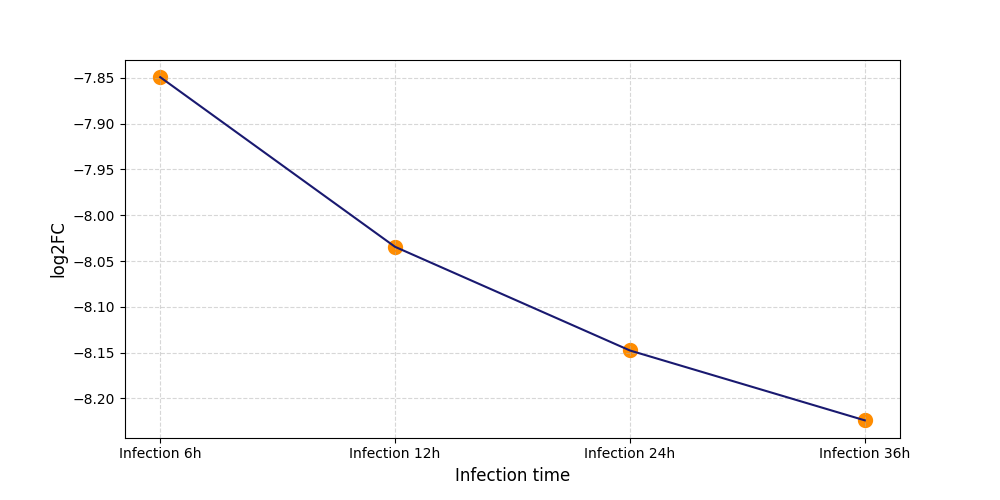
| Differential Gene Expression During SARS-COV-2 Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Phosphorylation after Virus Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
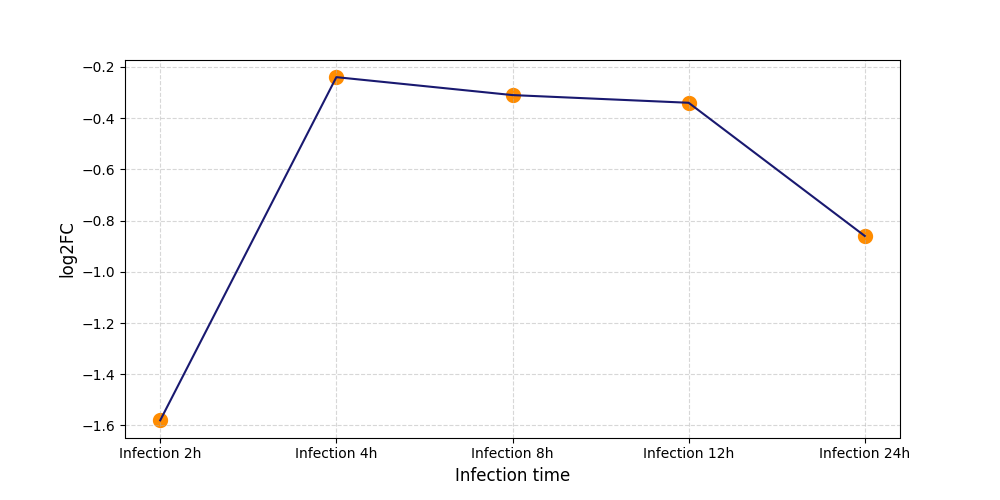
S1071
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
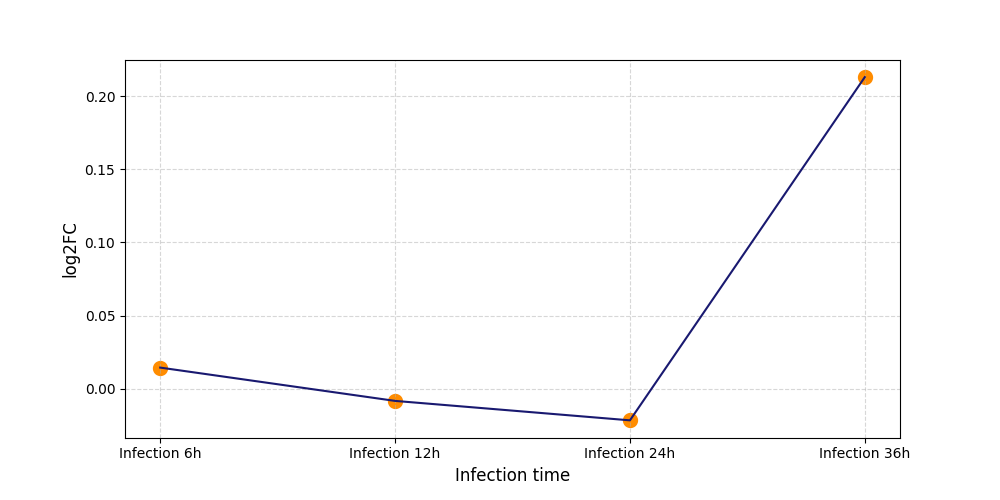
S1071
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1131
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1131
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
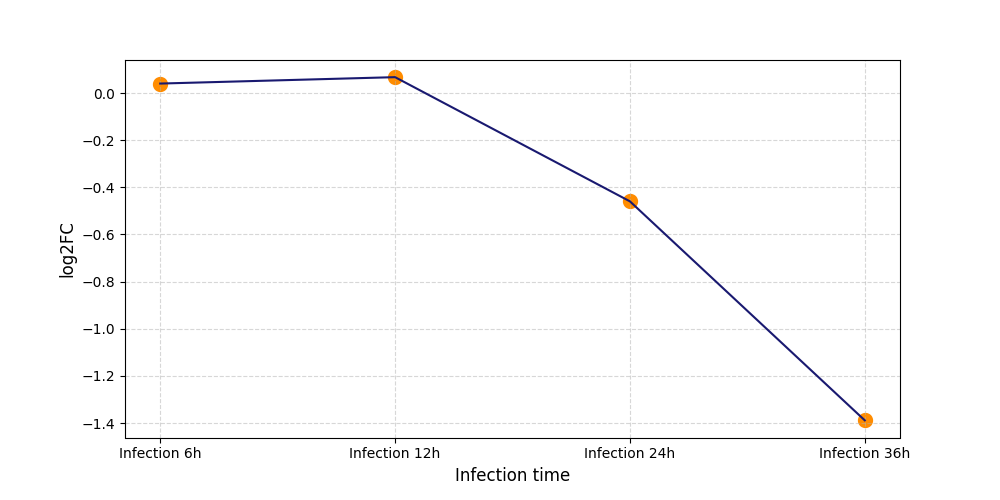
S1207
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
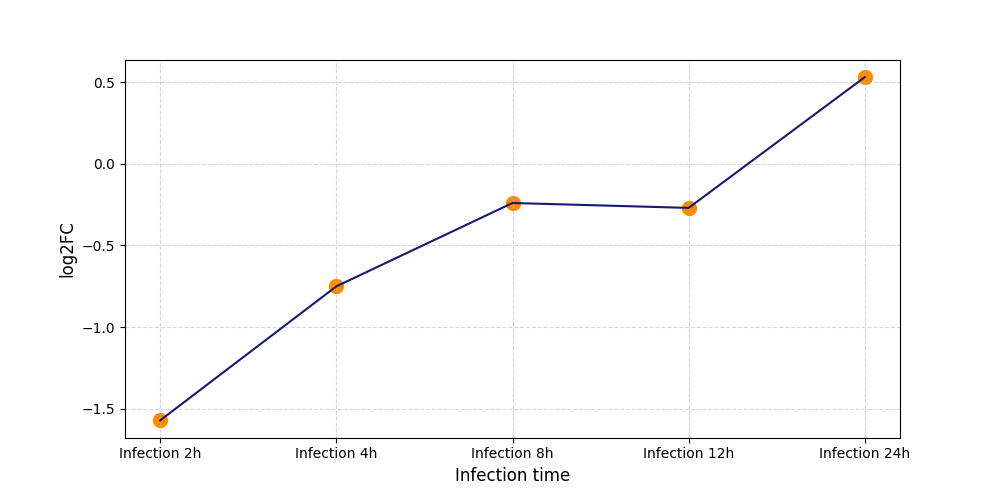
S1253
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
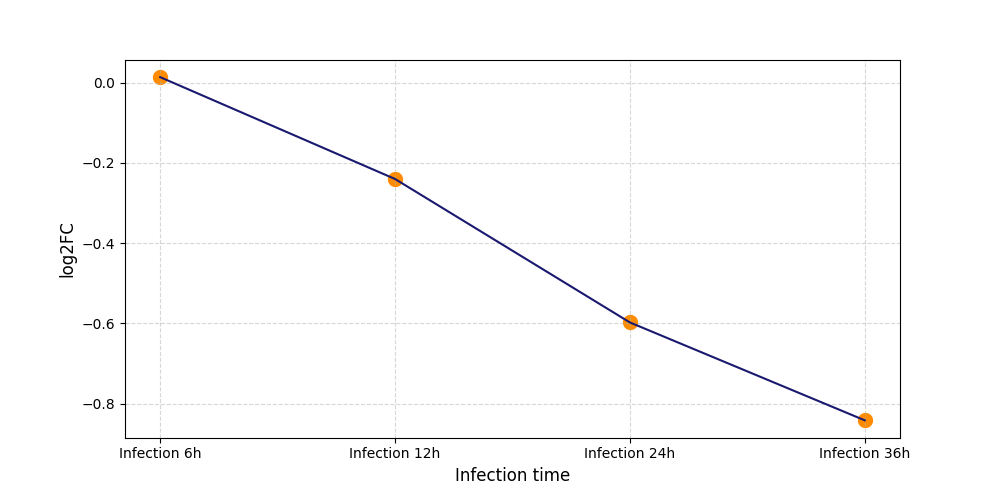
S1253
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
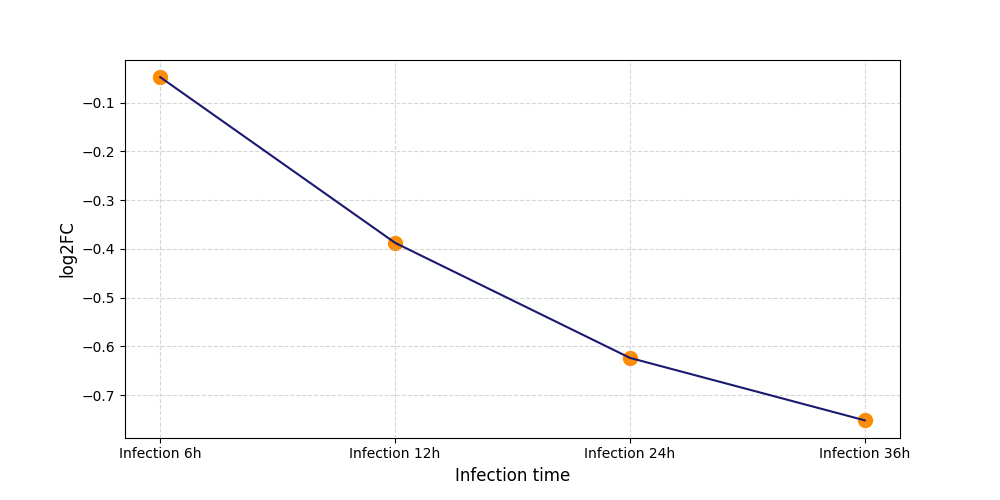
S1256
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
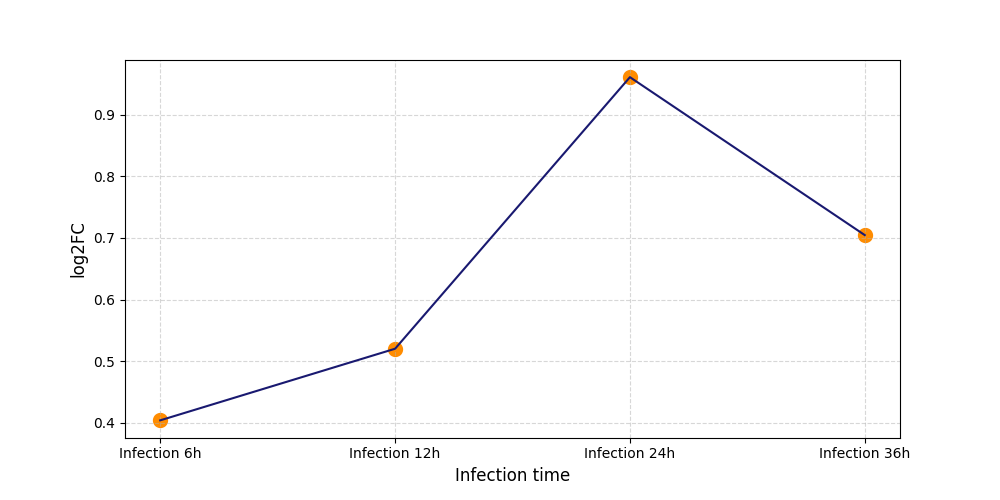
S128
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
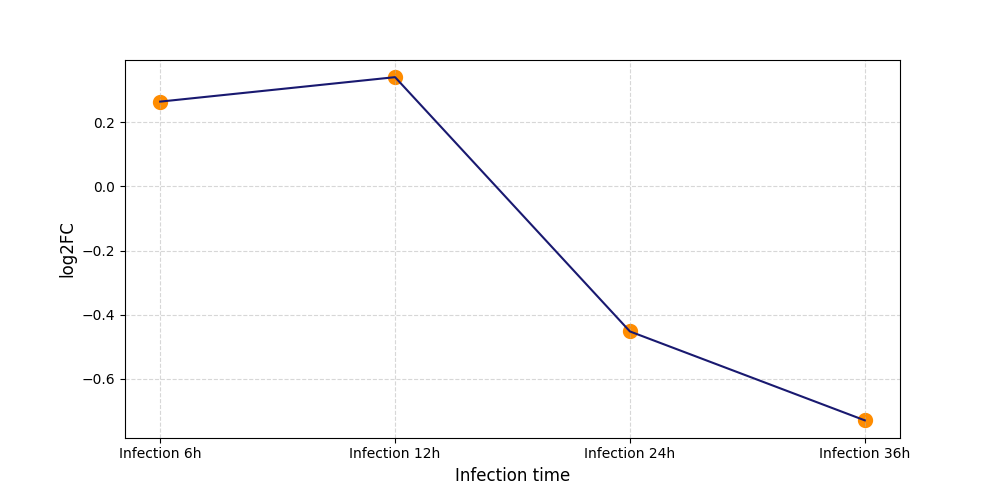
S1291
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1329
[6] |
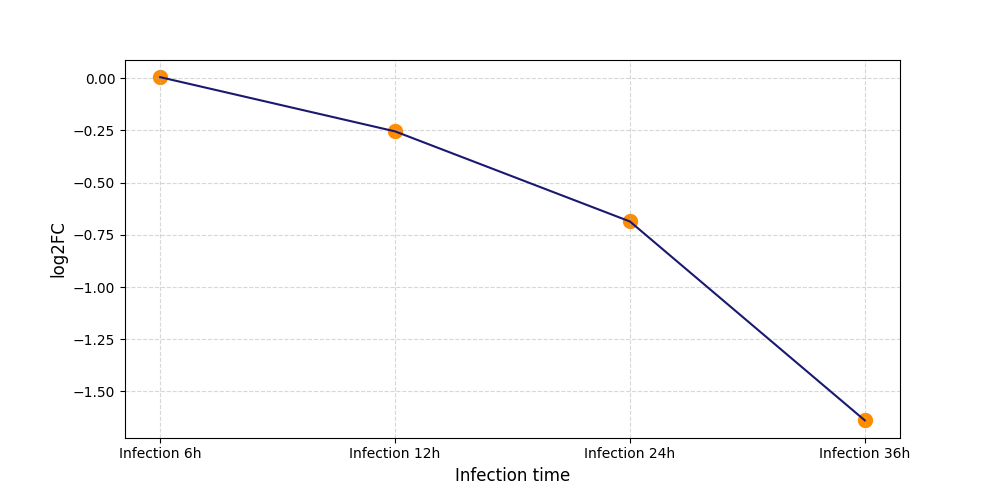 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1375
[6] |
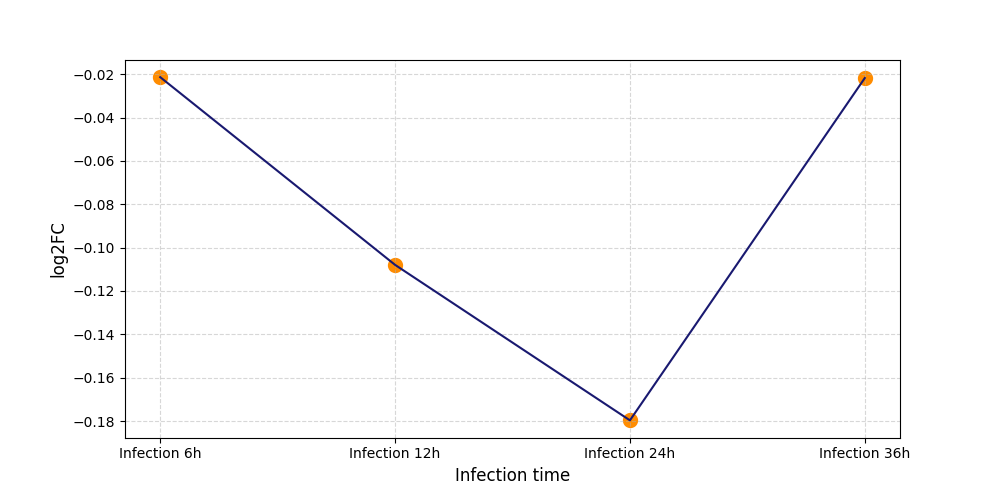 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1376
[5] |
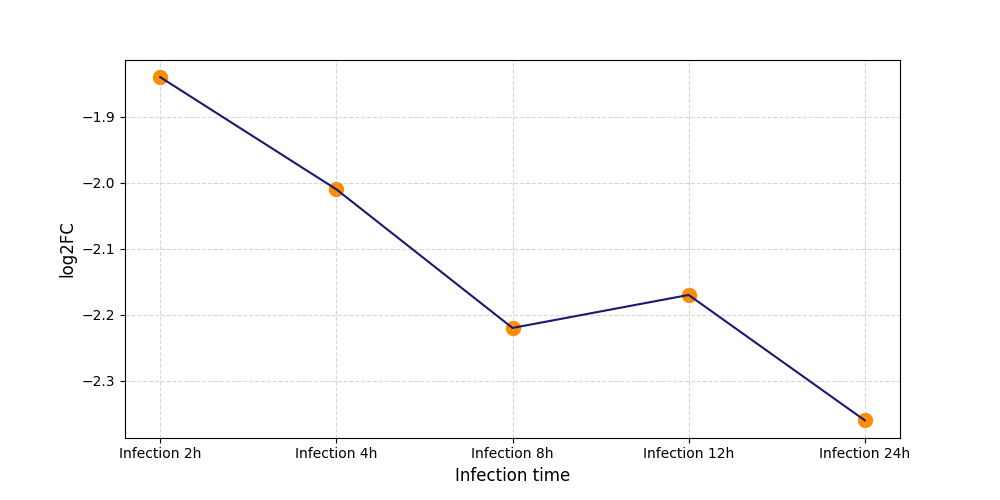 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1376
[6] |
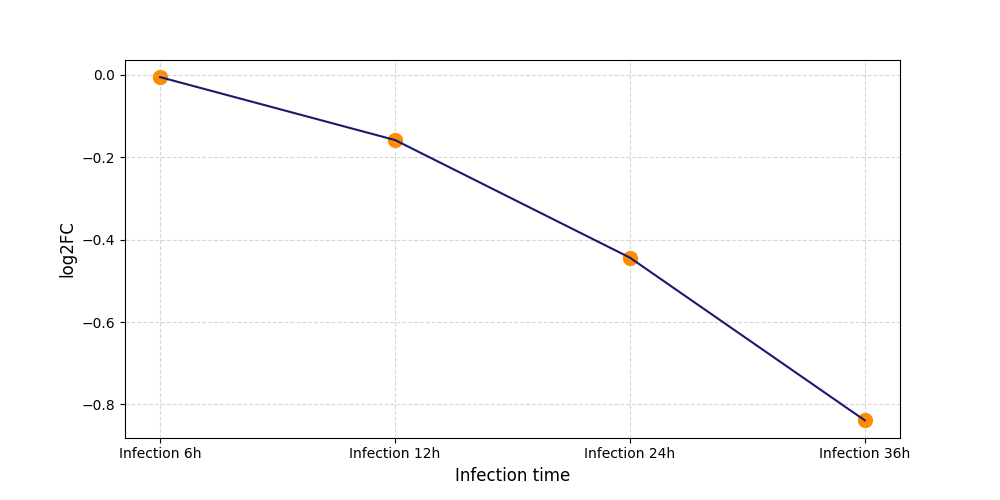 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
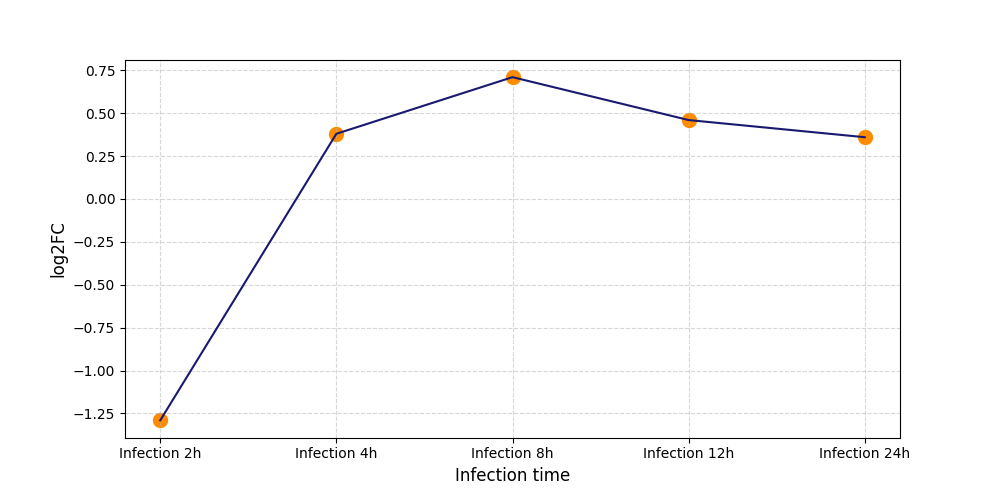
|
S1496
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
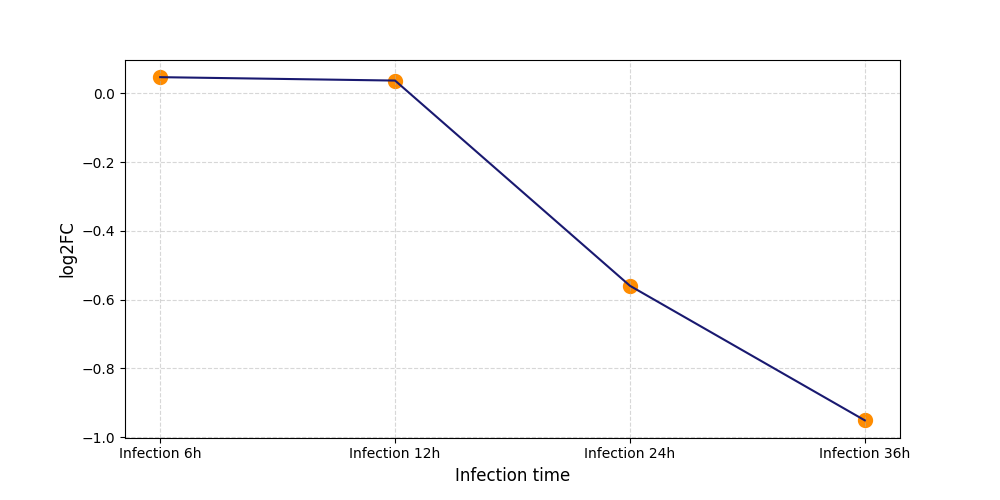
|
S1636
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
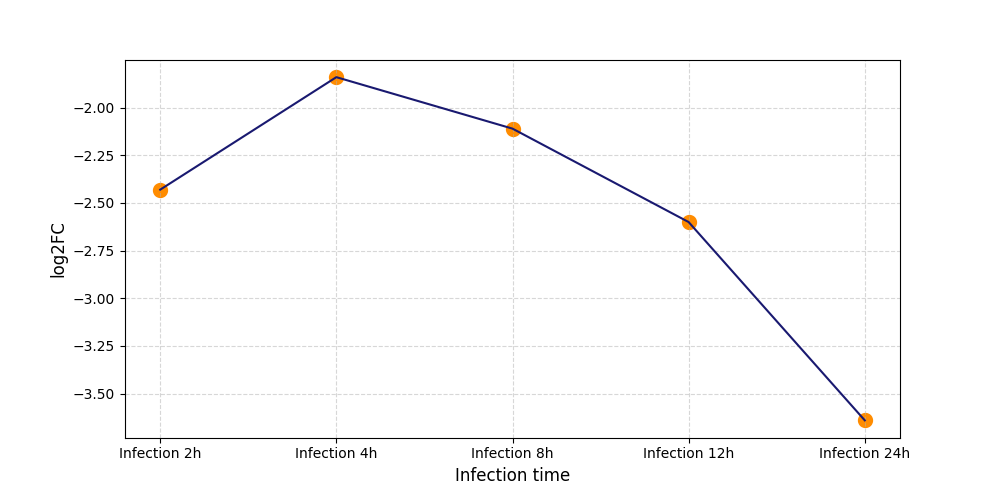
|
S1679
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
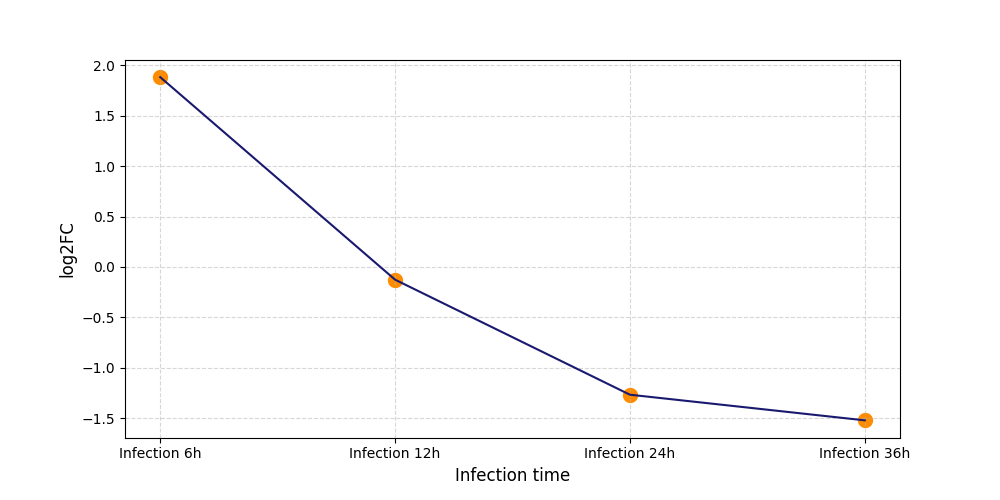
|
S1679
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1751
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1758
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1815
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1861
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1861
[6] |
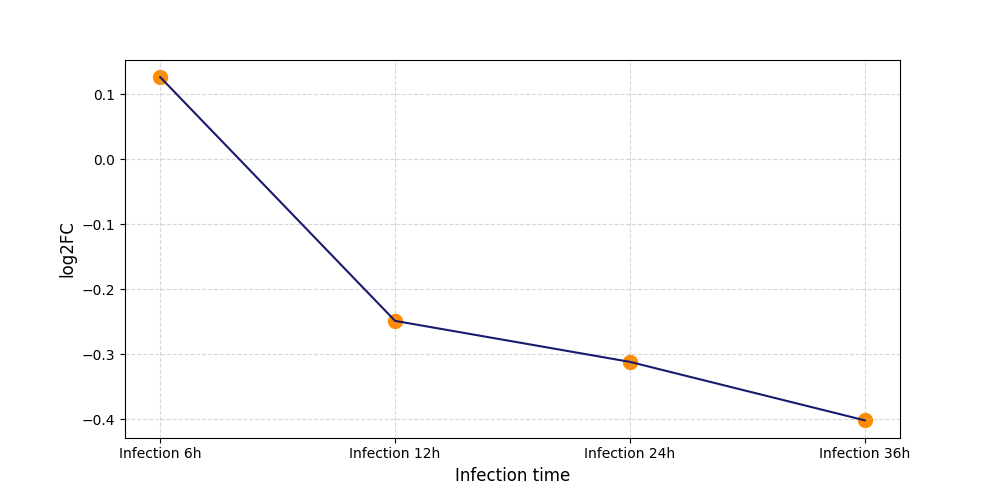 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1937
[6] |
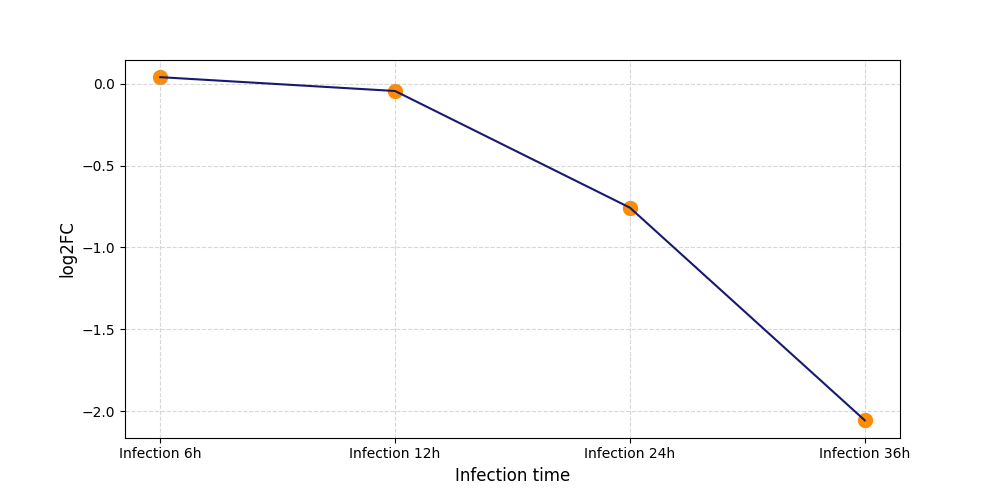 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1983
[6] |
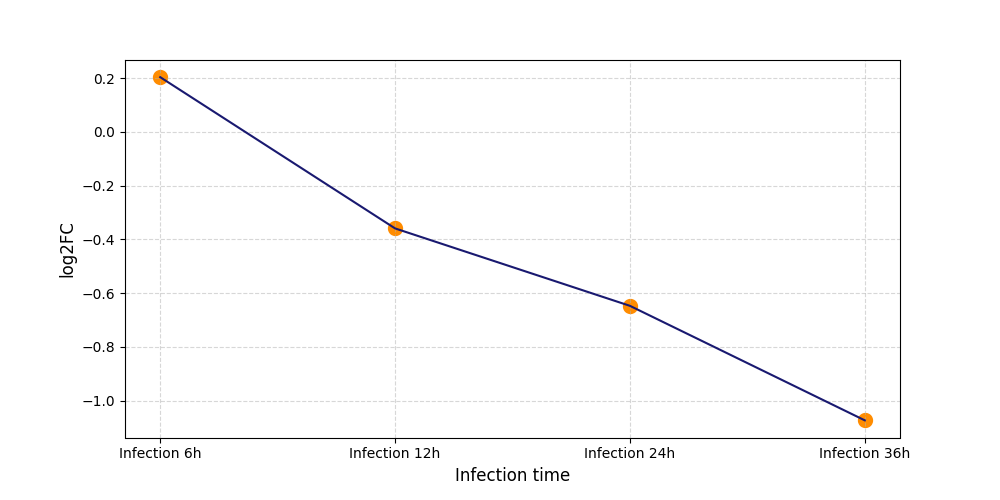 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2002
[6] |
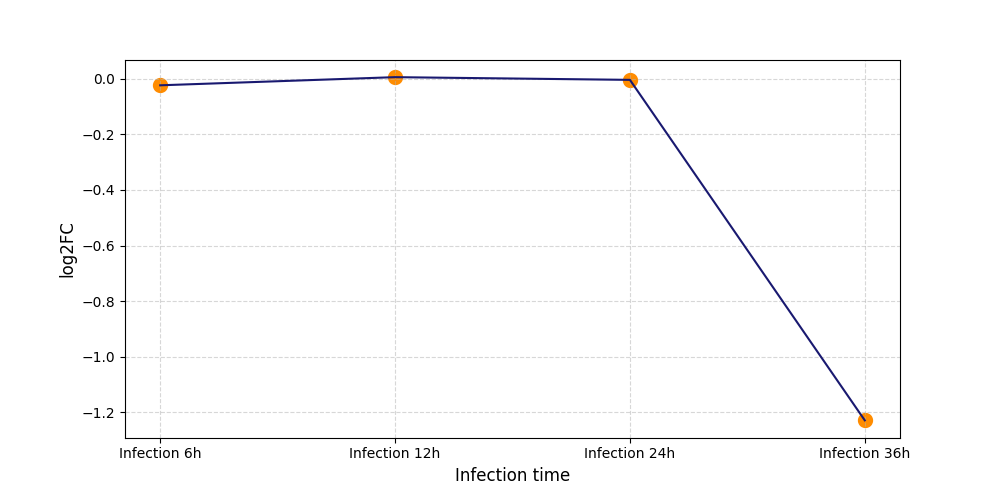 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2105
[5] |
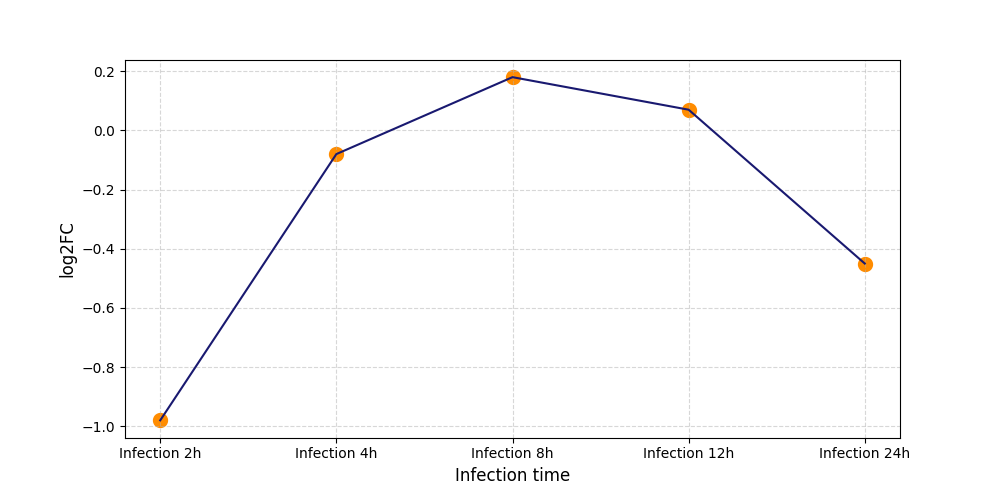 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2105
[6] |
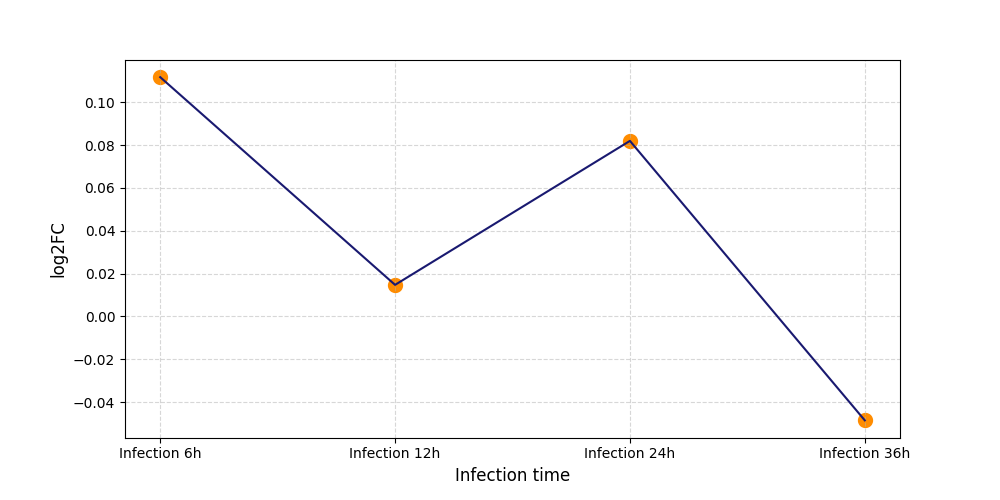 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2223
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2223
[6] |
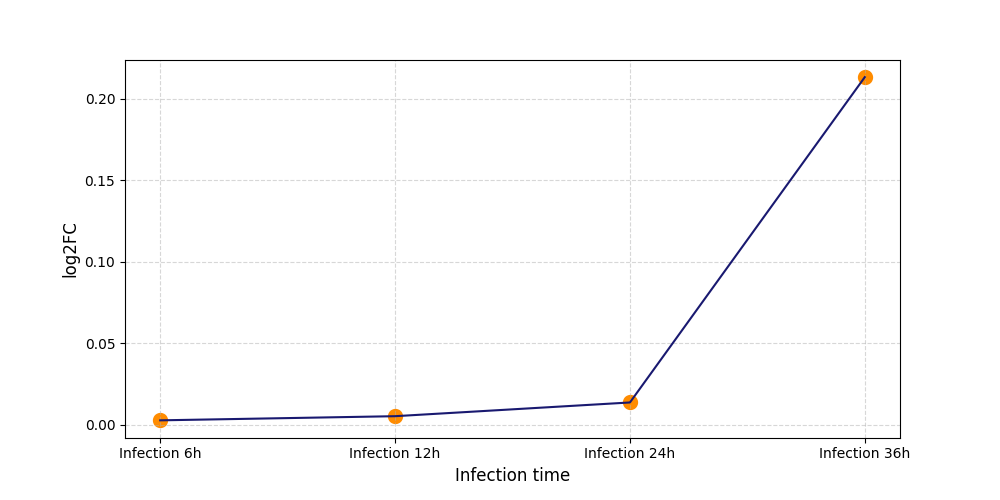 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2261
[6] |
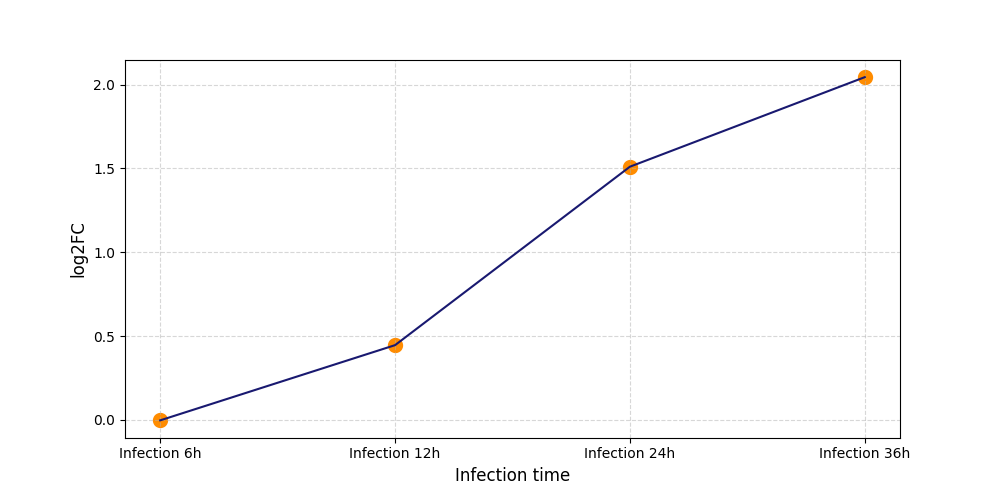 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2299
[6] |
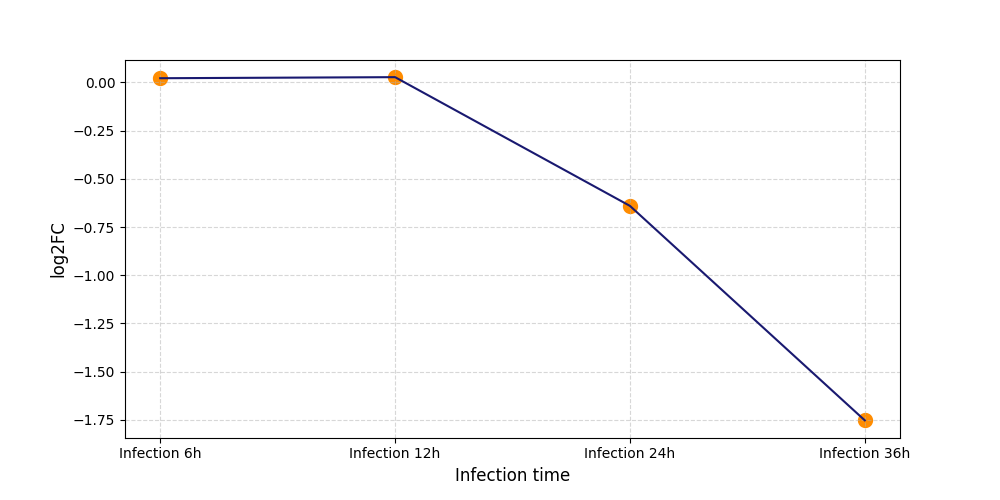 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2344
[6] |
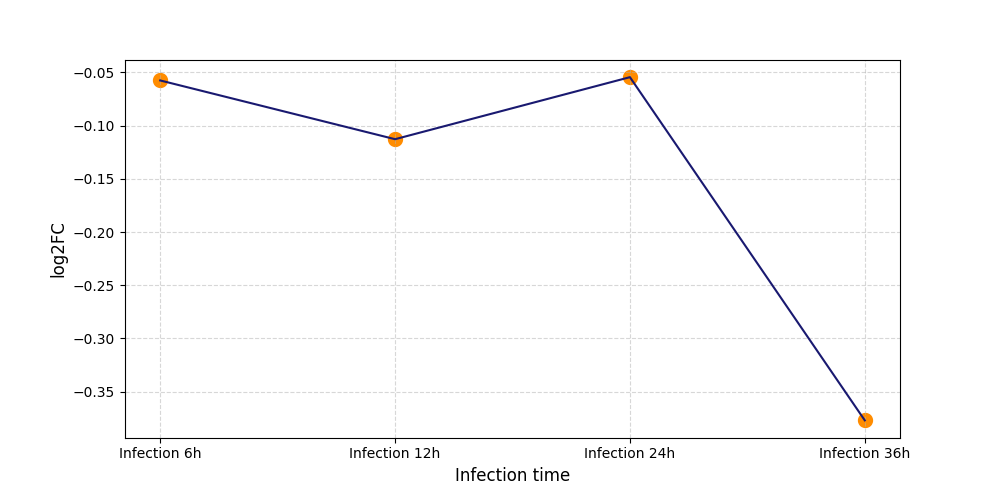 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2466
[6] |
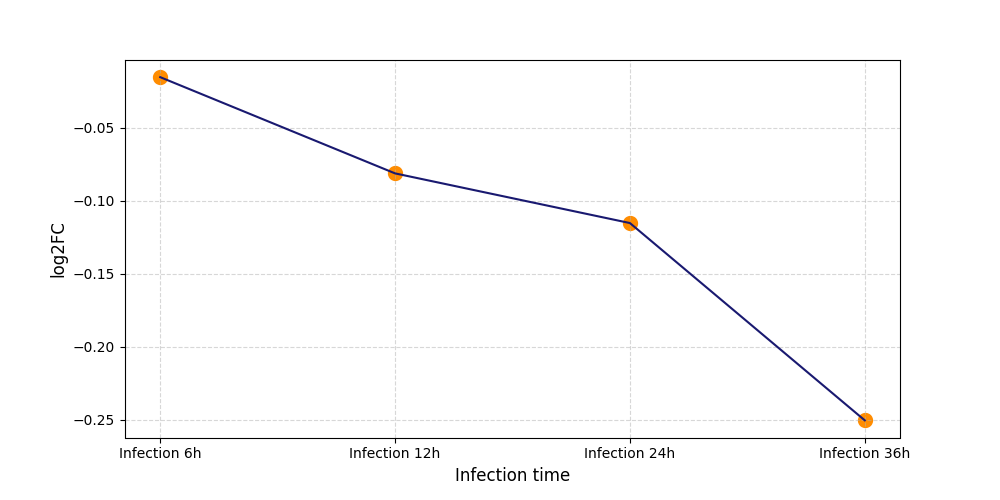 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2505
[6] |
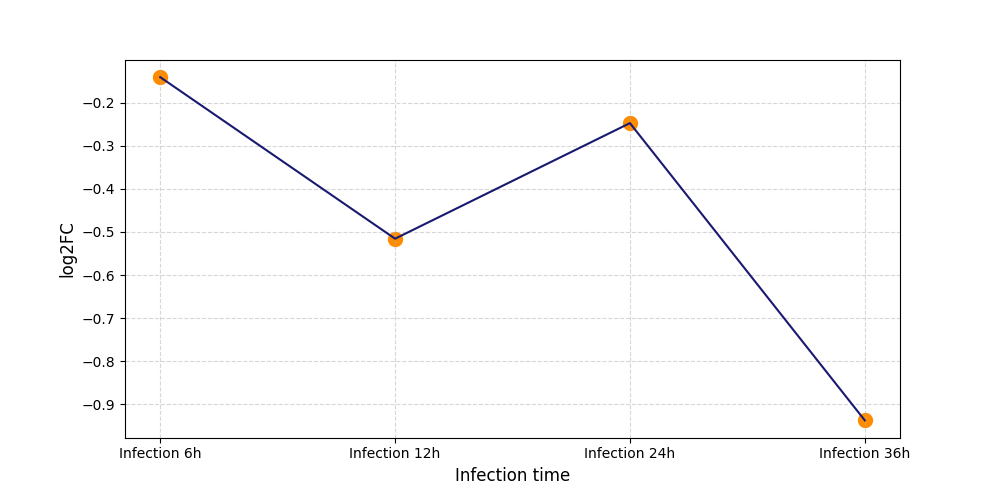 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2528
[5] |
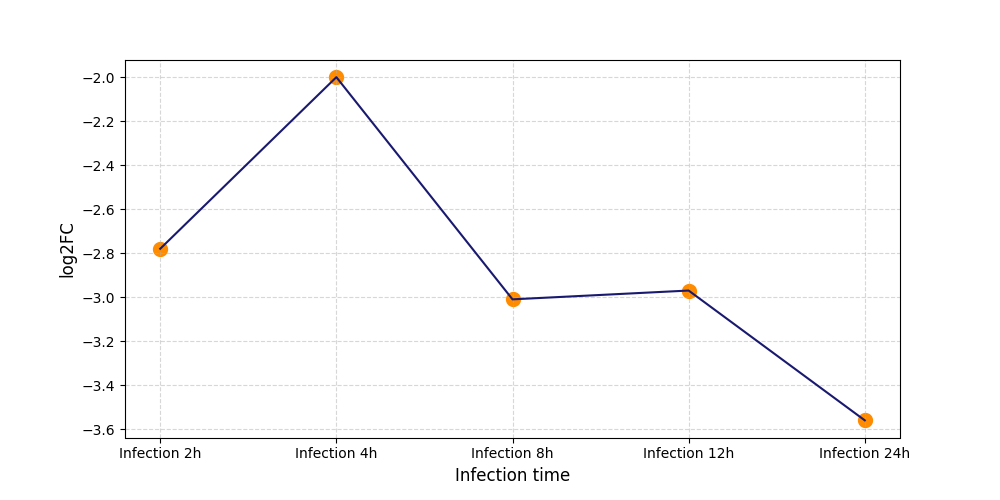 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2528
[6] |
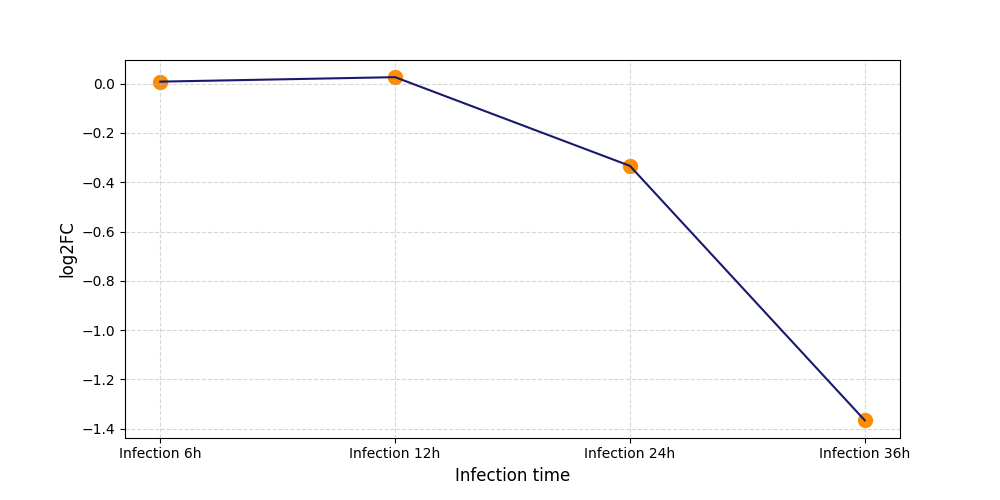 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2638
[6] |
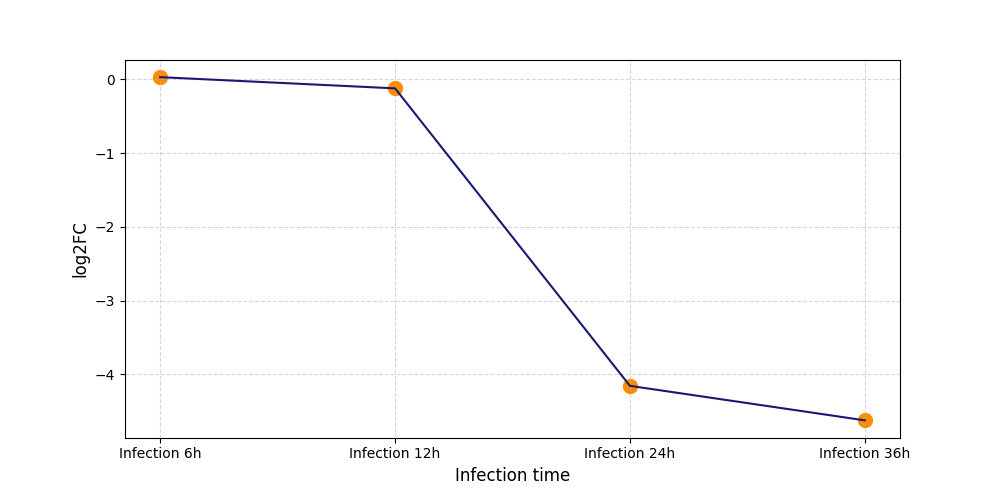 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2708
[5] |
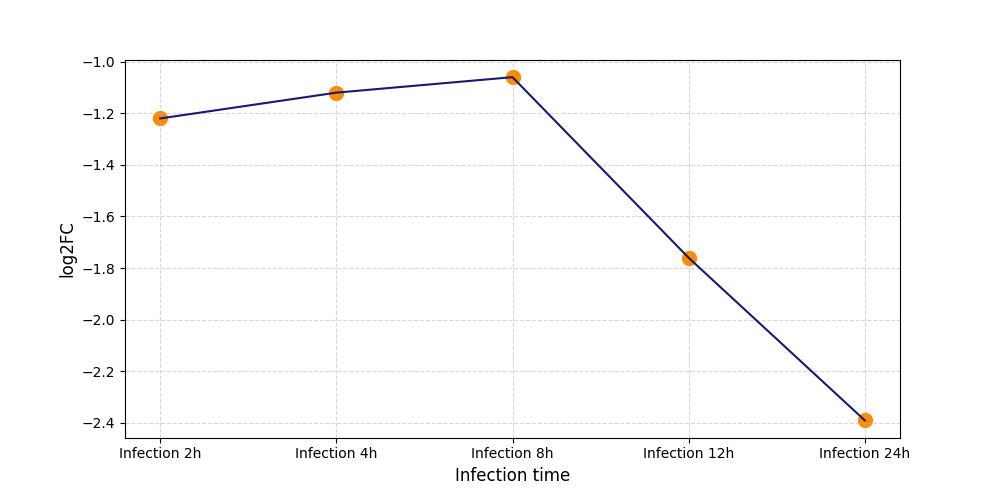 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2708
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2793
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2827
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2828
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S299
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S3041
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S3041
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S3042
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S308
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S308
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S330
[6] |
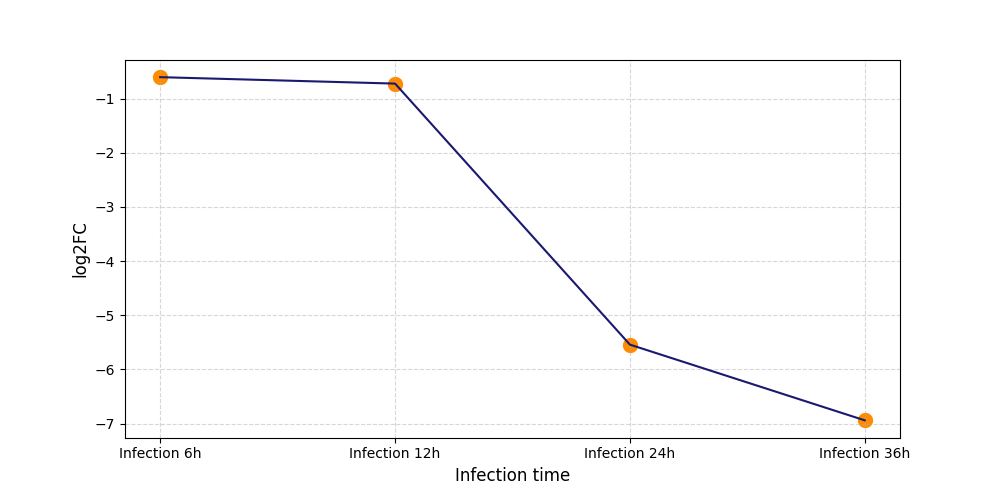 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S357
[6] |
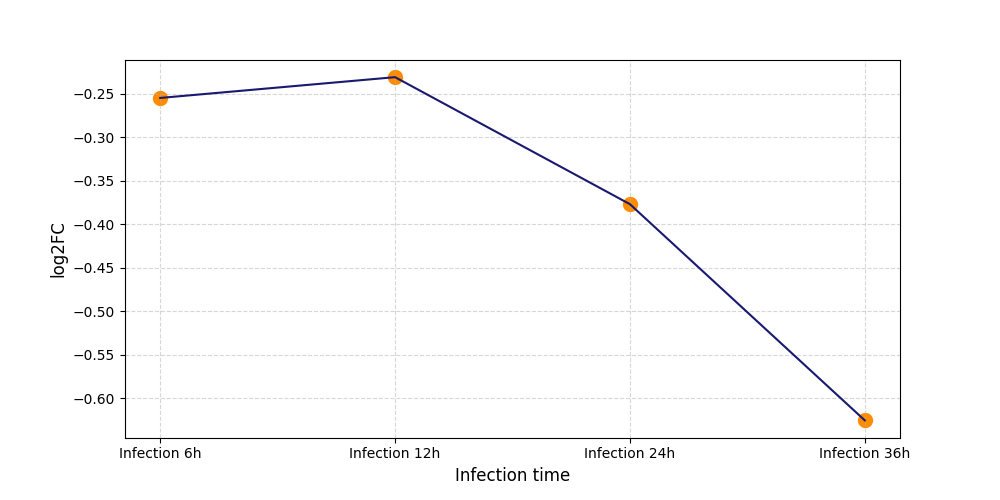 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S374
[6] |
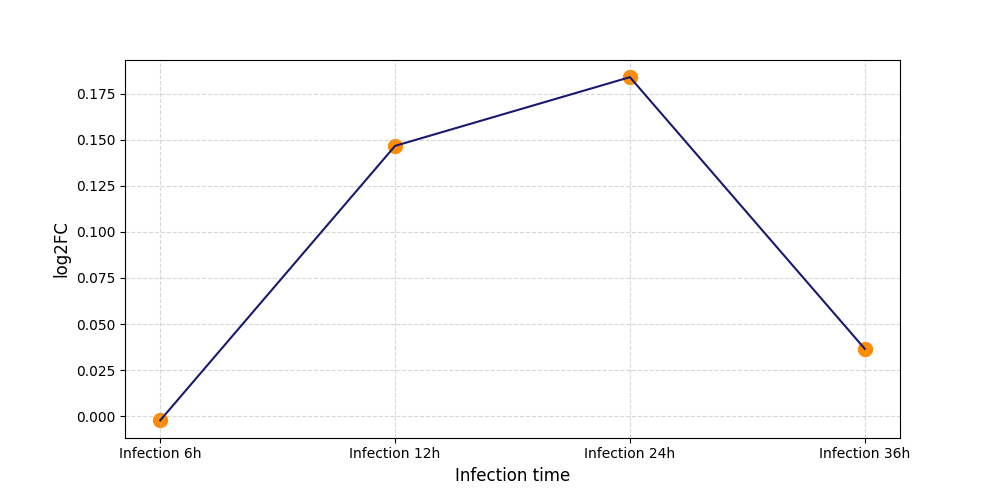 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S507
[6] |
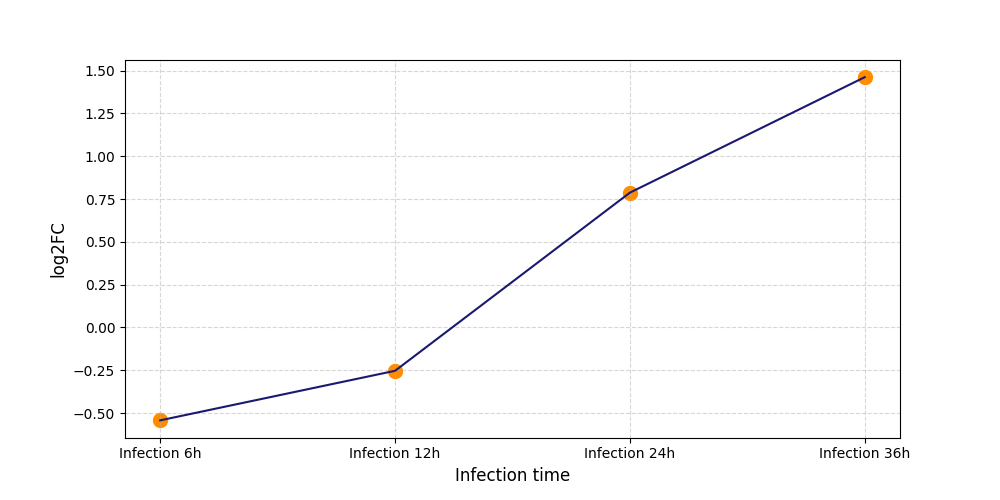 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
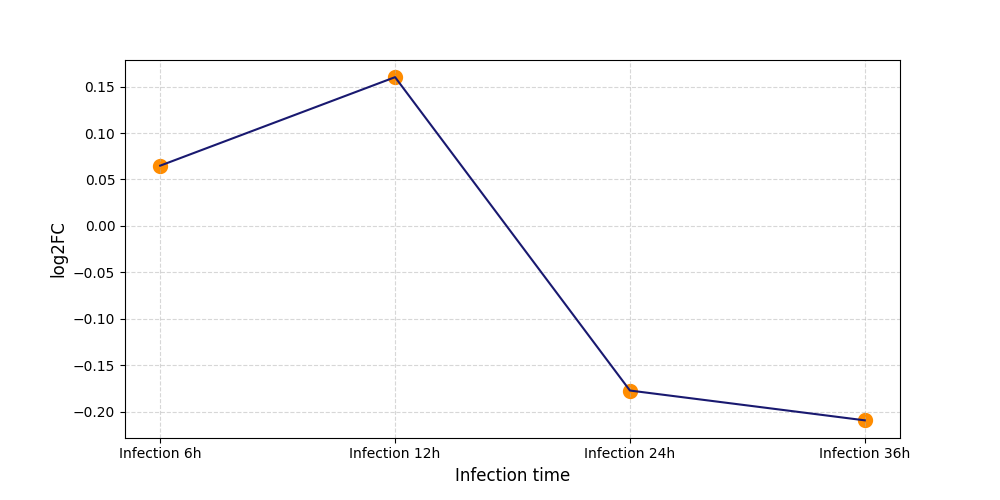
|
S538
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
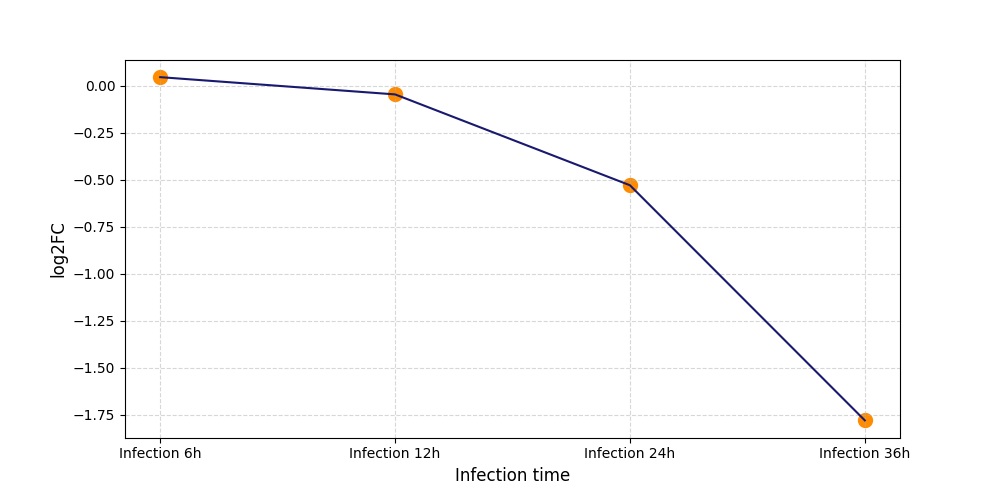
|
S584
[5] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S584
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S621
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S648
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
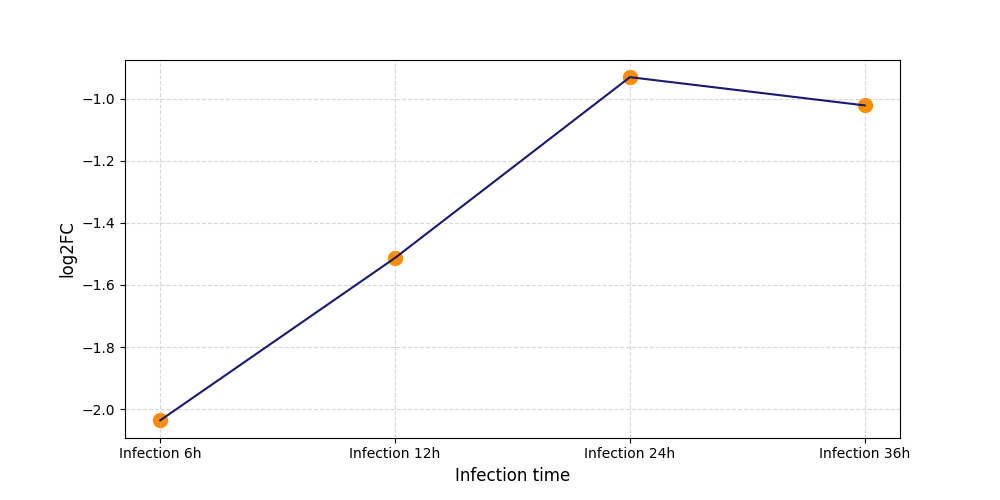
S704
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
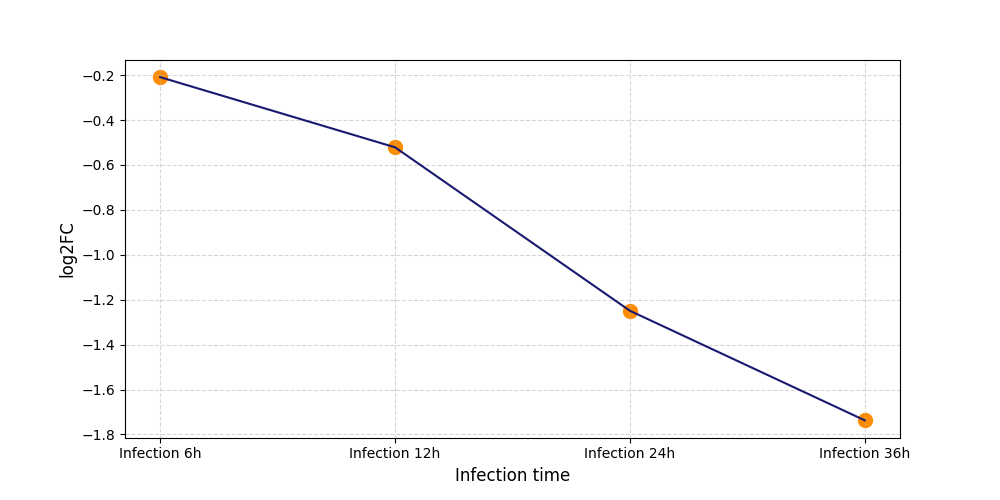
S713
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
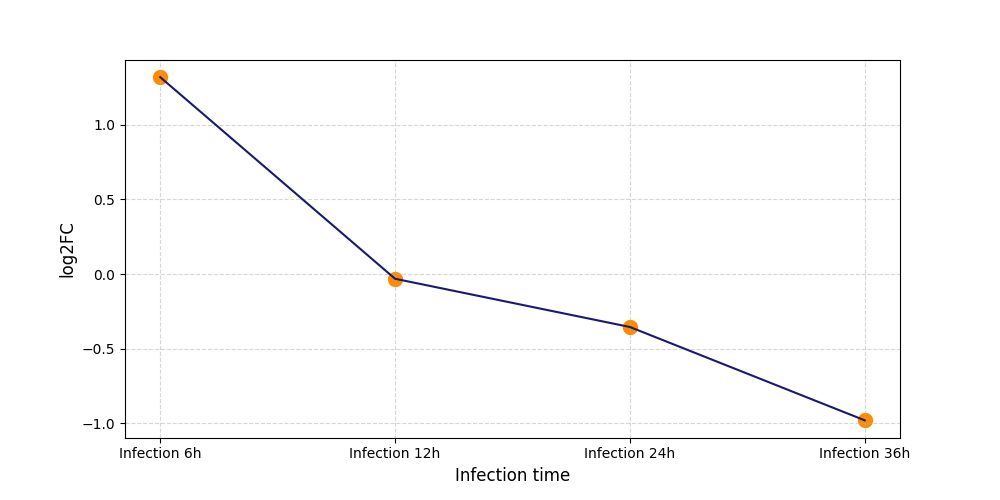
S827
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
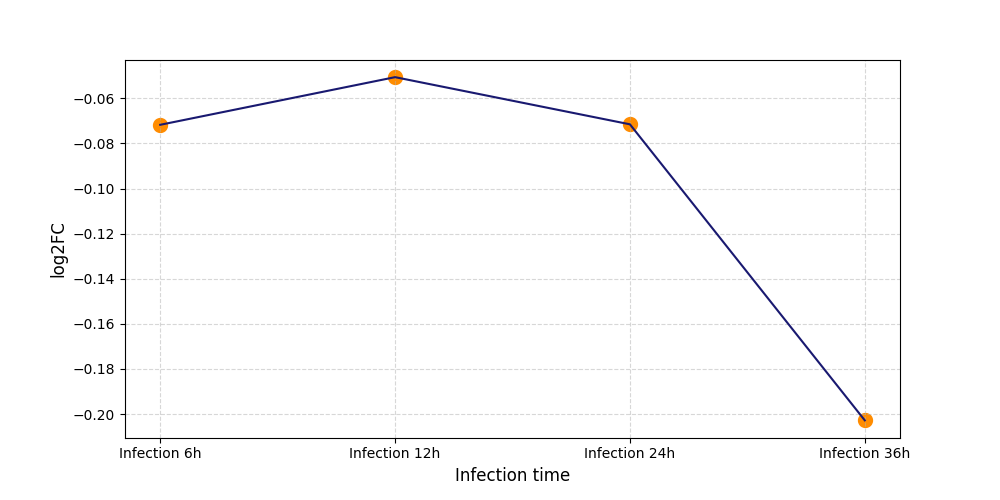
S859
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1017
[6] |
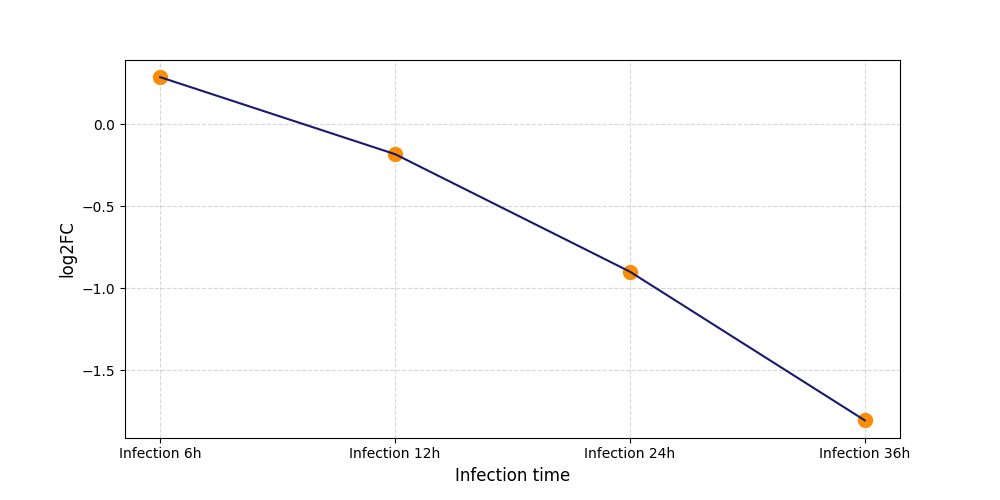 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1019
[6] |
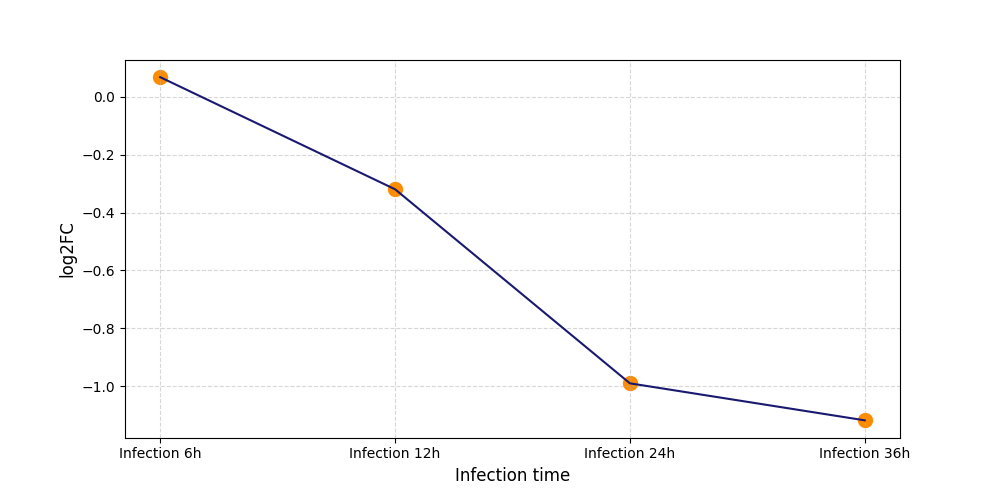 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1167
[6] |
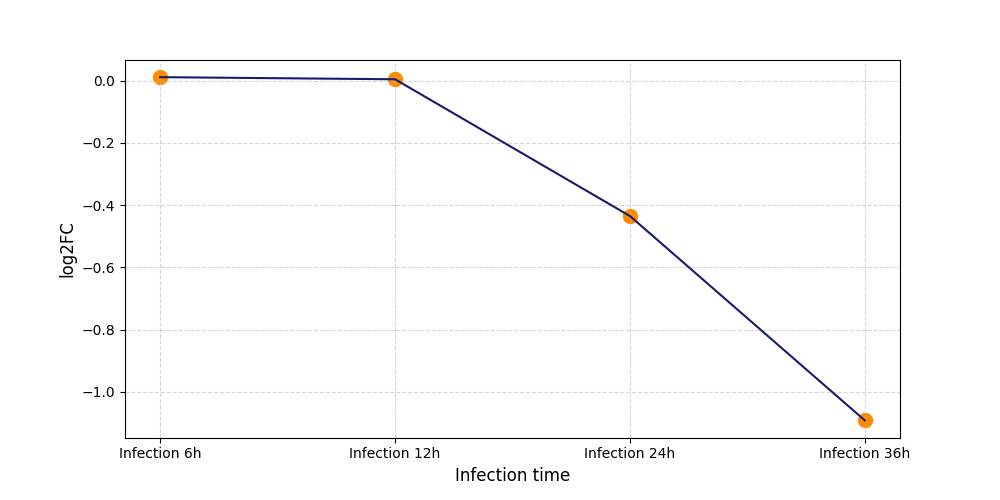 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1176
[6] |
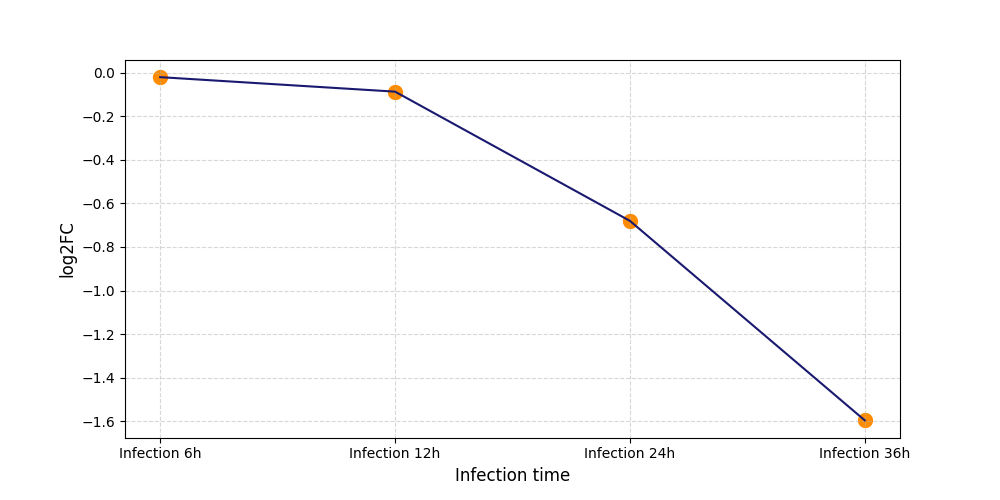 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1193
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1233
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1237
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1298
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1315
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1327
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1335
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1355
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1359
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1476
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1503
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1557
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
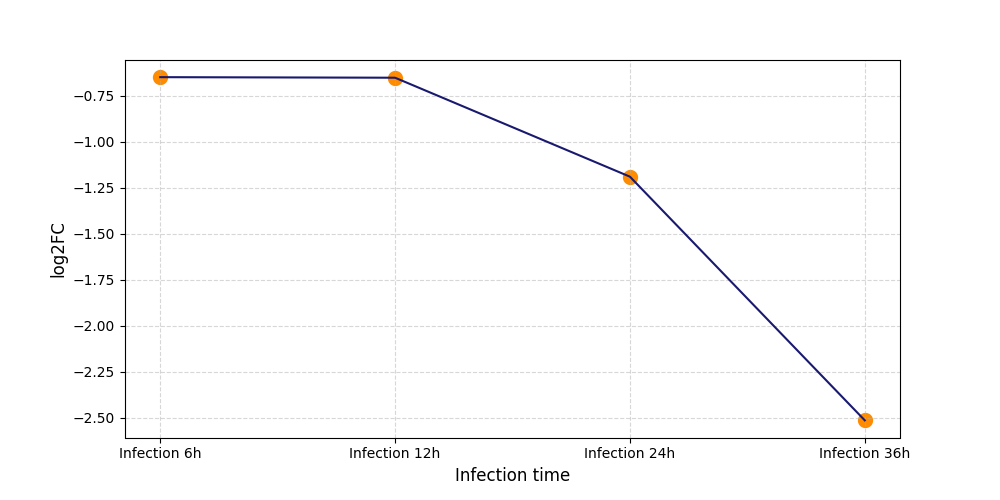
T1719
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
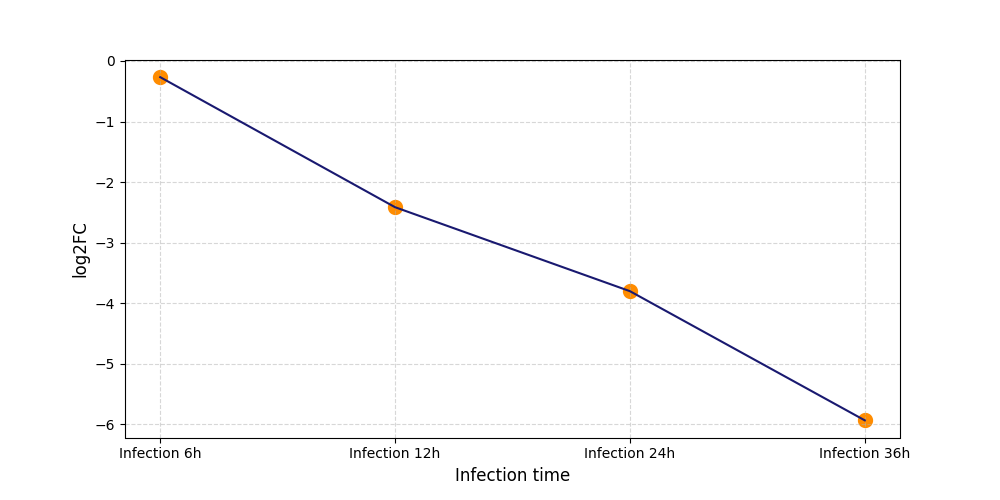
T1747
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
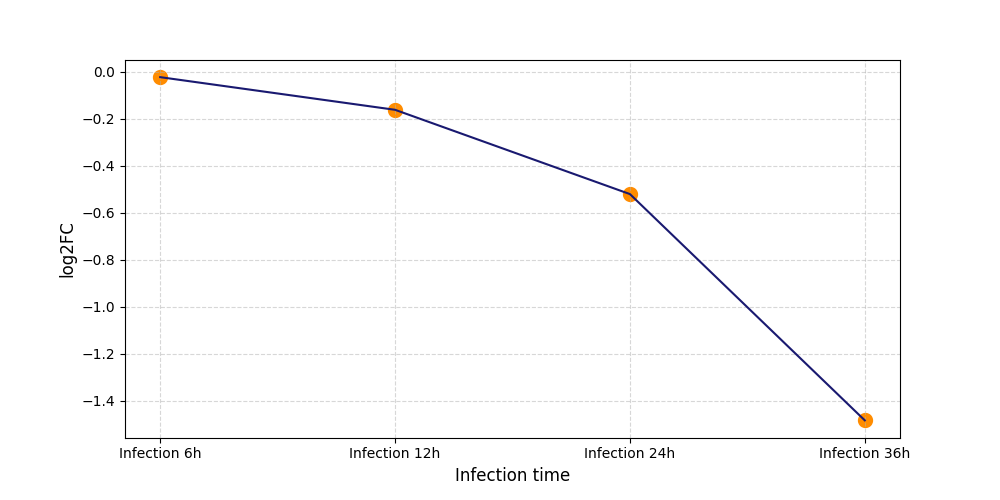
T1784
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
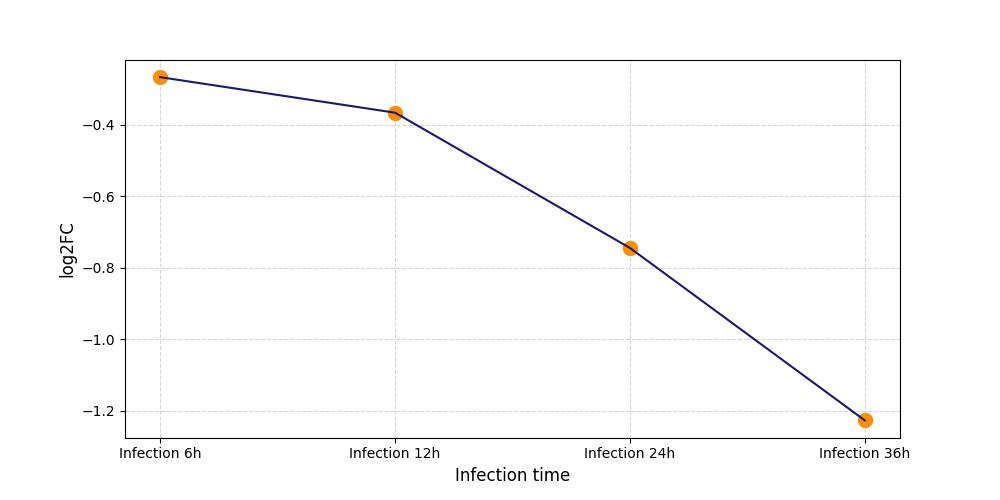
T1801
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1923
[6] |
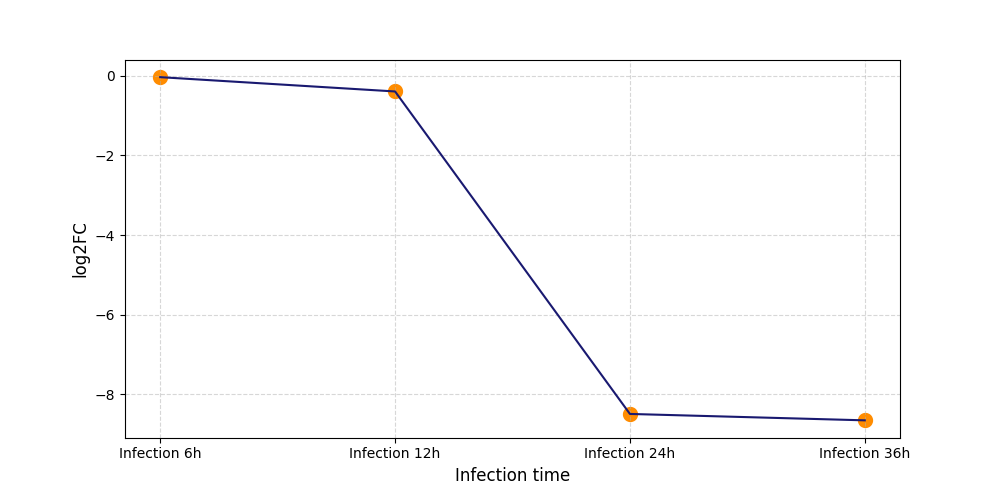 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1963
[6] |
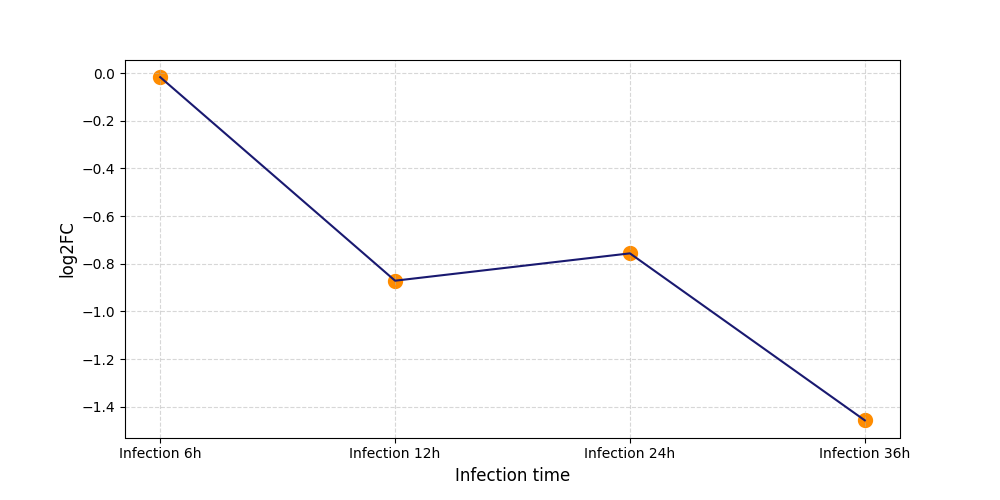 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2085
[6] |
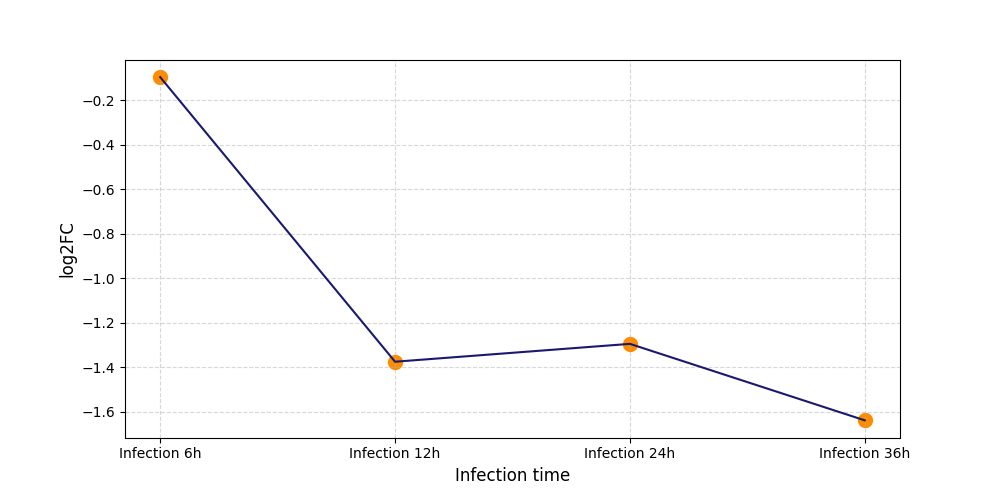 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2203
[6] |
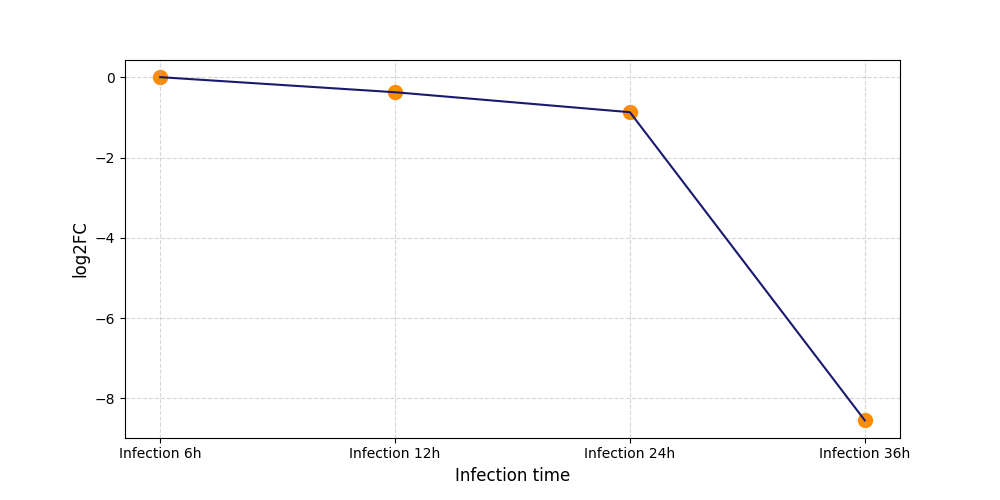 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2231
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2268
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2305
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2325
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2389
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2406
[6] |
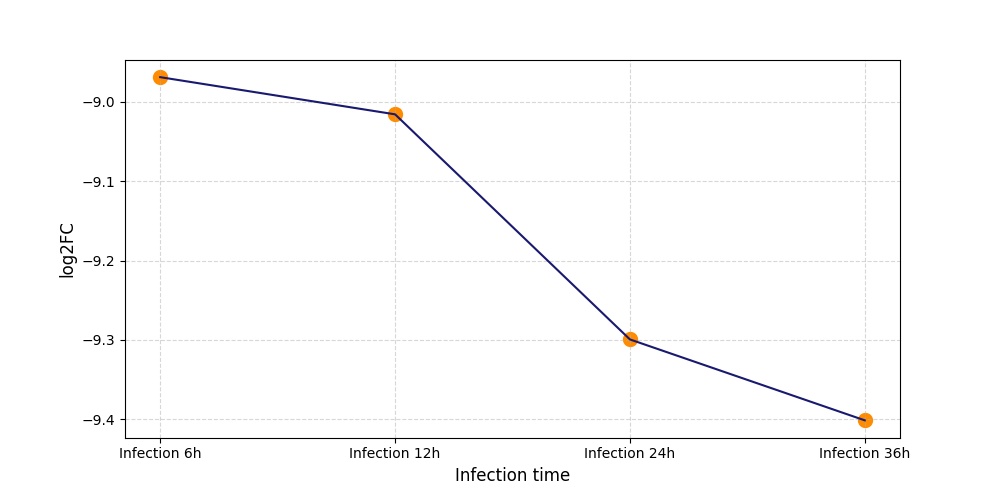 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T328
[6] |
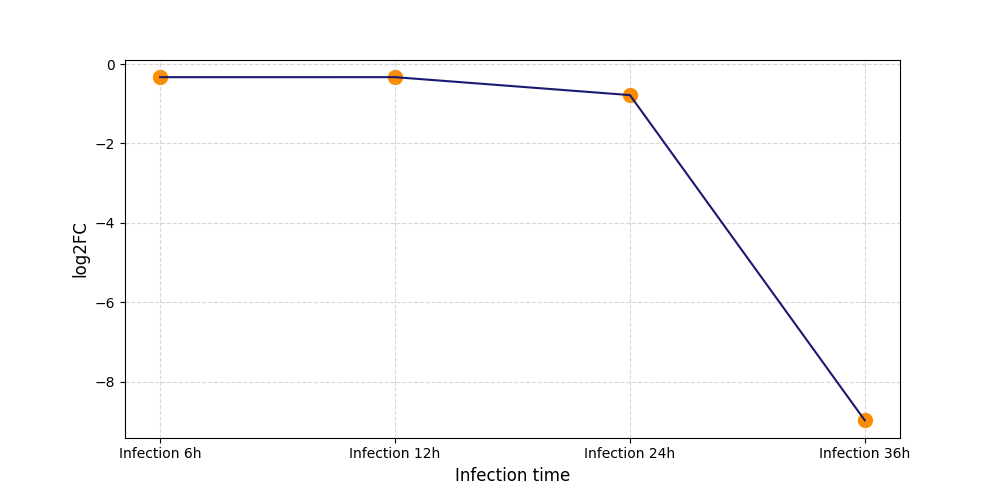 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T413
[6] |
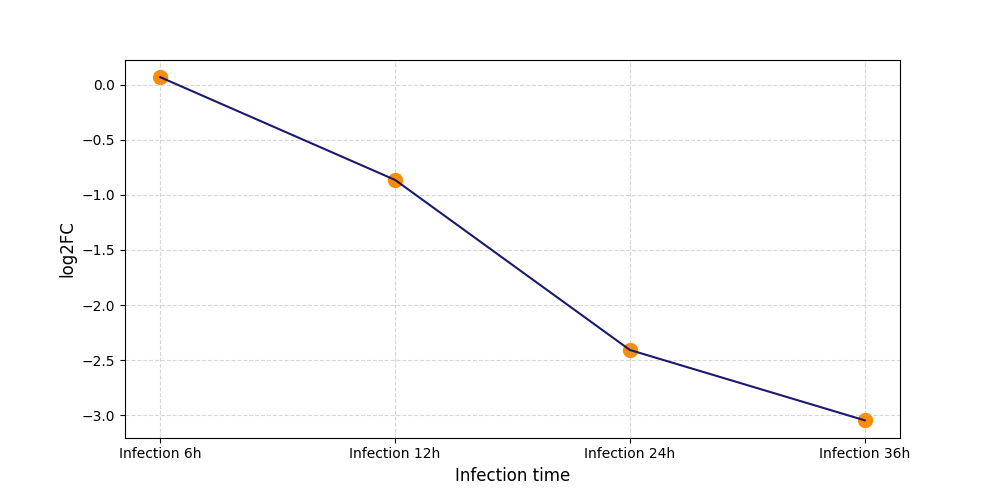 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T525
[6] |
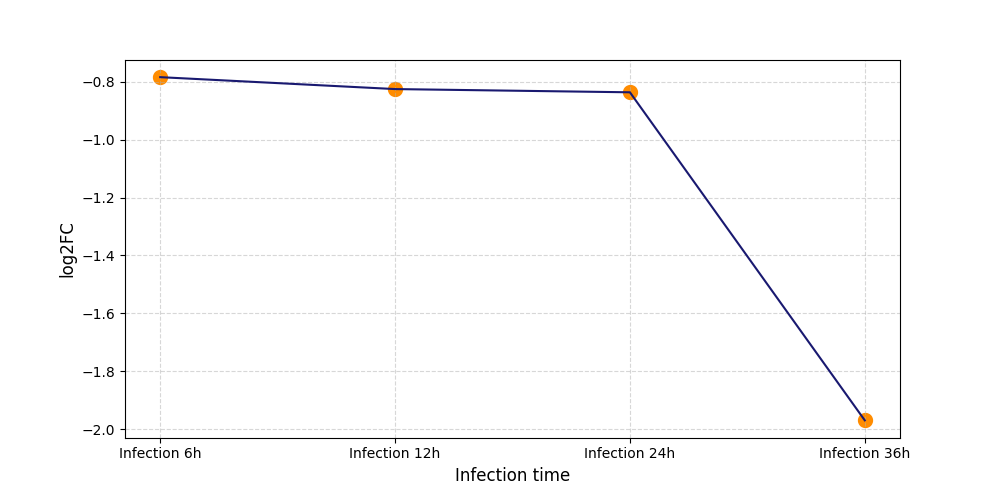 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T543
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T588
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T761
[6] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Sequence Information |
MWPTRRLVTIKRSGVDGPHFPLSLSTCLFGRGIECDIRIQLPVVSKQHCKIEIHEQEAILHNFSSTNPTQVNGSVIDEPVRLKHGDVITIIDRSFRYENESLQNGRKSTEFPRKIREQEPARRVSRSSFSSDPDEKAQDSKAYSKITEGKVSGNPQVHIKNVKEDSTADDSKDSVAQGTTNVHSSEHAGRNGRNAADPISGDFKEISSVKLVSRYGELKSVPTTQCLDNSKKNESPFWKLYESVKKELDVKSQKENVLQYCRKSGLQTDYATEKESADGLQGETQLLVSRKSRPKSGGSGHAVAEPASPEQELDQNKGKGRDVESVQTPSKAVGASFPLYEPAKMKTPVQYSQQQNSPQKHKNKDLYTTGRRESVNLGKSEGFKAGDKTLTPRKLSTRNRTPAKVEDAADSATKPENLSSKTRGSIPTDVEVLPTETEIHNEPFLTLWLTQVERKIQKDSLSKPEKLGTTAGQMCSGLPGLSSVDINNFGDSINESEGIPLKRRRVSFGGHLRPELFDENLPPNTPLKRGEAPTKRKSLVMHTPPVLKKIIKEQPQPSGKQESGSEIHVEVKAQSLVISPPAPSPRKTPVASDQRRRSCKTAPASSSKSQTEVPKRGGRKSGNLPSKRVSISRSQHDILQMICSKRRSGASEANLIVAKSWADVVKLGAKQTQTKVIKHGPQRSMNKRQRRPATPKKPVGEVHSQFSTGHANSPCTIIIGKAHTEKVHVPARPYRVLNNFISNQKMDFKEDLSGIAEMFKTPVKEQPQLTSTCHIAISNSENLLGKQFQGTDSGEEPLLPTSESFGGNVFFSAQNAAKQPSDKCSASPPLRRQCIRENGNVAKTPRNTYKMTSLETKTSDTETEPSKTVSTANRSGRSTEFRNIQKLPVESKSEETNTEIVECILKRGQKATLLQQRREGEMKEIERPFETYKENIELKENDEKMKAMKRSRTWGQKCAPMSDLTDLKSLPDTELMKDTARGQNLLQTQDHAKAPKSEKGKITKMPCQSLQPEPINTPTHTKQQLKASLGKVGVKEELLAVGKFTRTSGETTHTHREPAGDGKSIRTFKESPKQILDPAARVTGMKKWPRTPKEEAQSLEDLAGFKELFQTPGPSEESMTDEKTTKIACKSPPPESVDTPTSTKQWPKRSLRKADVEEEFLALRKLTPSAGKAMLTPKPAGGDEKDIKAFMGTPVQKLDLAGTLPGSKRQLQTPKEKAQALEDLAGFKELFQTPGHTEELVAAGKTTKIPCDSPQSDPVDTPTSTKQRPKRSIRKADVEGELLACRNLMPSAGKAMHTPKPSVGEEKDIIIFVGTPVQKLDLTENLTGSKRRPQTPKEEAQALEDLTGFKELFQTPGHTEEAVAAGKTTKMPCESSPPESADTPTSTRRQPKTPLEKRDVQKELSALKKLTQTSGETTHTDKVPGGEDKSINAFRETAKQKLDPAASVTGSKRHPKTKEKAQPLEDLAGLKELFQTPVCTDKPTTHEKTTKIACRSQPDPVDTPTSSKPQSKRSLRKVDVEEEFFALRKRTPSAGKAMHTPKPAVSGEKNIYAFMGTPVQKLDLTENLTGSKRRLQTPKEKAQALEDLAGFKELFQTRGHTEESMTNDKTAKVACKSSQPDPDKNPASSKRRLKTSLGKVGVKEELLAVGKLTQTSGETTHTHTEPTGDGKSMKAFMESPKQILDSAASLTGSKRQLRTPKGKSEVPEDLAGFIELFQTPSHTKESMTNEKTTKVSYRASQPDLVDTPTSSKPQPKRSLRKADTEEEFLAFRKQTPSAGKAMHTPKPAVGEEKDINTFLGTPVQKLDQPGNLPGSNRRLQTRKEKAQALEELTGFRELFQTPCTDNPTTDEKTTKKILCKSPQSDPADTPTNTKQRPKRSLKKADVEEEFLAFRKLTPSAGKAMHTPKAAVGEEKDINTFVGTPVEKLDLLGNLPGSKRRPQTPKEKAKALEDLAGFKELFQTPGHTEESMTDDKITEVSCKSPQPDPVKTPTSSKQRLKISLGKVGVKEEVLPVGKLTQTSGKTTQTHRETAGDGKSIKAFKESAKQMLDPANYGTGMERWPRTPKEEAQSLEDLAGFKELFQTPDHTEESTTDDKTTKIACKSPPPESMDTPTSTRRRPKTPLGKRDIVEELSALKQLTQTTHTDKVPGDEDKGINVFRETAKQKLDPAASVTGSKRQPRTPKGKAQPLEDLAGLKELFQTPICTDKPTTHEKTTKIACRSPQPDPVGTPTIFKPQSKRSLRKADVEEESLALRKRTPSVGKAMDTPKPAGGDEKDMKAFMGTPVQKLDLPGNLPGSKRWPQTPKEKAQALEDLAGFKELFQTPGTDKPTTDEKTTKIACKSPQPDPVDTPASTKQRPKRNLRKADVEEEFLALRKRTPSAGKAMDTPKPAVSDEKNINTFVETPVQKLDLLGNLPGSKRQPQTPKEKAEALEDLVGFKELFQTPGHTEESMTDDKITEVSCKSPQPESFKTSRSSKQRLKIPLVKVDMKEEPLAVSKLTRTSGETTQTHTEPTGDSKSIKAFKESPKQILDPAASVTGSRRQLRTRKEKARALEDLVDFKELFSAPGHTEESMTIDKNTKIPCKSPPPELTDTATSTKRCPKTRPRKEVKEELSAVERLTQTSGQSTHTHKEPASGDEGIKVLKQRAKKKPNPVEEEPSRRRPRAPKEKAQPLEDLAGFTELSETSGHTQESLTAGKATKIPCESPPLEVVDTTASTKRHLRTRVQKVQVKEEPSAVKFTQTSGETTDADKEPAGEDKGIKALKESAKQTPAPAASVTGSRRRPRAPRESAQAIEDLAGFKDPAAGHTEESMTDDKTTKIPCKSSPELEDTATSSKRRPRTRAQKVEVKEELLAVGKLTQTSGETTHTDKEPVGEGKGTKAFKQPAKRKLDAEDVIGSRRQPRAPKEKAQPLEDLASFQELSQTPGHTEELANGAADSFTSAPKQTPDSGKPLKISRRVLRAPKVEPVGDVVSTRDPVKSQSKSNTSLPPLPFKRGGGKDGSVTGTKRLRCMPAPEEIVEELPASKKQRVAPRARGKSSEPVVIMKRSLRTSAKRIEPAEELNSNDMKTNKEEHKLQDSVPENKGISLRSRRQNKTEAEQQITEVFVLAERIEINRNEKKPMKTSPEMDIQNPDDGARKPIPRDKVTENKRCLRSARQNESSQPKVAEESGGQKSAKVLMQNQKGKGEAGNSDSMCLRSRKTKSQPAASTLESKSVQRVTRSVKRCAENPKKAEDNVCVKKIRTRSHRDSEDI
Click to Show/Hide
|
|---|