Details of Host Protein
| Host Protein General Information (ID: PT1022) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name |
Serine/arginine repetitive matrix protein 1 (SRRM1)
|
Gene Name |
SRRM1
|
||||||
| Host Species |
Homo sapiens
|
Uniprot Entry Name |
SRRM1_HUMAN
|
||||||
| Protein Families |
Splicing factor SR family
|
||||||||
| Subcellular Location |
Nucleus
|
||||||||
| External Link | |||||||||
| NCBI Gene ID | |||||||||
| Uniprot ID | |||||||||
| Ensembl ID | |||||||||
| HGNC ID | |||||||||
| Function in Host |
Part of pre- and post-splicing multiprotein mRNP complexes. Involved in numerous pre-mRNA processing events. Promotes constitutiveand exonic splicing enhancer (ESE) -dependent splicing activation bybridging together sequence-specific (SR family proteins, SFRS4, SFRS5and TRA2B/SFRS10) and basal snRNP (SNRP70 and SNRPA1) factors of thespliceosome. Stimulates mRNA 3'-end cleavage independently of theformation of an exon junction complex. Binds both pre-mRNA and splicedmRNA 20-25 nt upstream of exon-exon junctions. Binds RNA and DNA withlow sequence specificity and has similar preference for eitherdouble- or single-stranded nucleic acid substrates.
[1-6]
Click to Show/Hide
|
||||||||
| Related KEGG Pathway | |||||||||
| Nucleocytoplasmic transport | hsa03013 |
Pathway Map 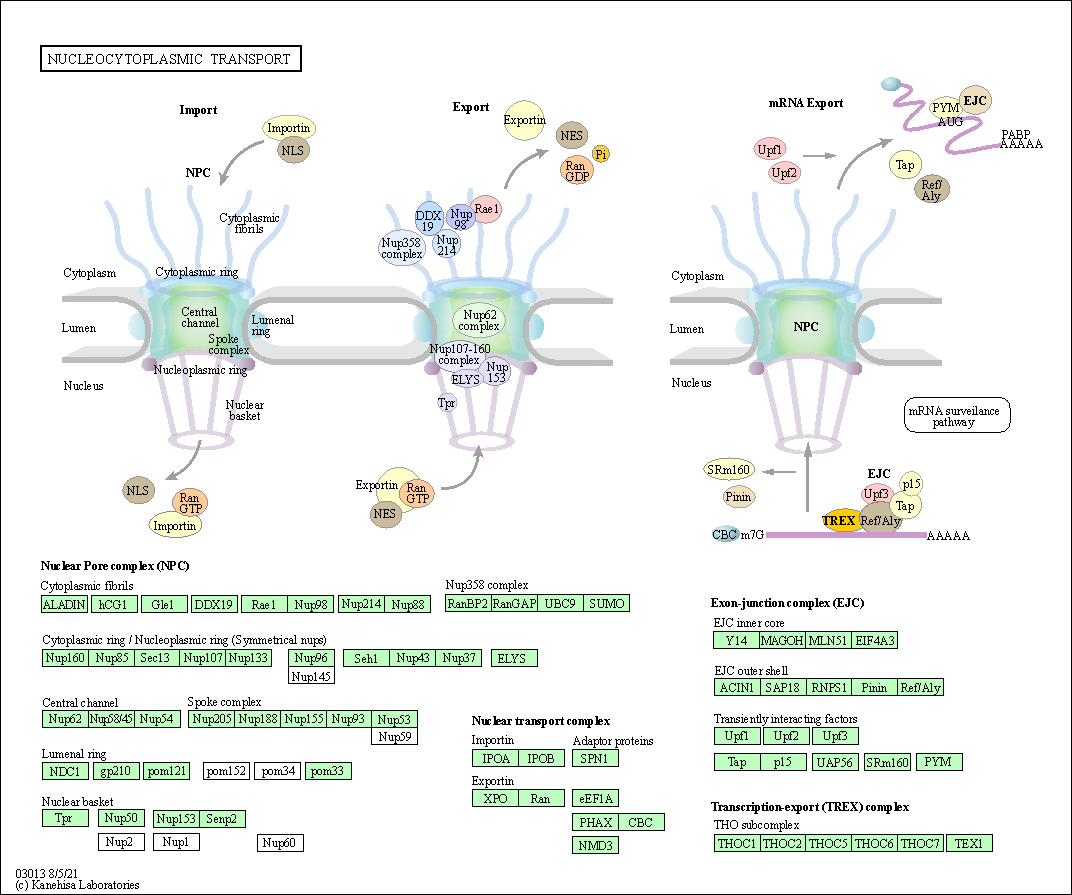
|
|||||||
| mRNA surveillance pathway | hsa03015 |
Pathway Map 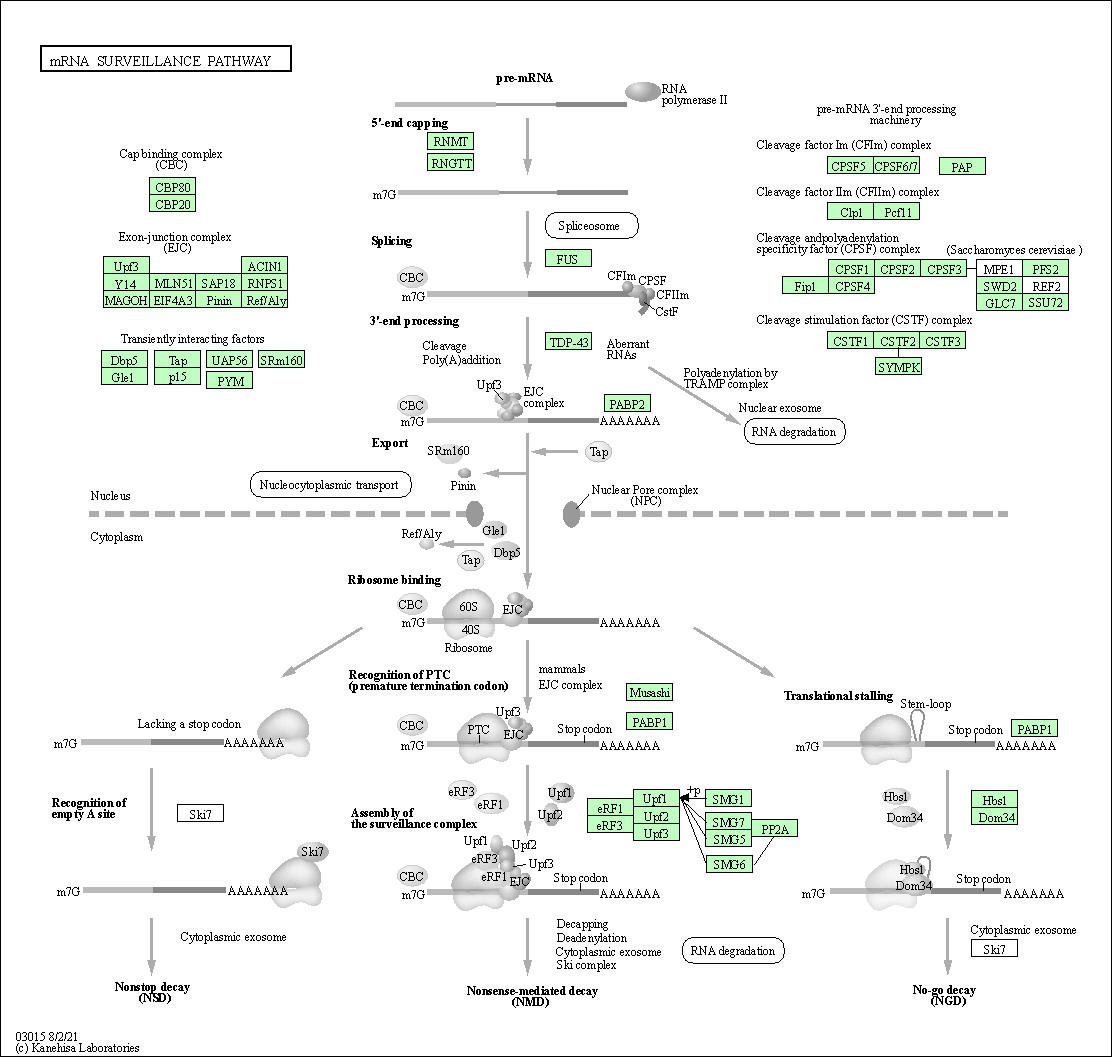
|
|||||||
| 3D Structure |
|
||||||||
| Full List of Virus RNA Interacting with This Protien | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| RNA Region: Not Specified Virus Region (hCoV-19/England/02/2020 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[7] | |||||||
| Strains Name |
hCoV-19/England/02/2020
|
||||||||
| Strains Family |
Beta (B.1.351)
|
||||||||
| RNA Binding Region |
Not Specified Virus Region
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Infection Cells | Calu-3 cells (Human lung cancer cell) (CVCL_0609 ) | ||||||||
| Cell Originated Tissue | Lung | ||||||||
| Infection Time | 24 h | ||||||||
| Interaction Score | P-adjust = 0.071 | ||||||||
| Method Description | UV protein-RNA crosslinking; RNA interactome capture (cRIC); RNA antisense purification coupled with mass spectrometry (RAP-MS) | ||||||||
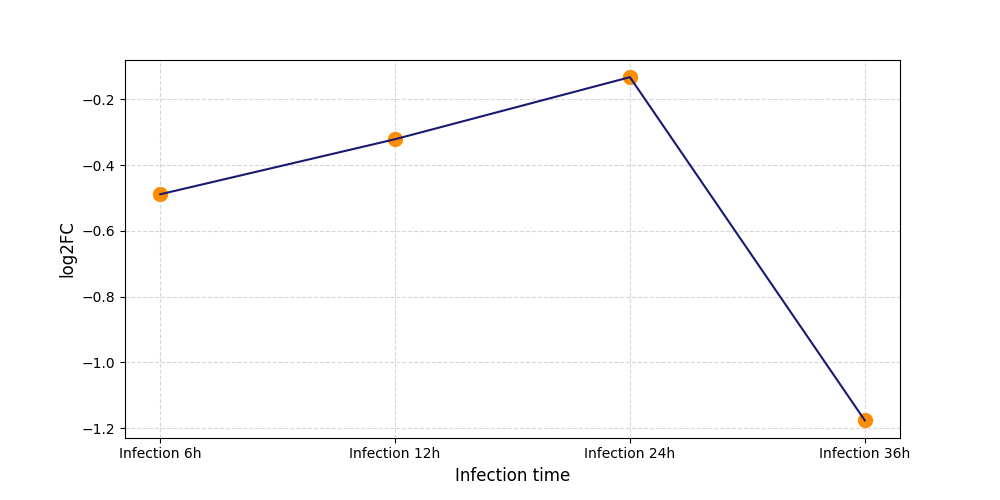
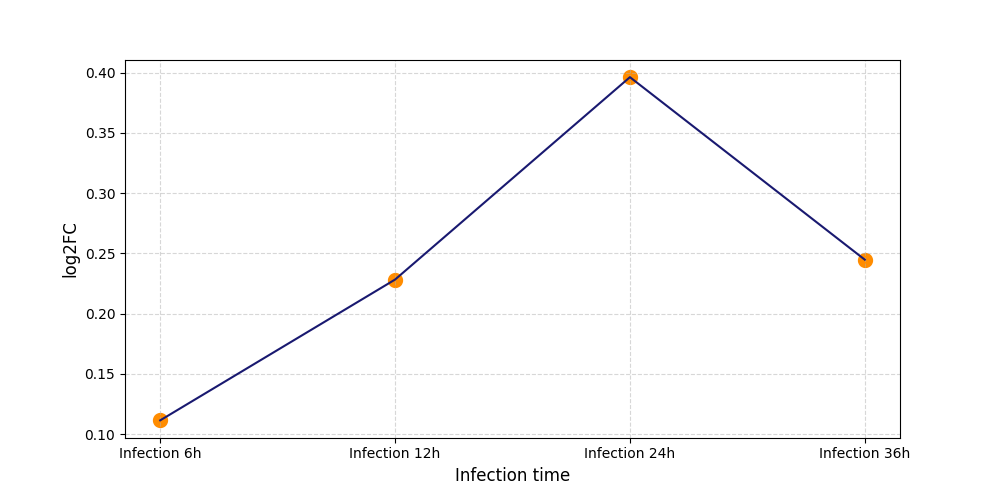
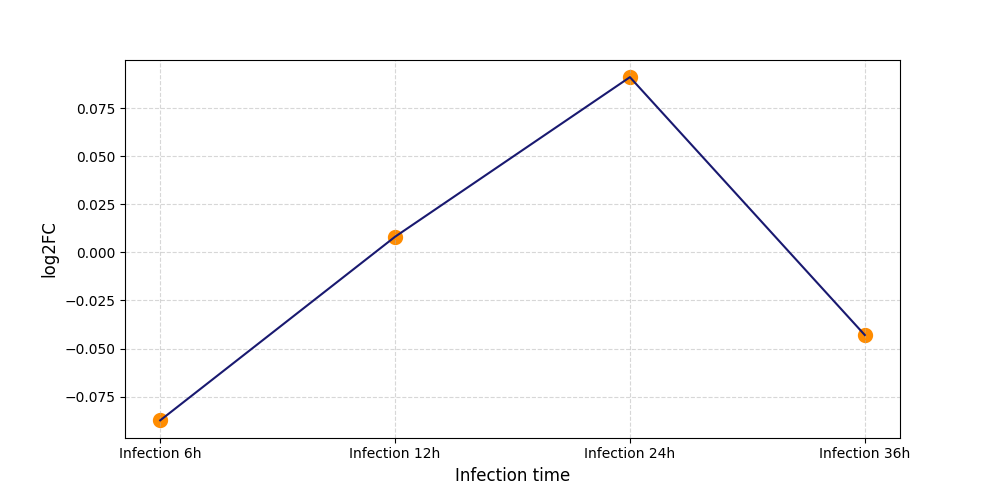
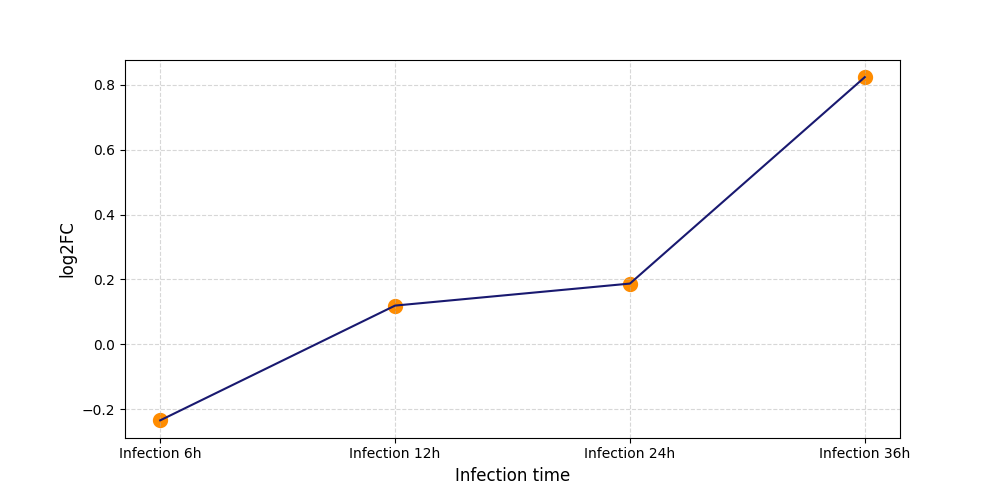
| Differential Gene Expression During SARS-COV-2 Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Phosphorylation after Virus Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
-543
[8] |
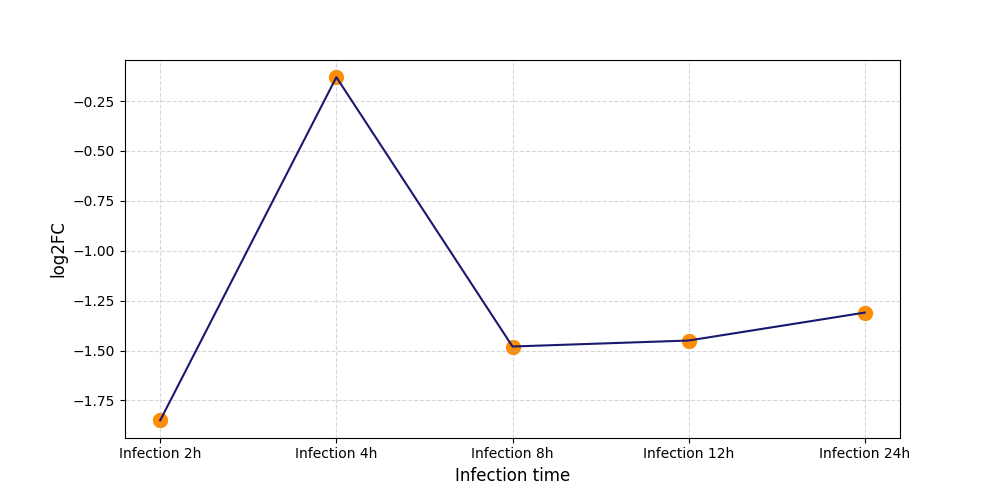 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S234
[9] |
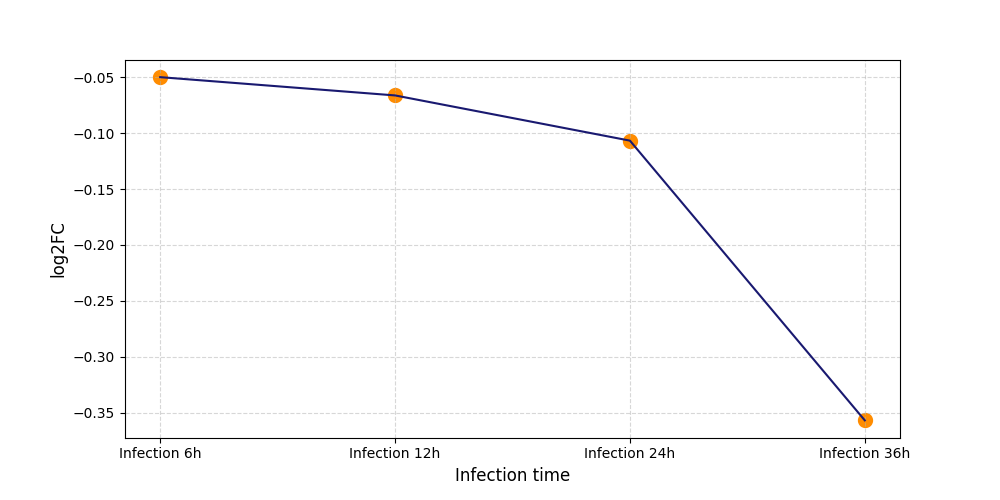 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S260
[9] |
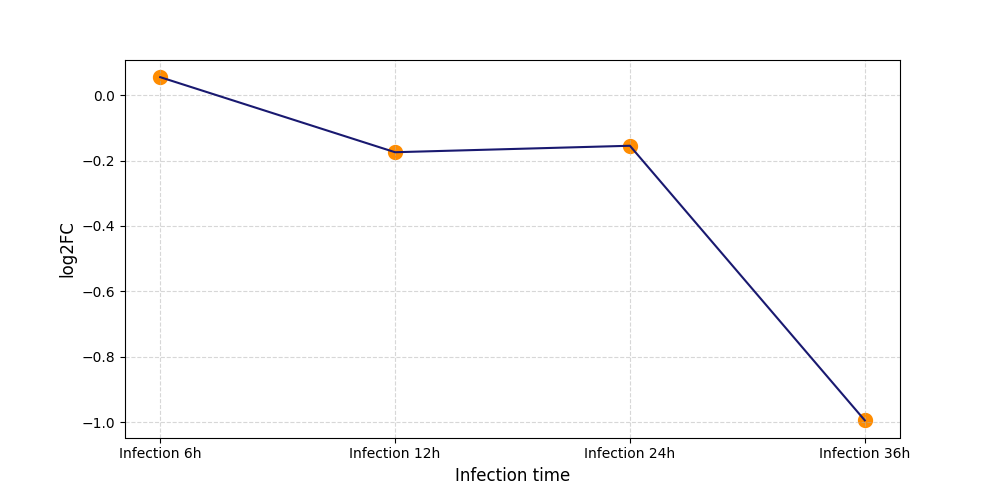 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
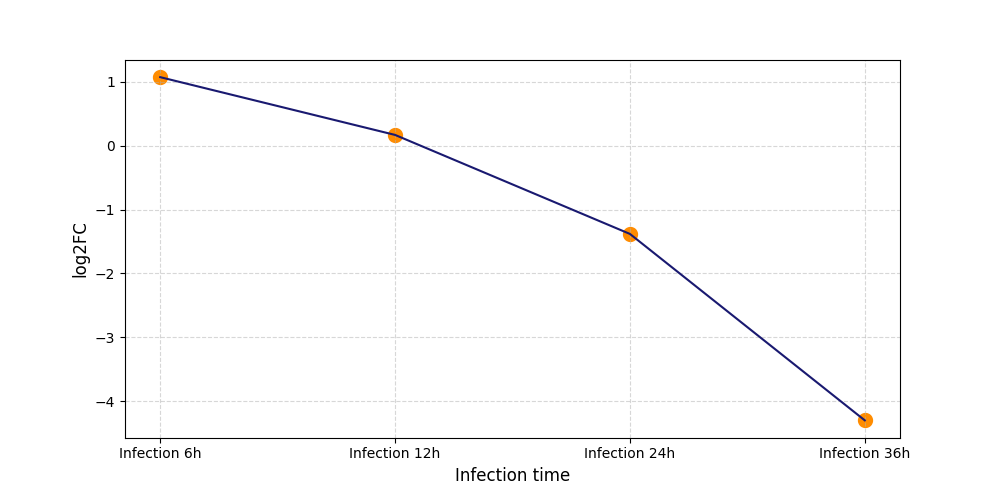
|
S380
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
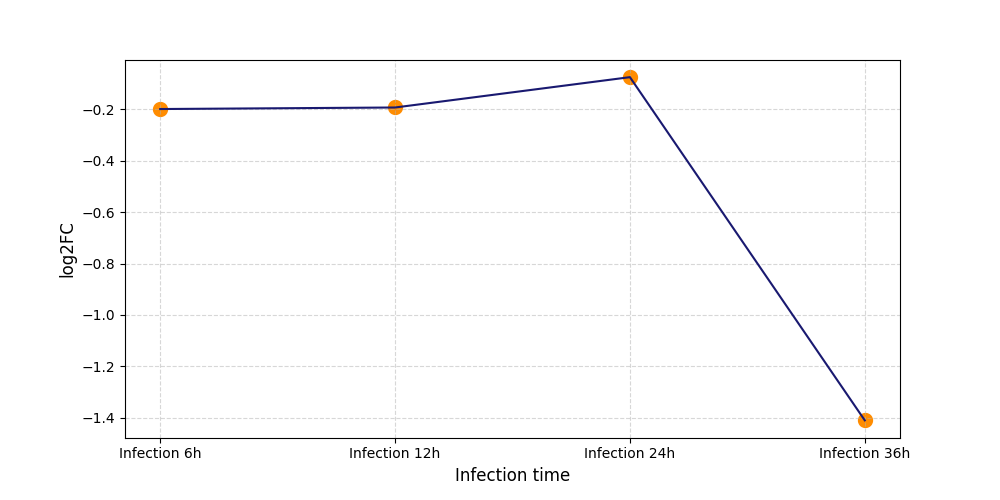
|
S389
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
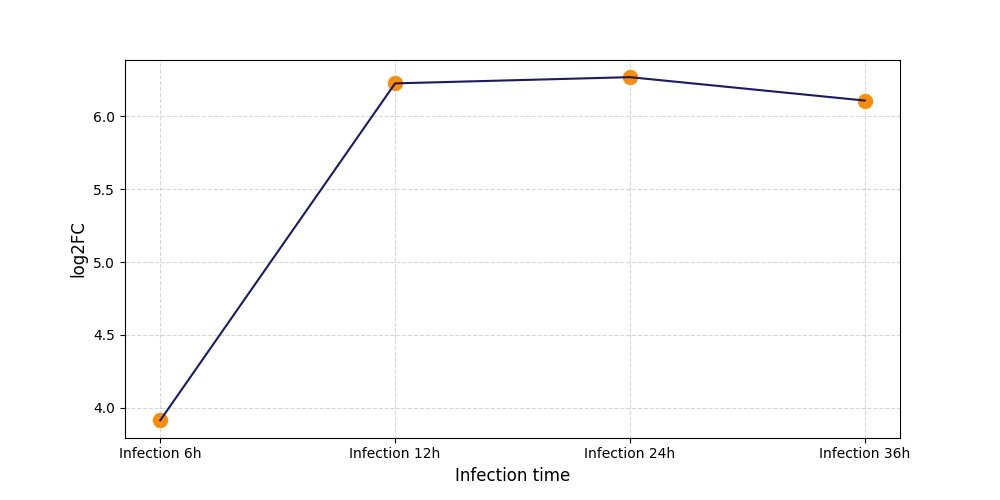
|
S391
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
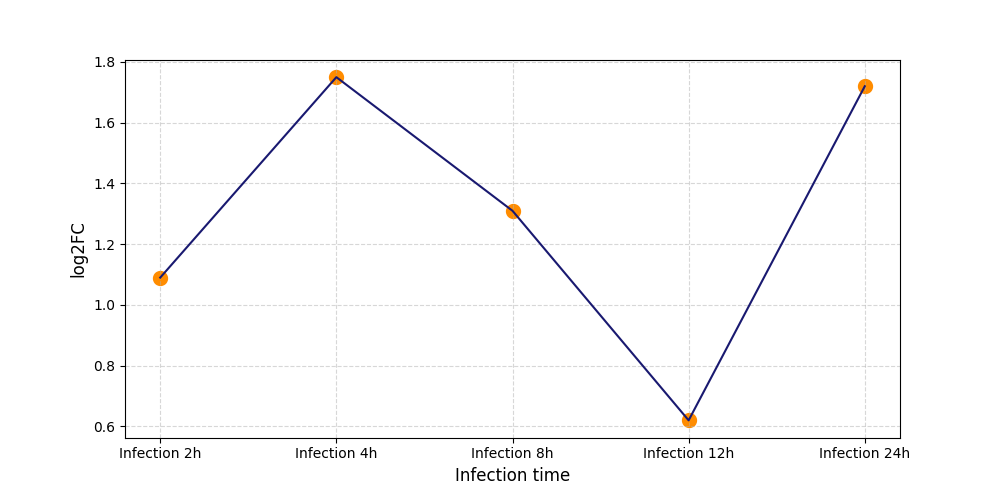
|
S393
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
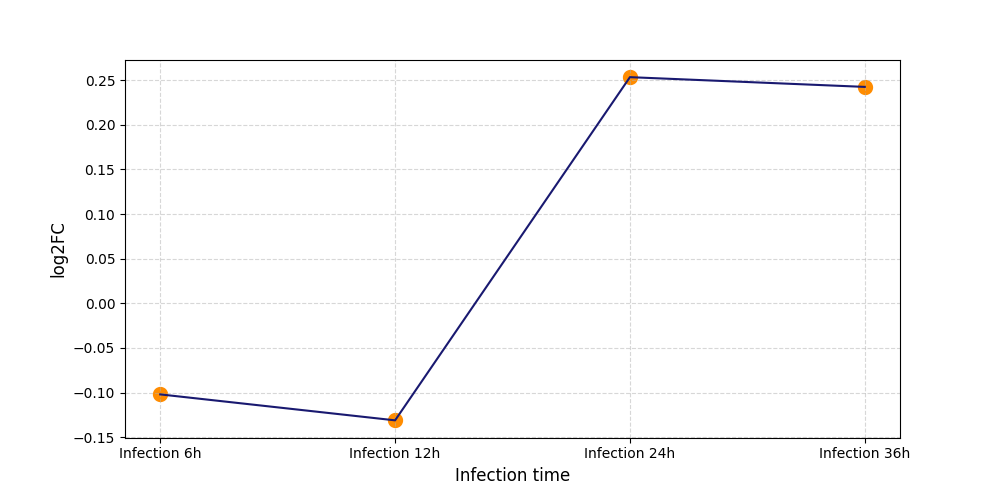
|
S393
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
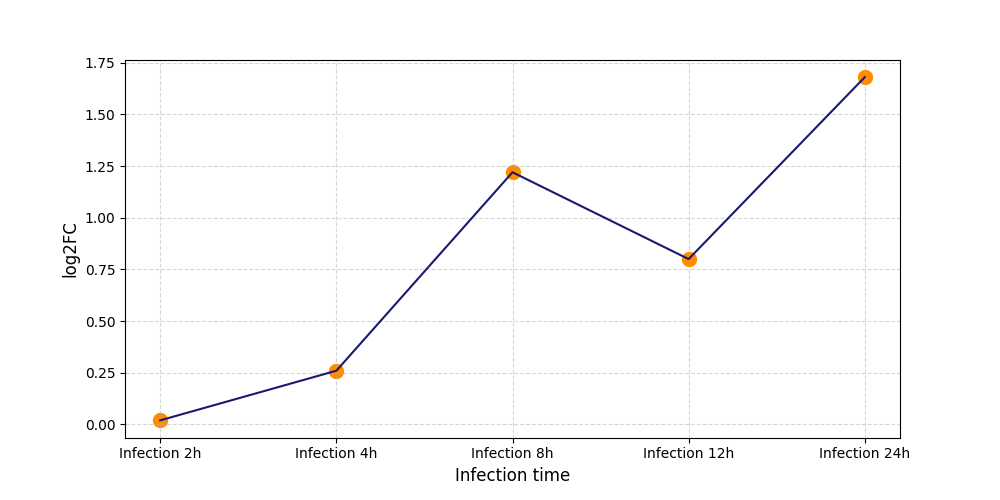
|
S402
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
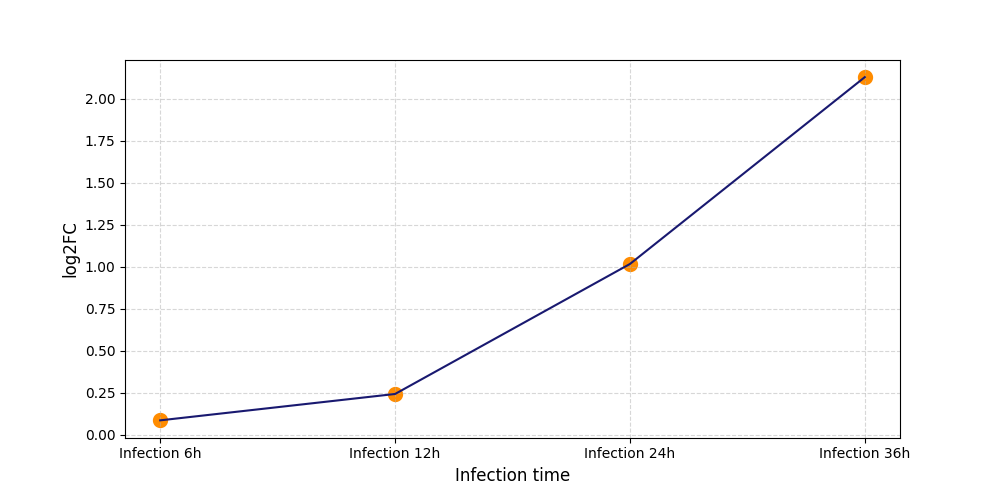
|
S402
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
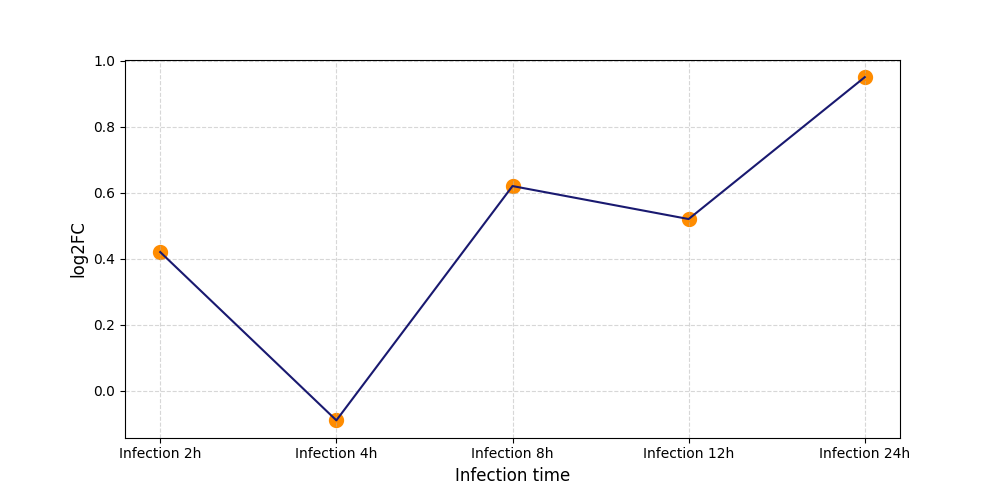
|
S414
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
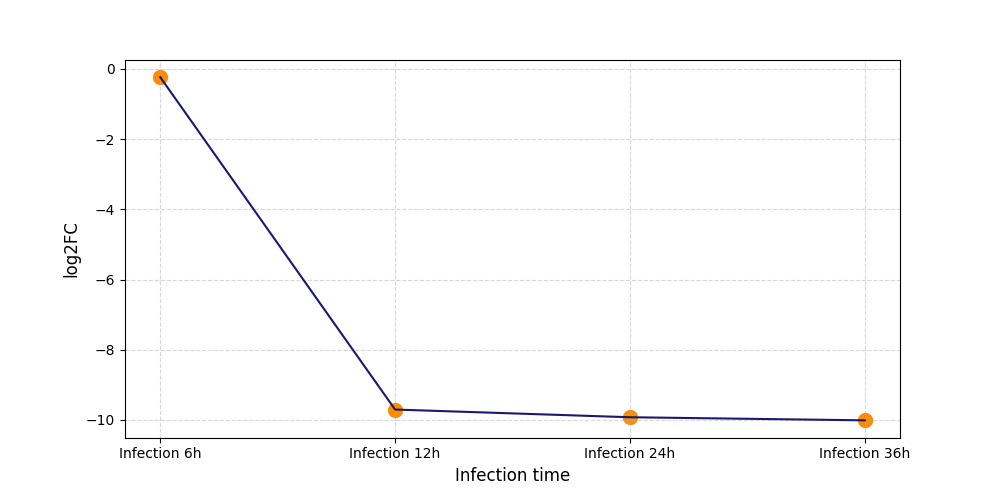
|
S414
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
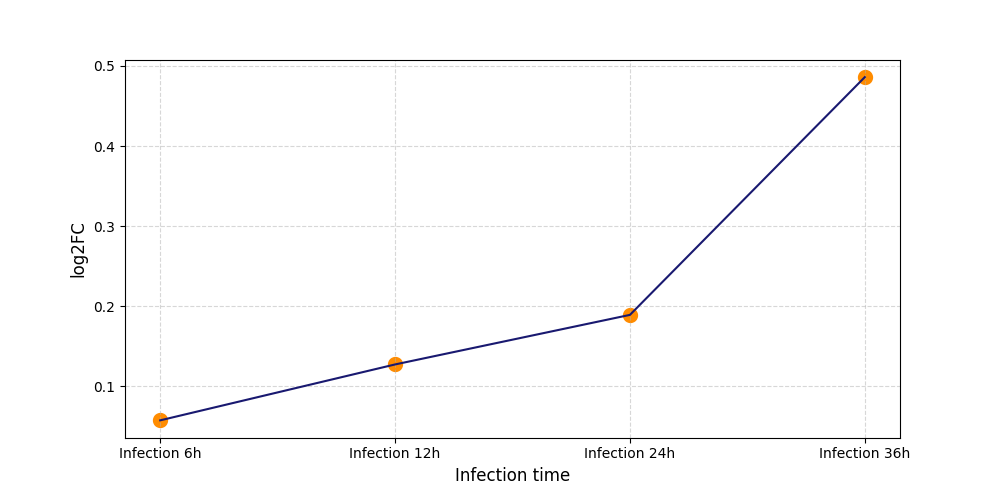
|
S426
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
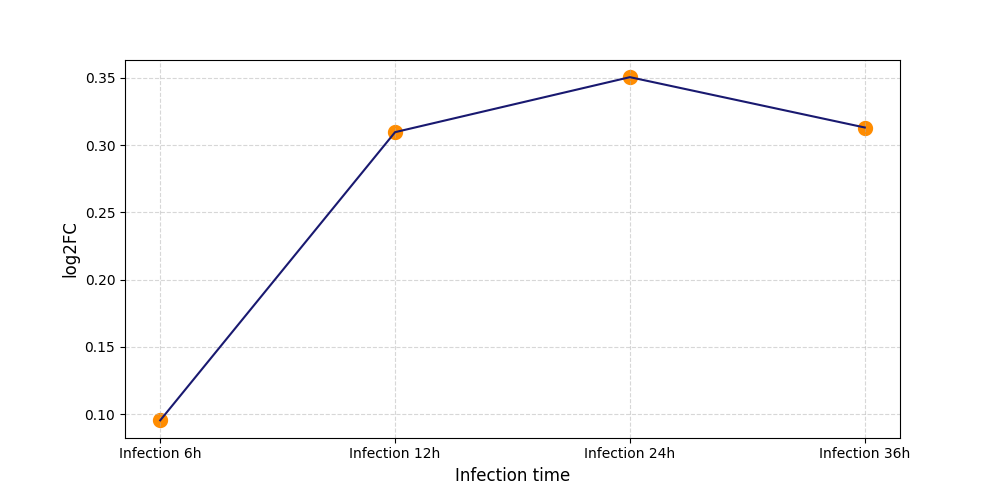
|
S429
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
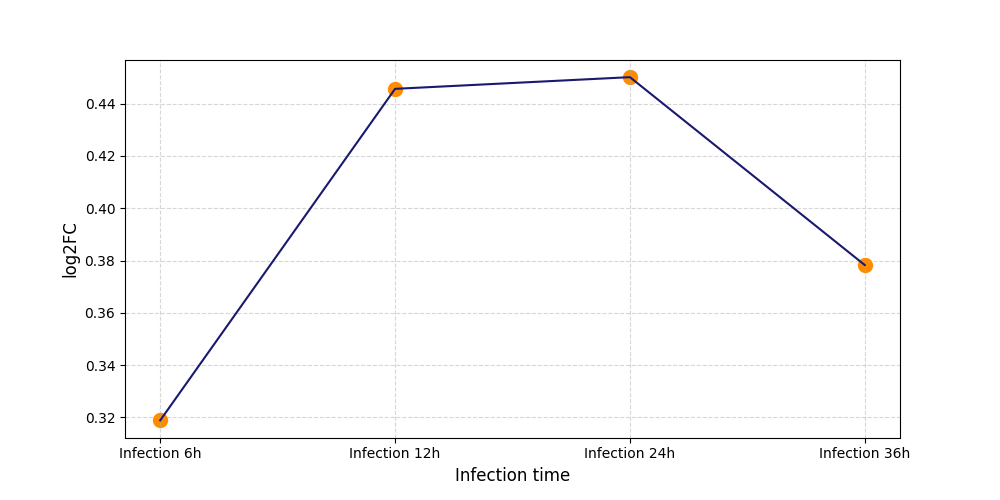
|
S431
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S450
[8] |
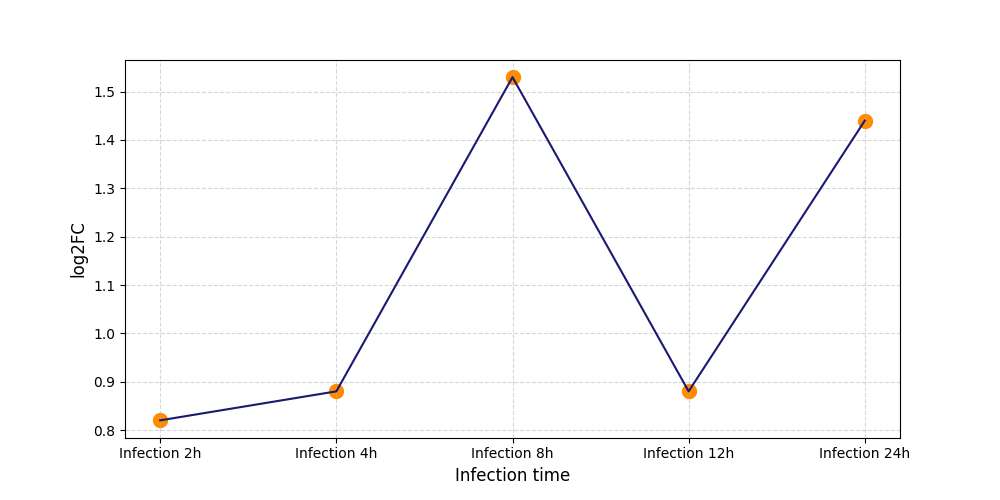 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S450
[9] |
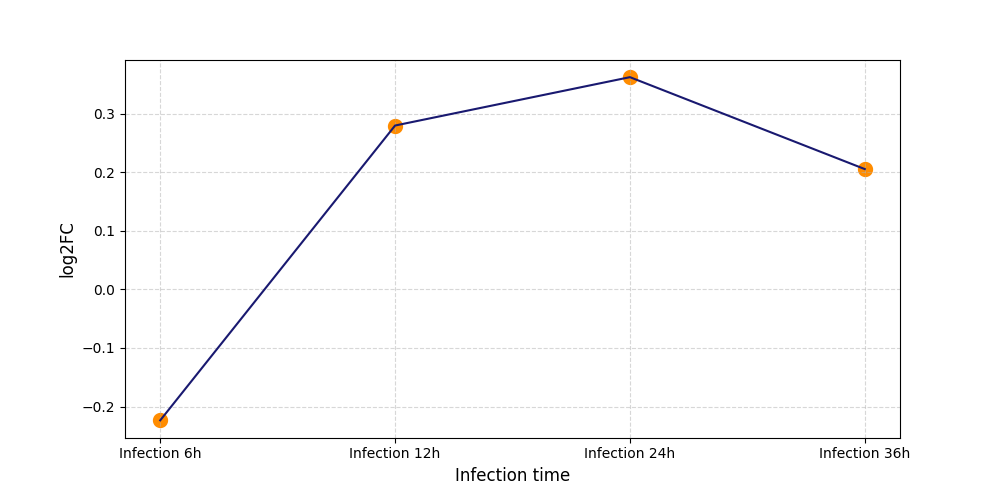 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S452
[8] |
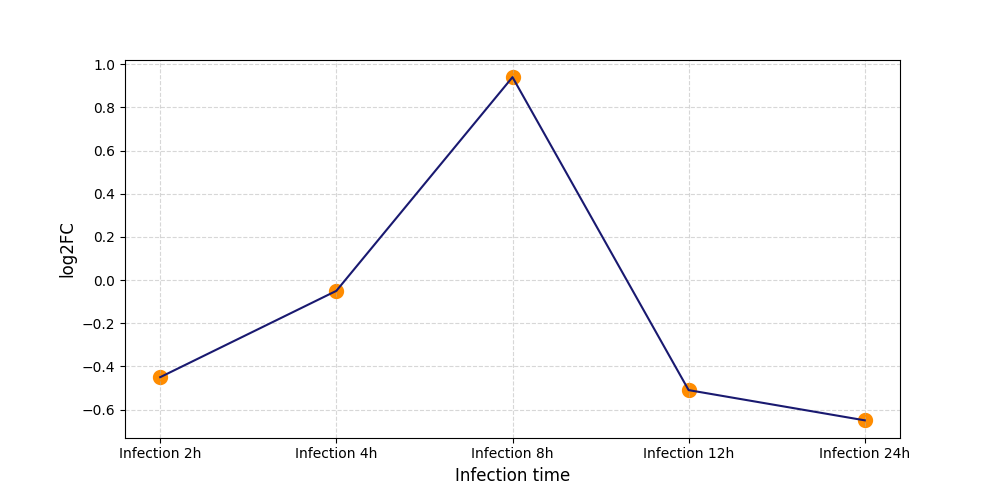 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S452
[9] |
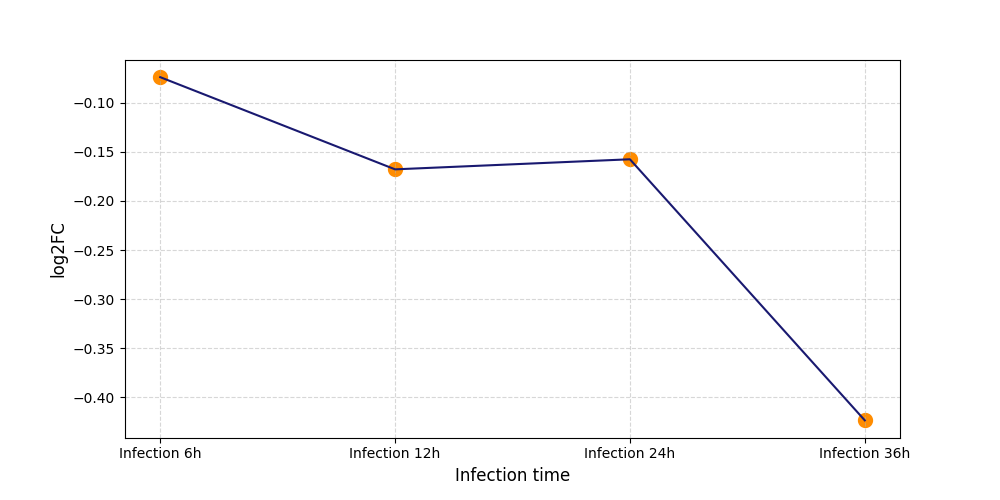 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S463
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S463
[9] |
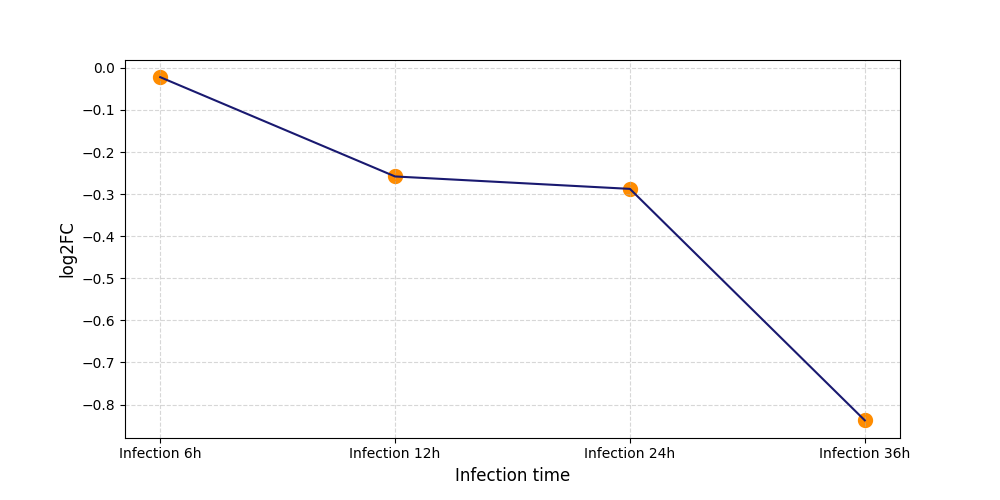 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S478
[9] |
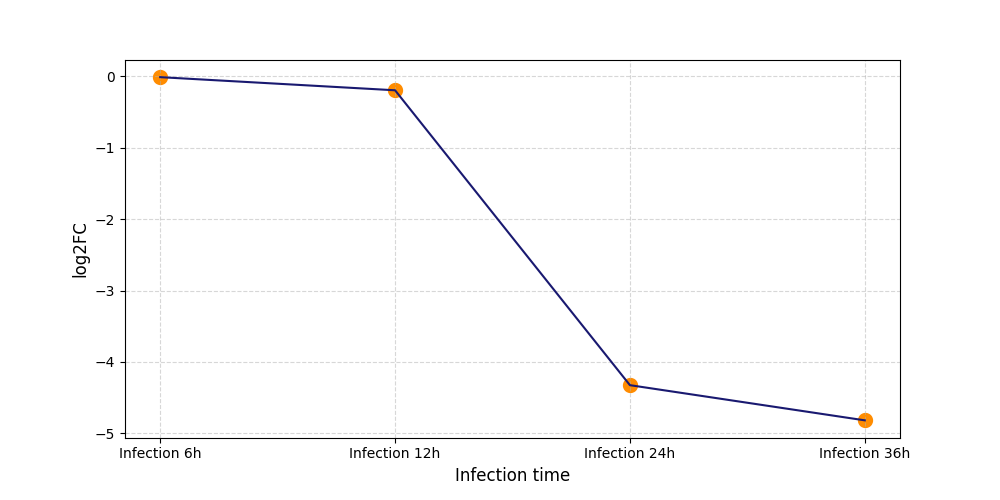 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S549
[9] |
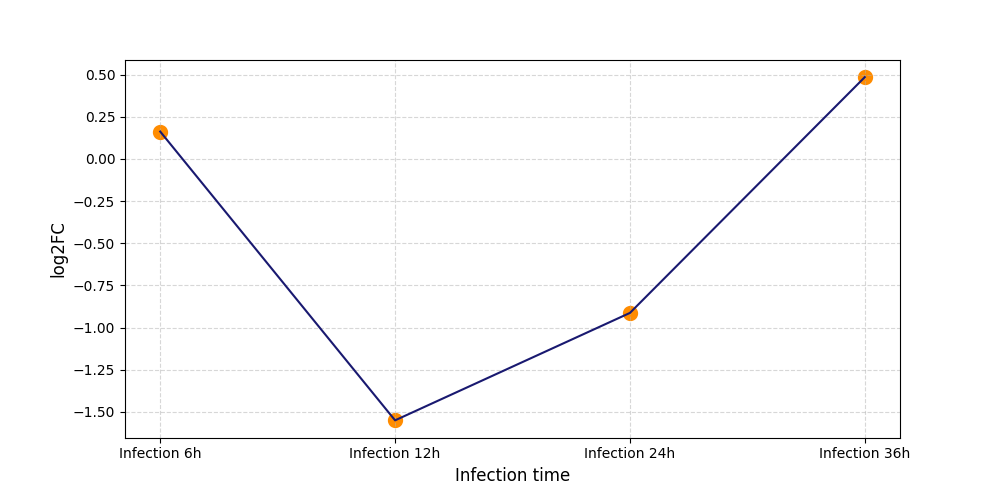 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S551
[9] |
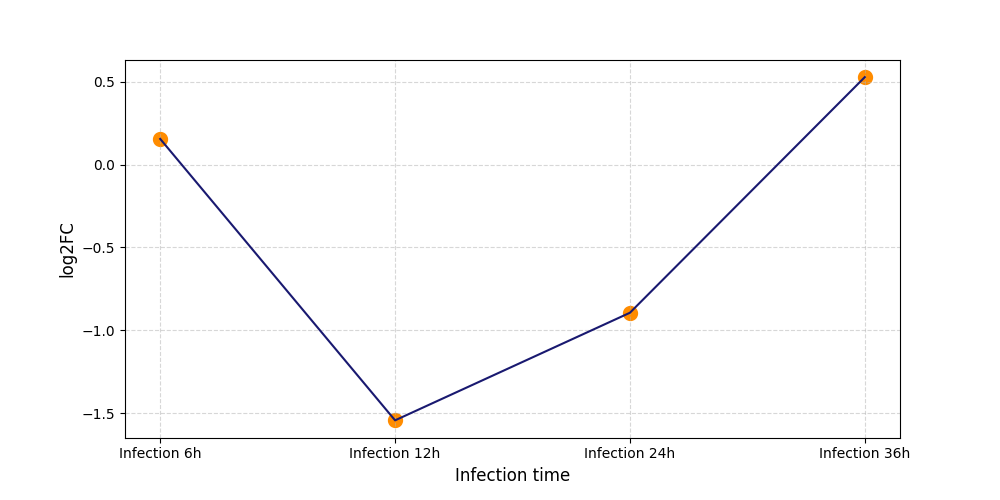 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S560
[8] |
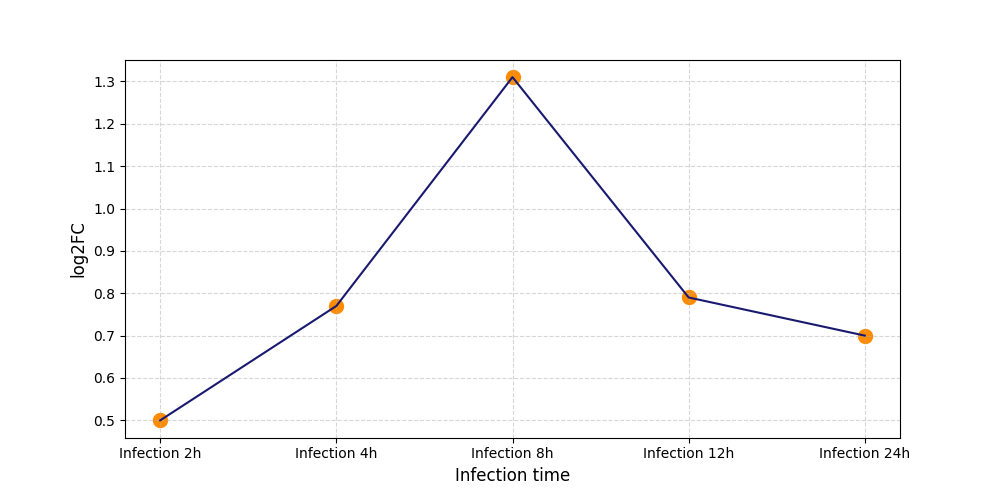 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S560
[9] |
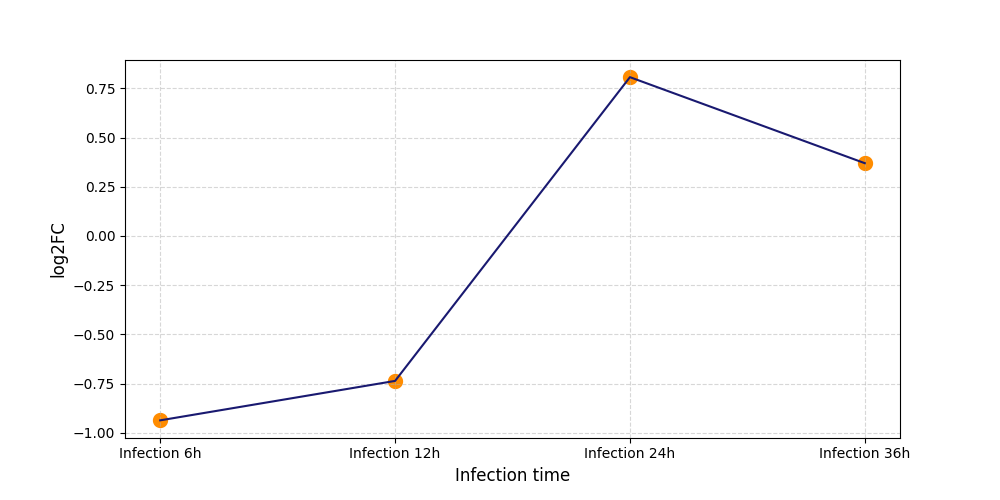 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S562
[8] |
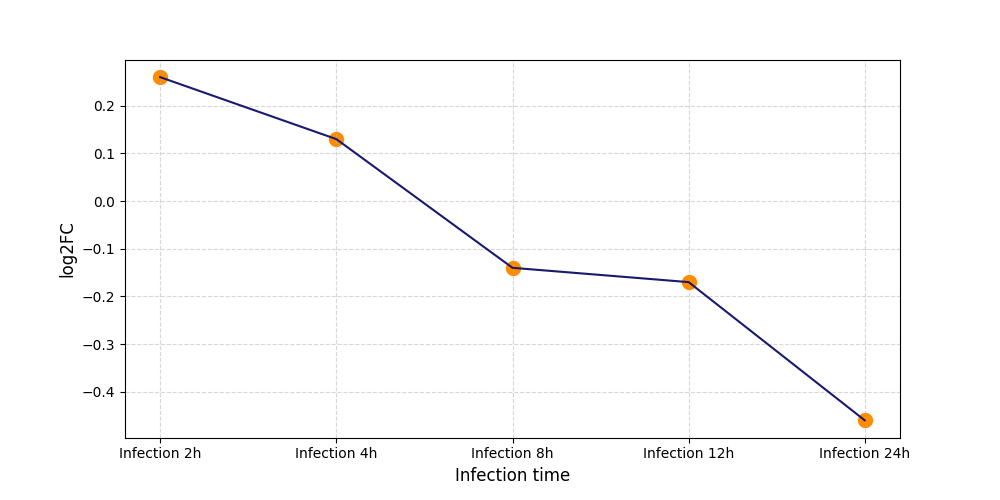 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S562
[9] |
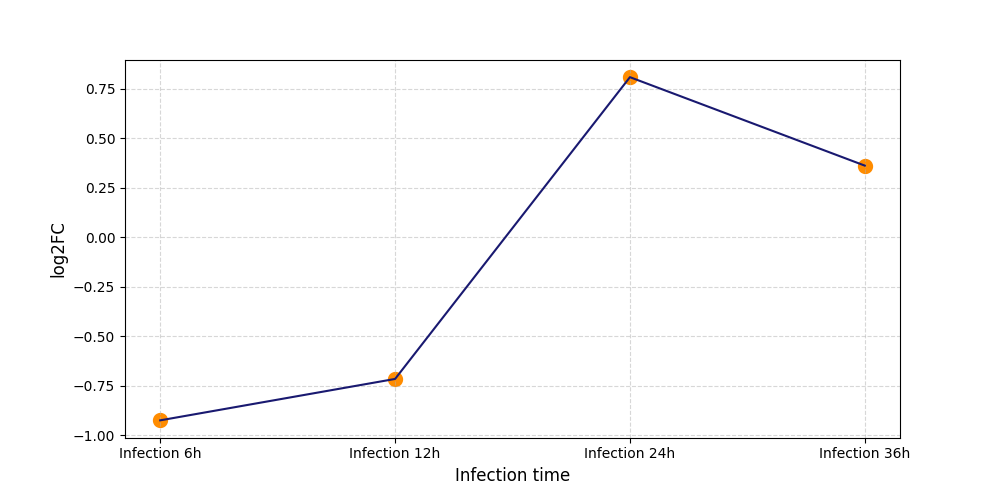 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S583
[9] |
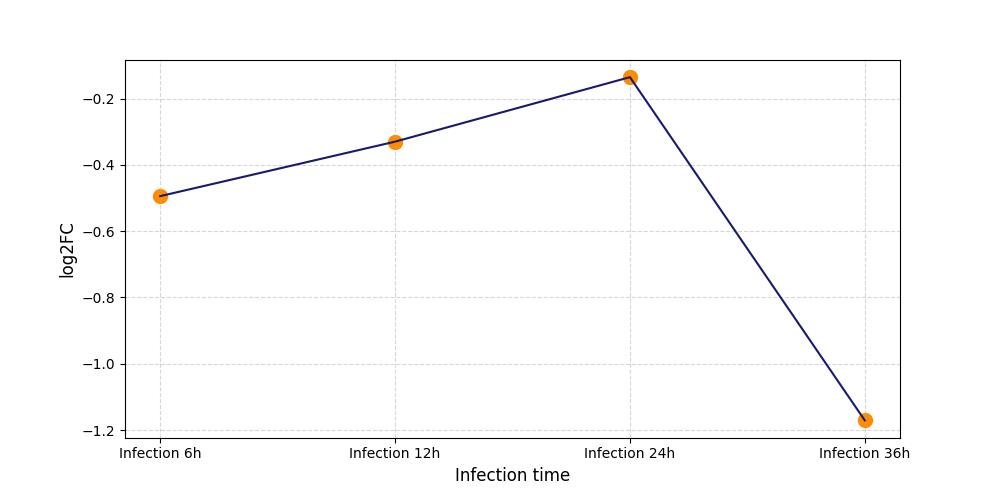 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S597
[8] |
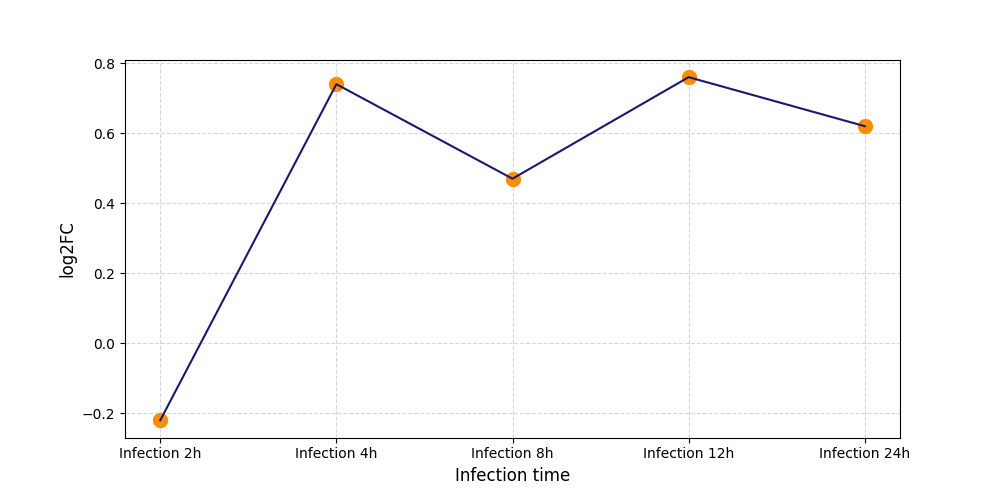 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S597
[9] |
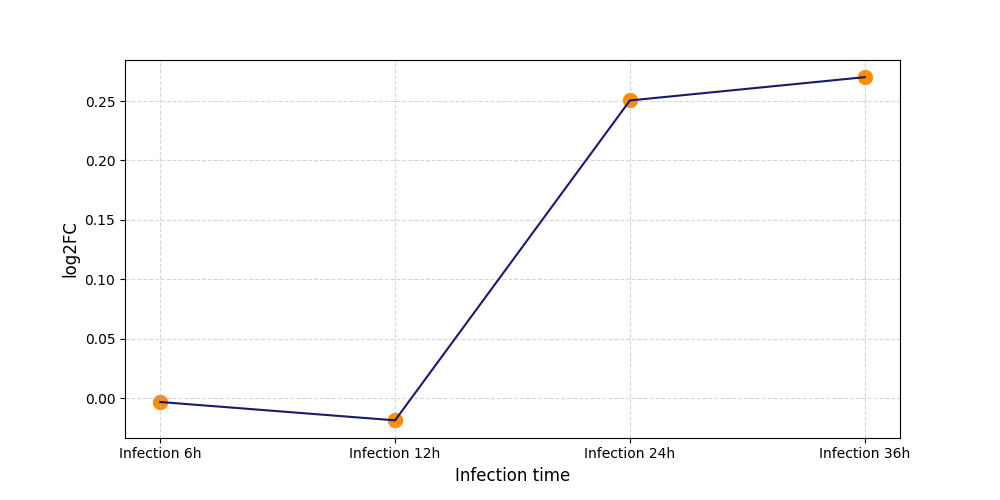 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S605
[9] |
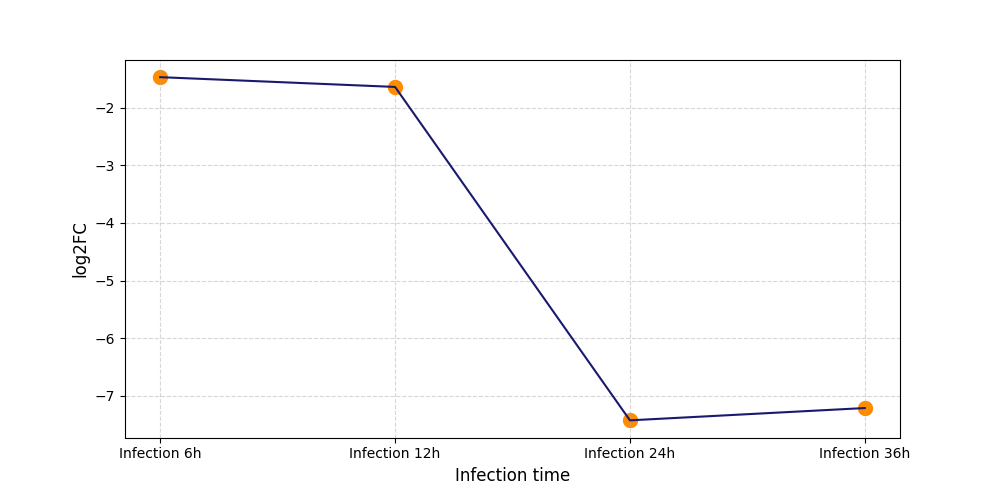 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S607
[9] |
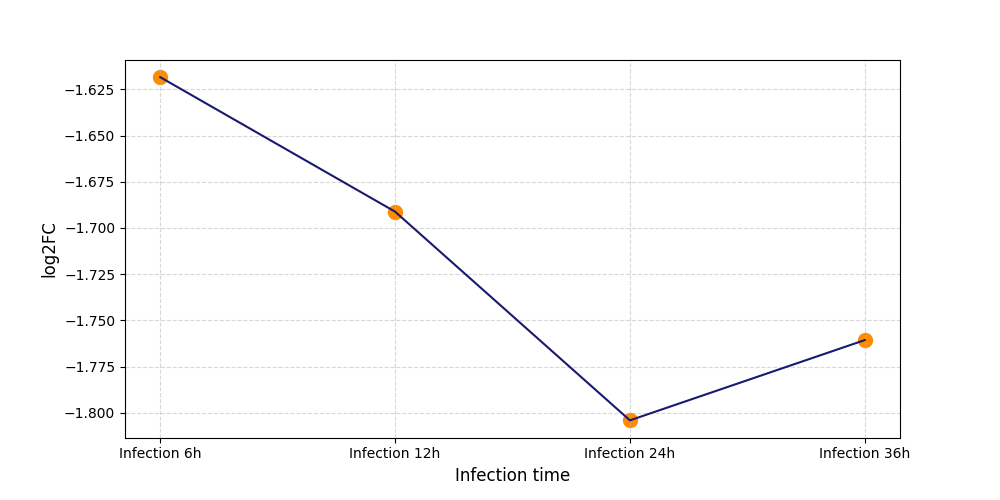 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S616
[8] |
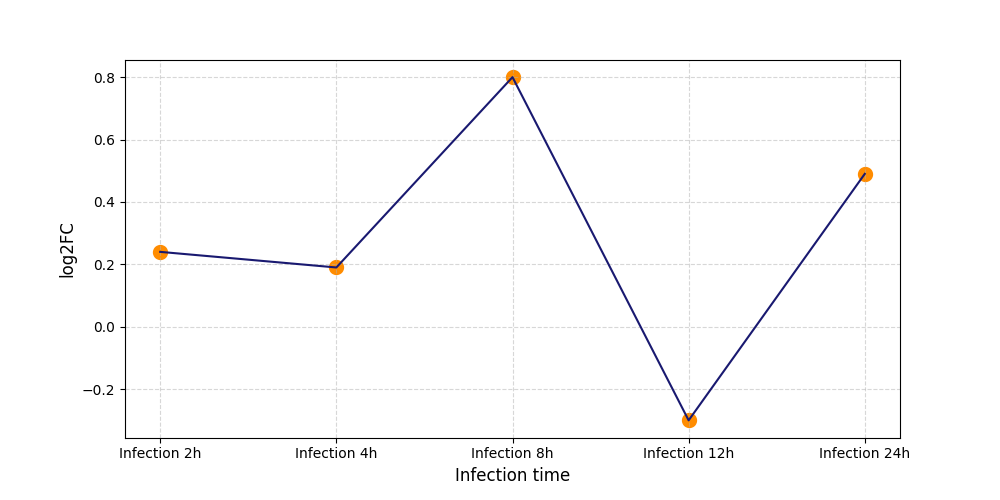 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S616
[9] |
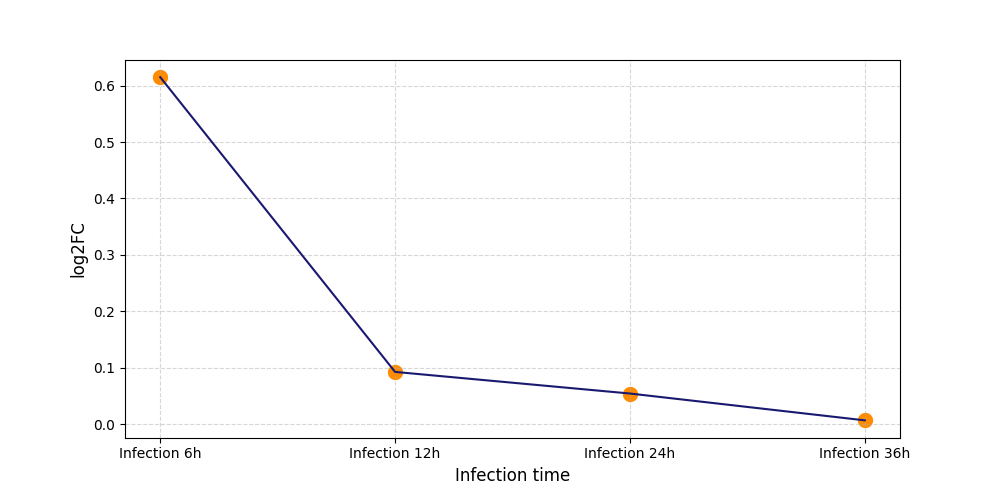 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S626
[9] |
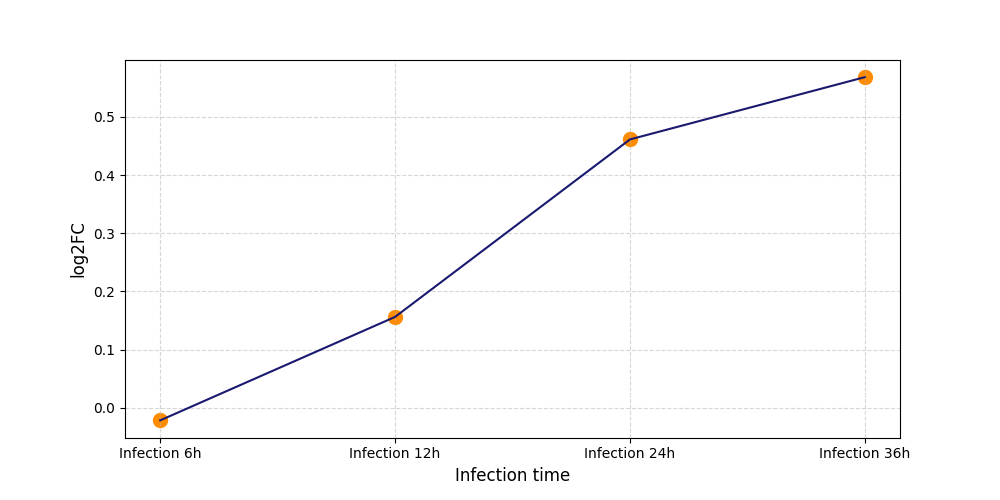 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S628
[9] |
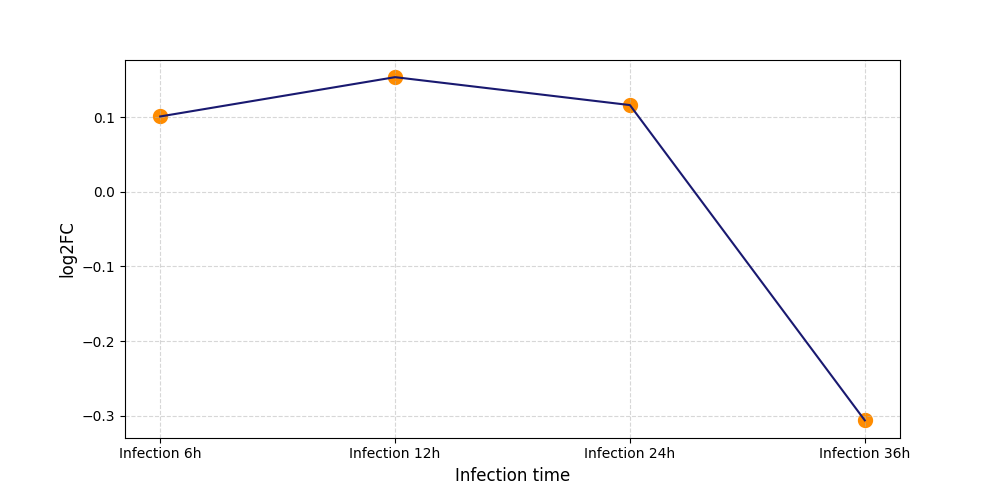 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S636
[9] |
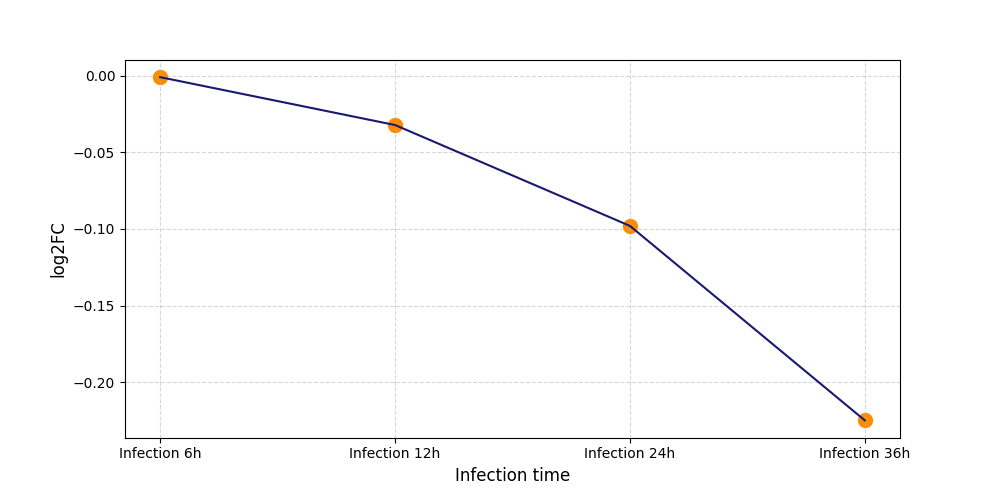 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S638
[9] |
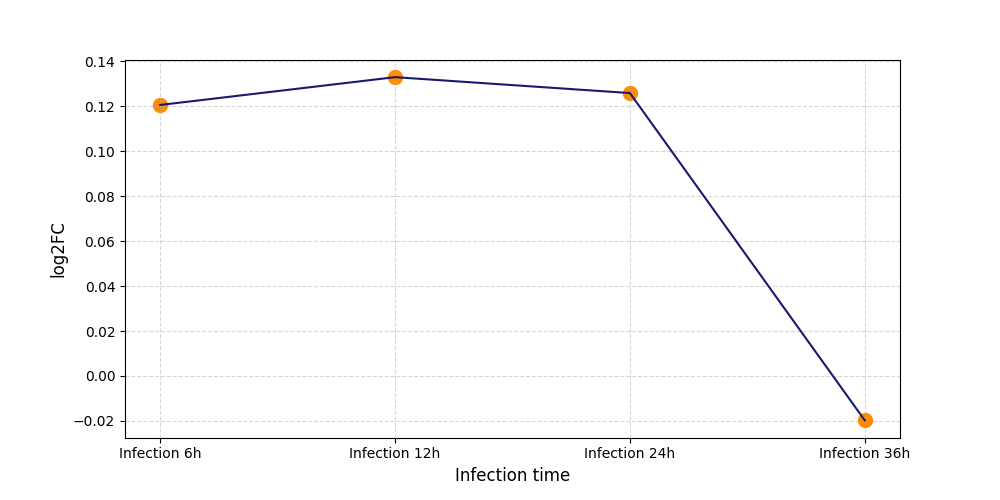 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S646
[9] |
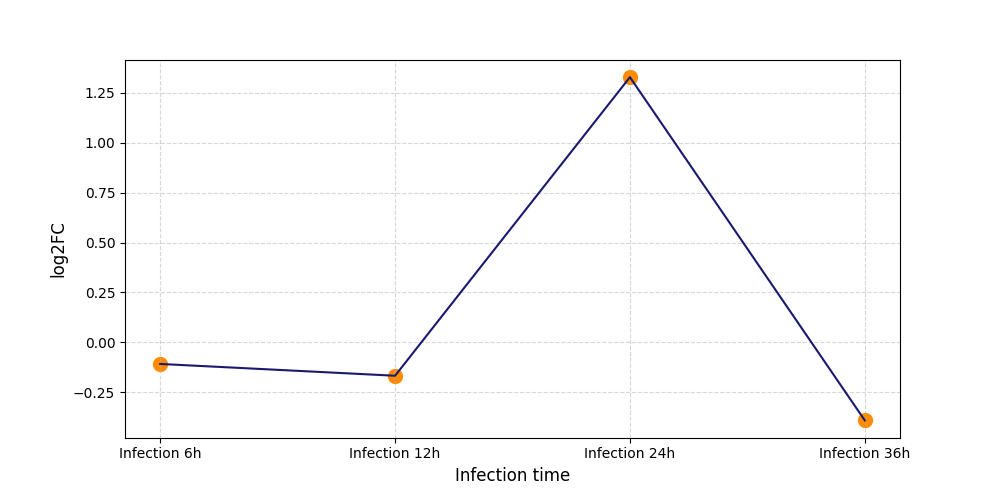 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S653
[9] |
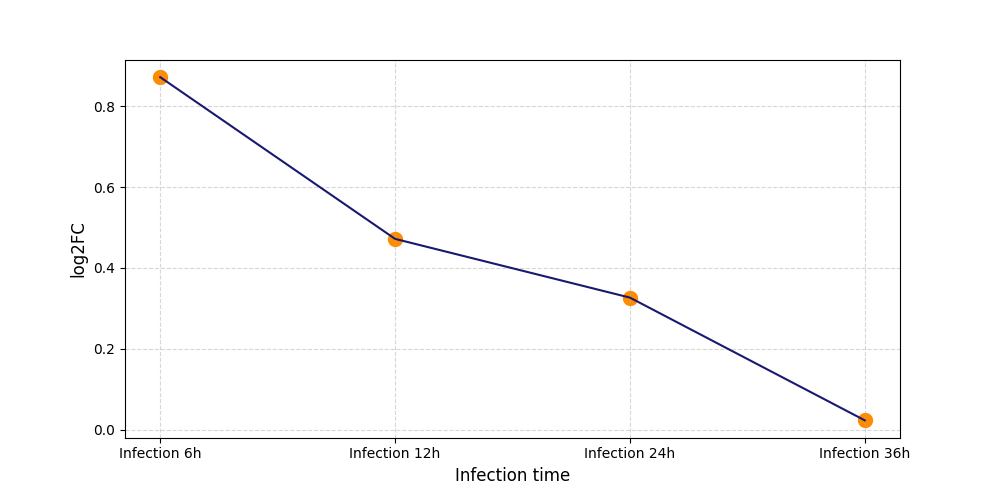 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S675
[9] |
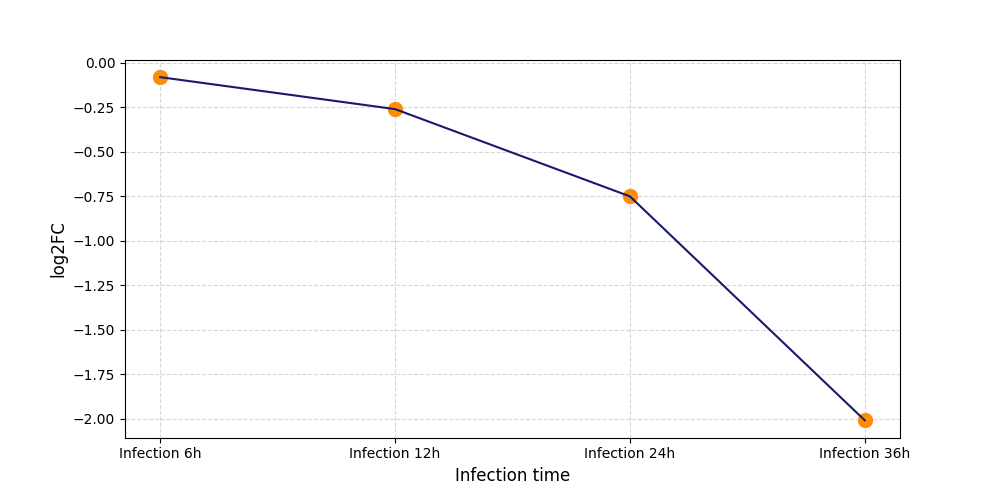 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S683
[9] |
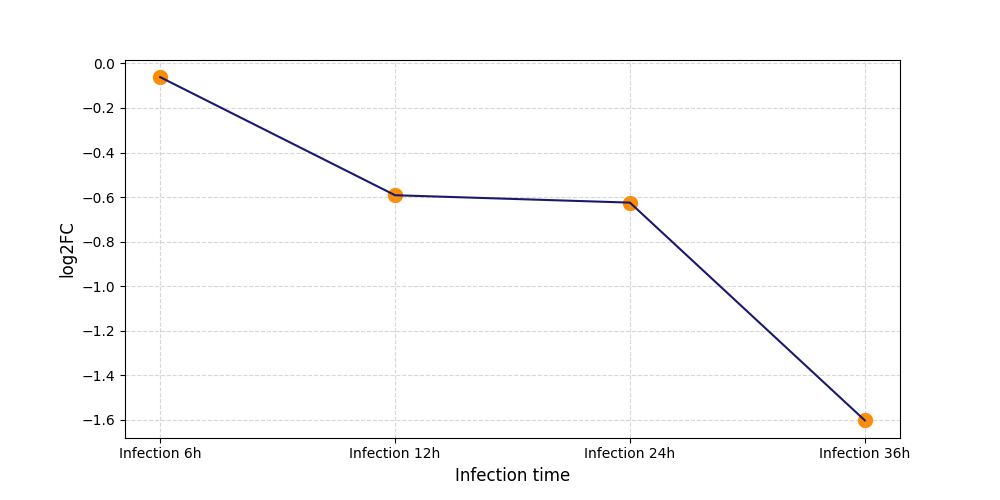 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S685
[9] |
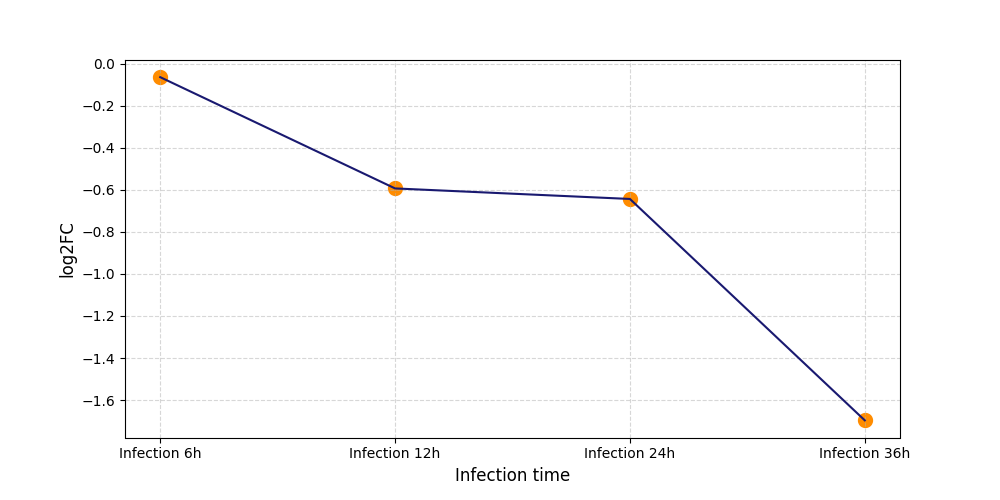 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
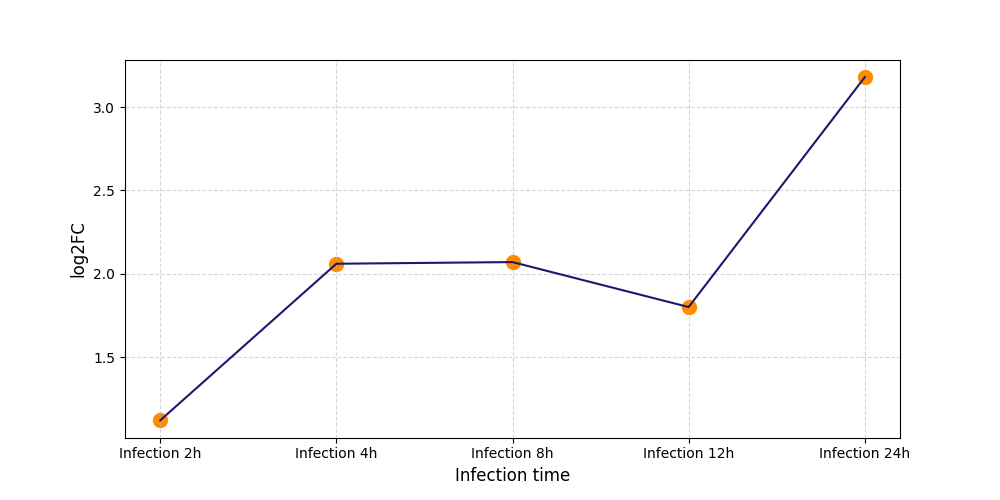
S696
[8] |
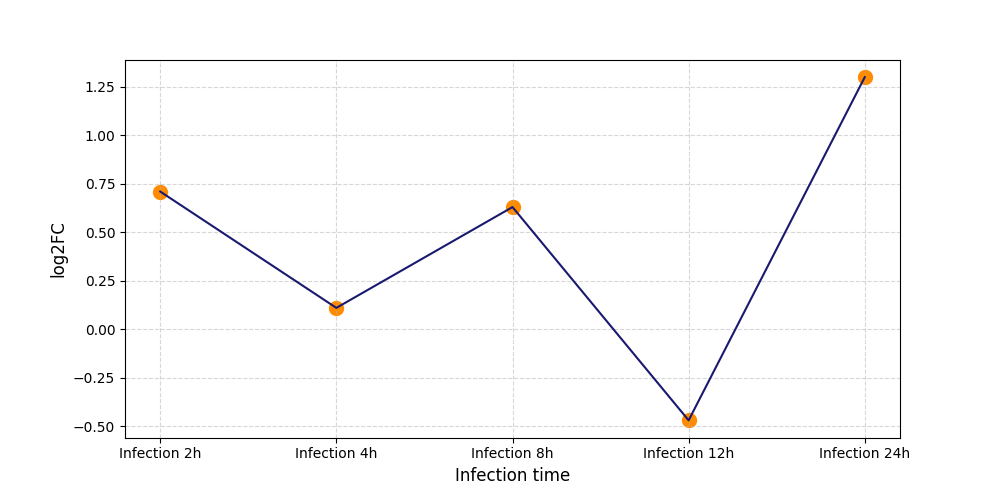 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S696
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
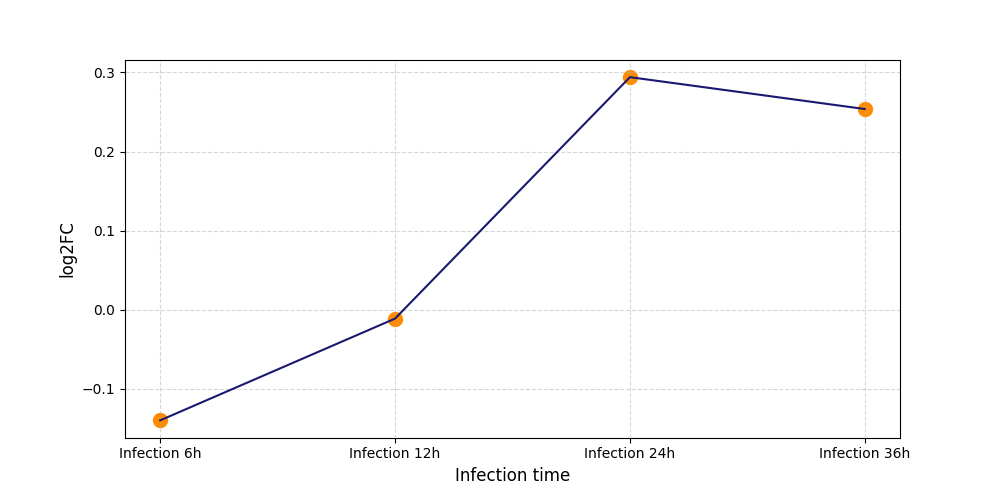
S713
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S713
[9] |
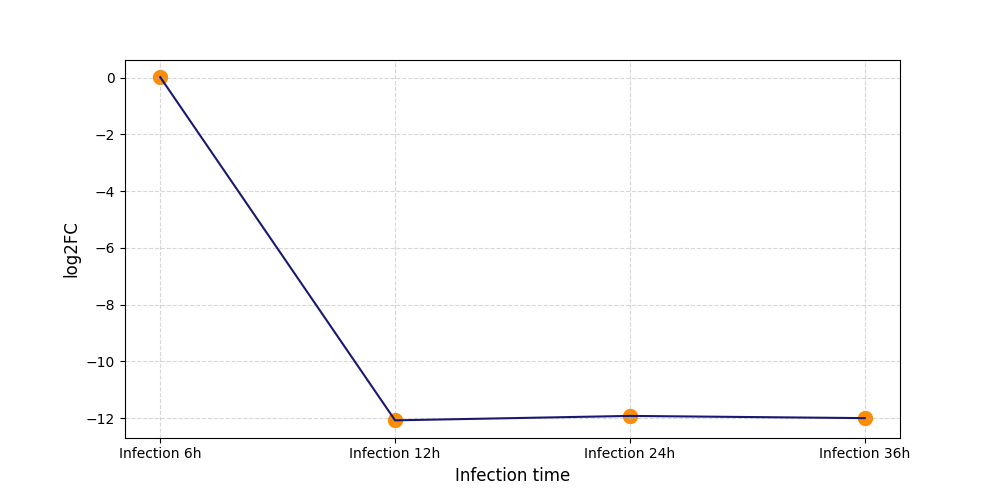 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S715
[8] |
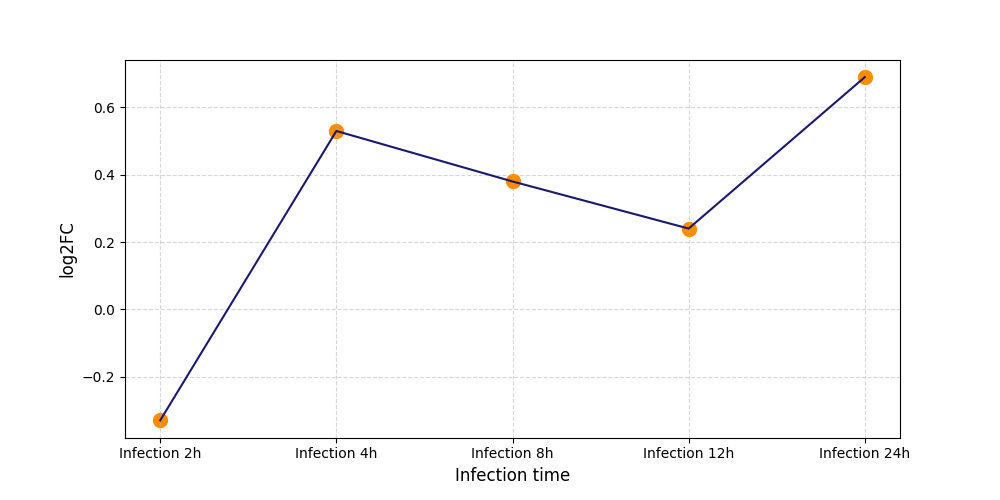 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S715
[9] |
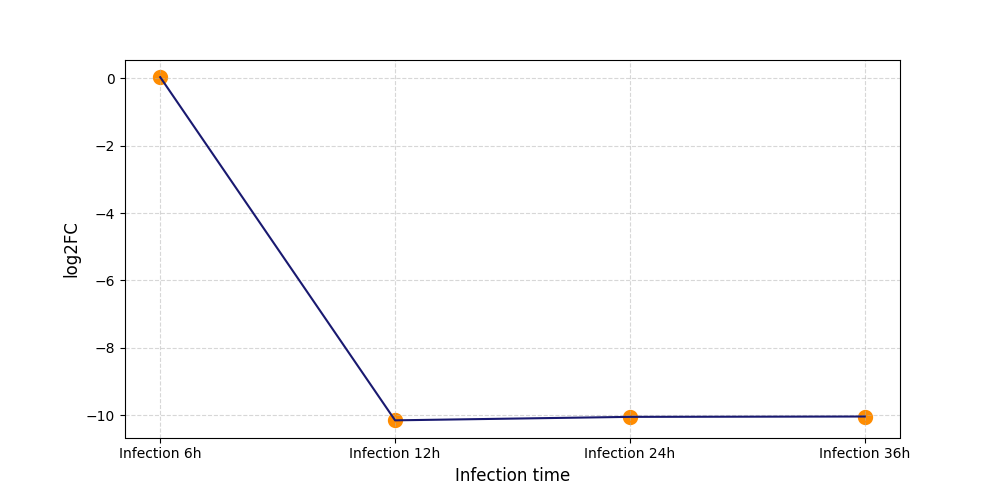 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S725
[9] |
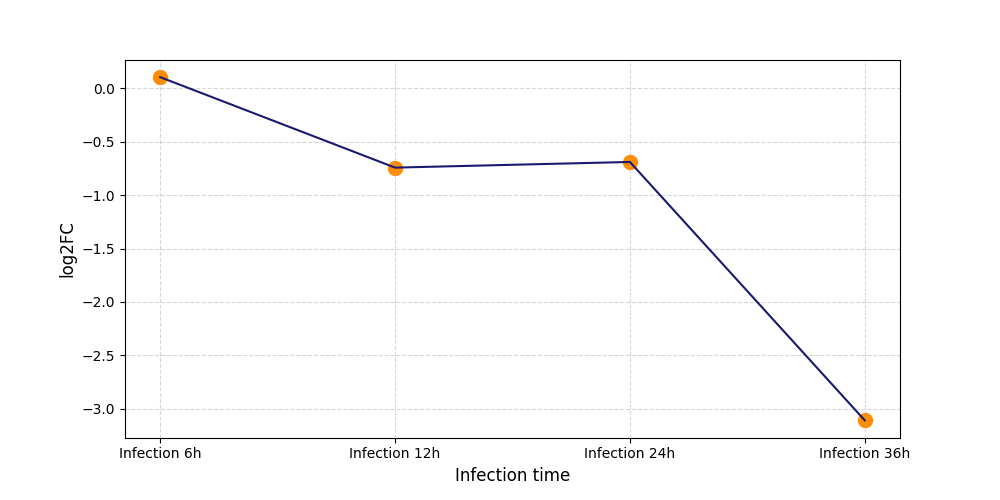 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S738
[8] |
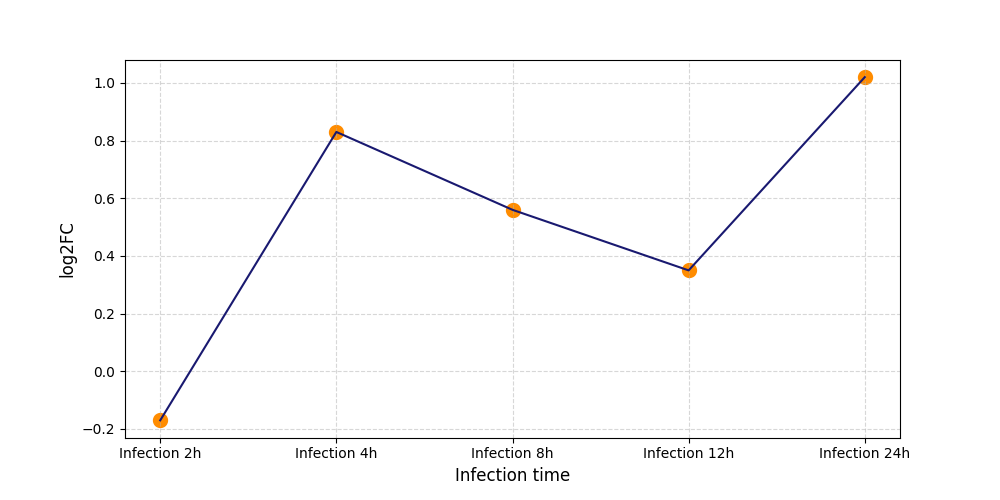 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S738
[9] |
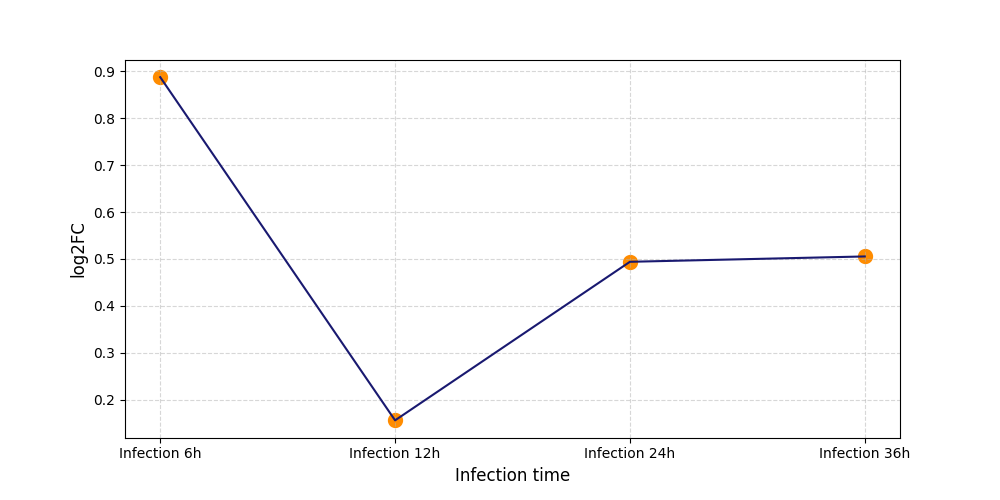 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S740
[9] |
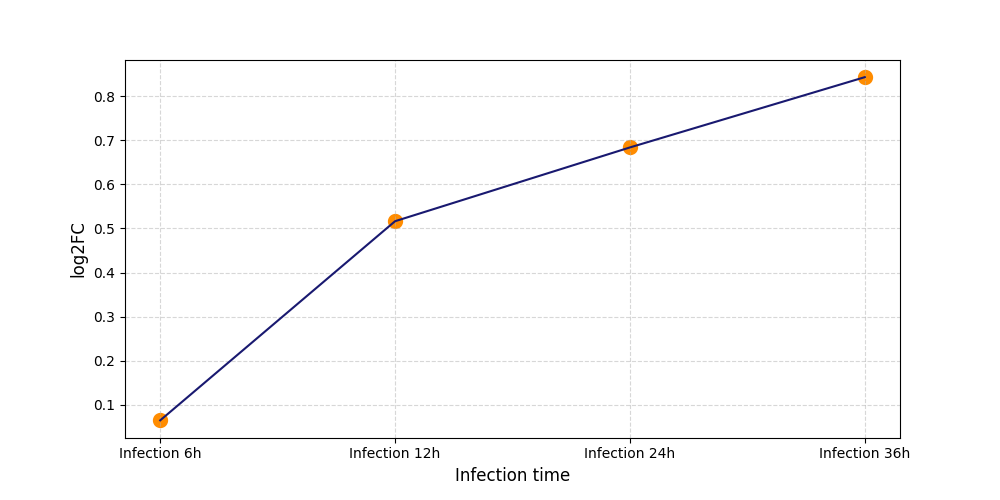 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S754
[8] |
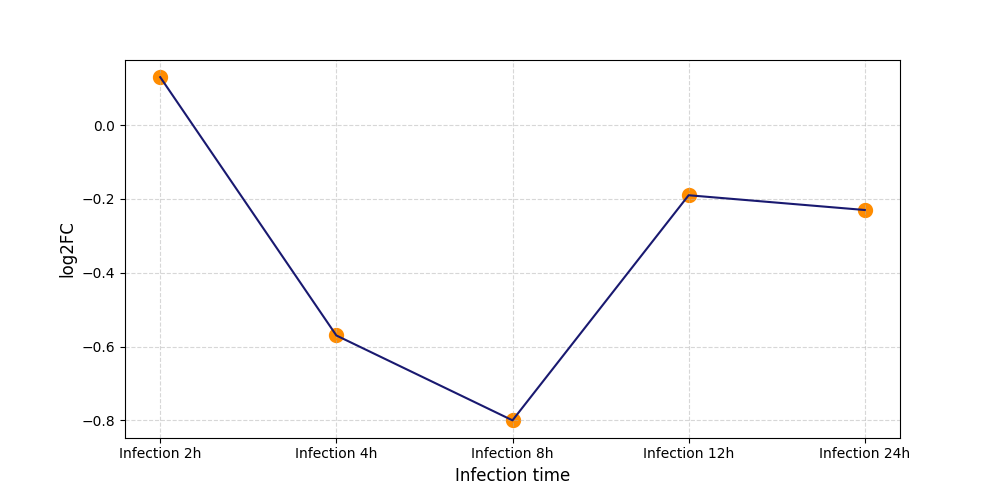 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S754
[9] |
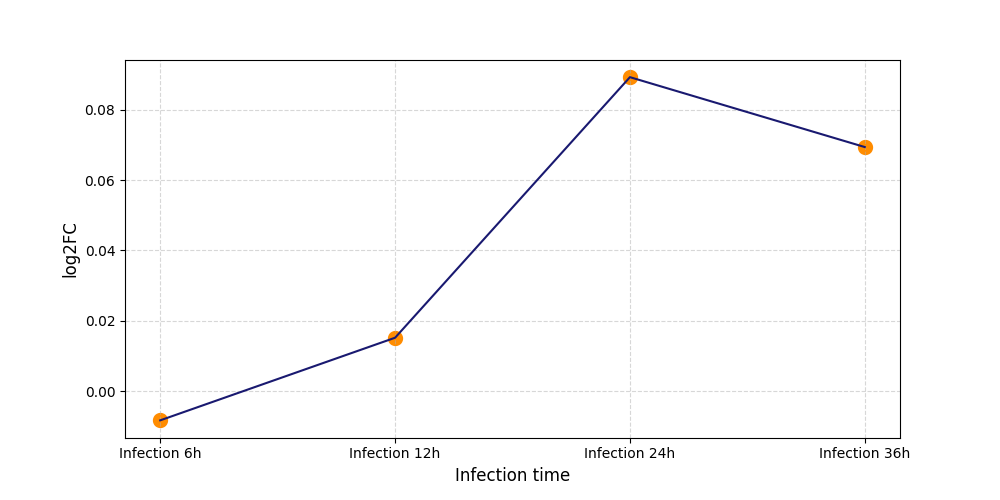 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S756
[8] |
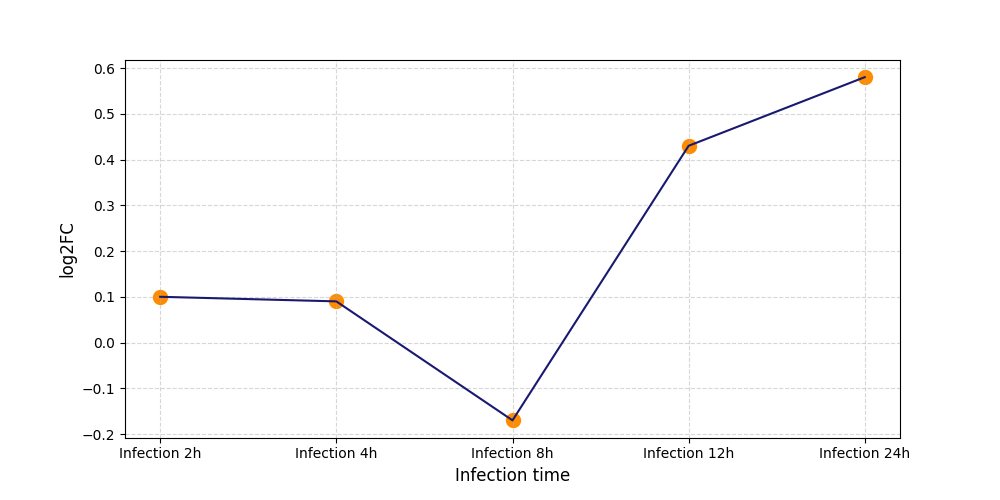 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S756
[9] |
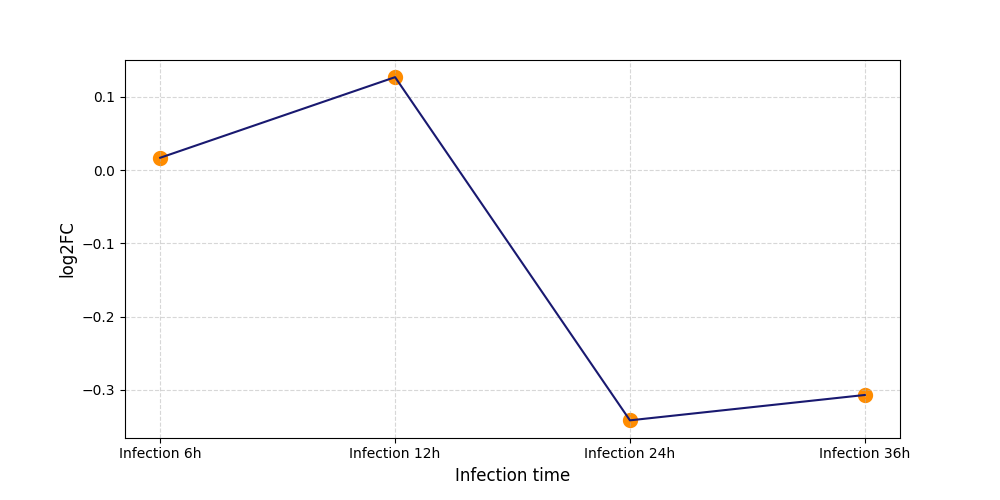 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S769
[9] |
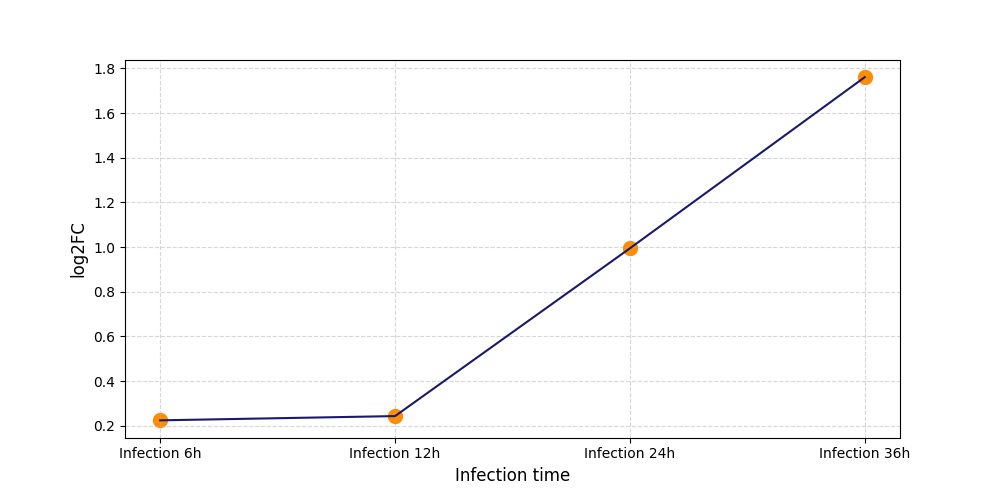 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S773
[9] |
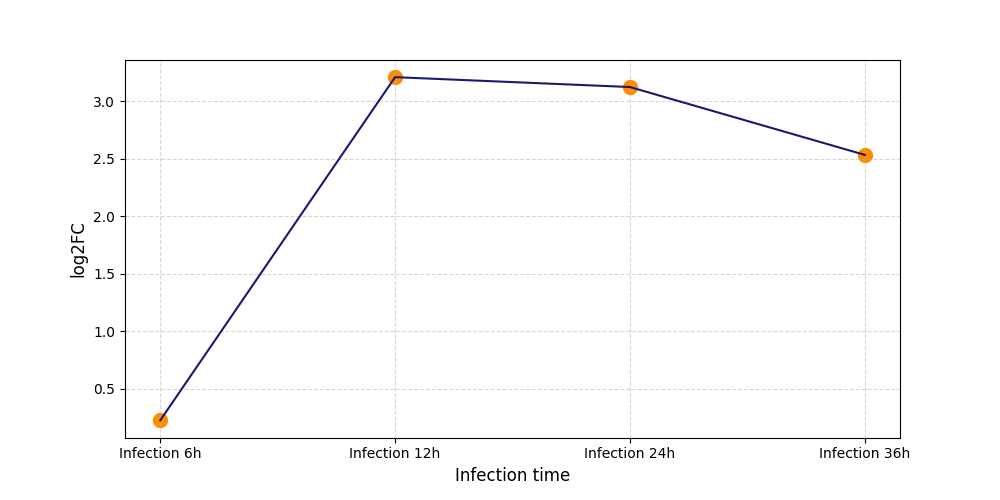 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S775
[9] |
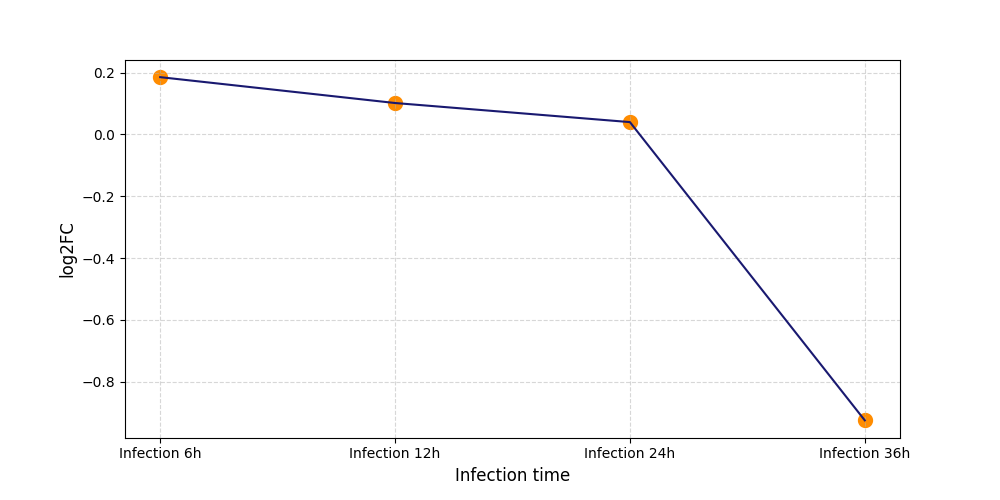 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S781
[9] |
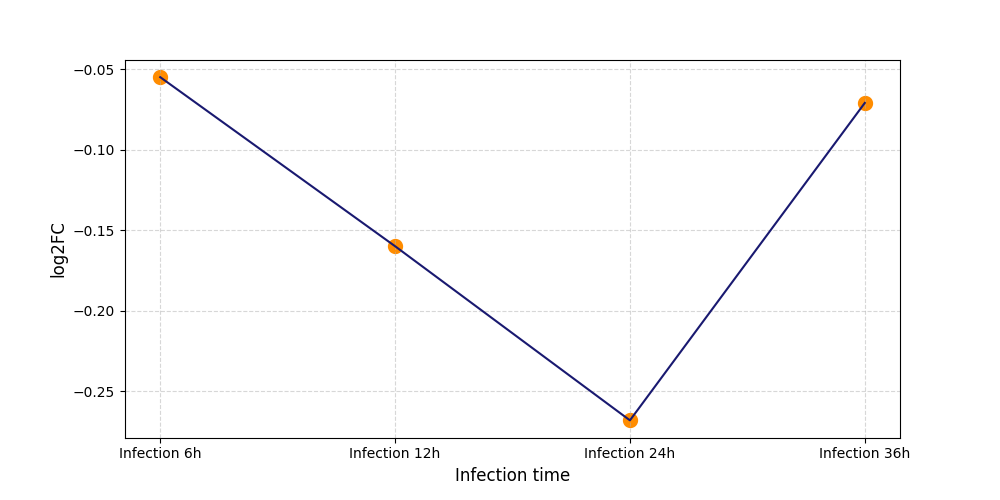 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S874
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T220
[9] |
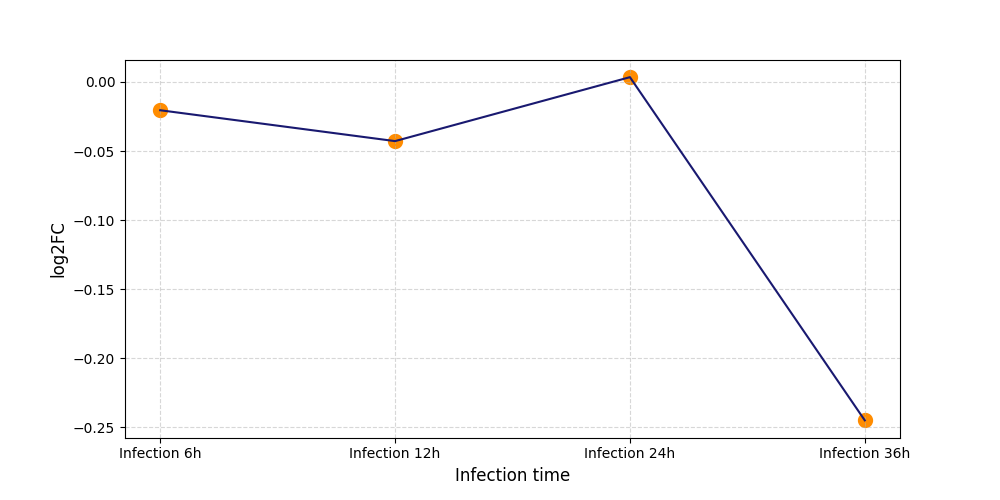 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T406
[9] |
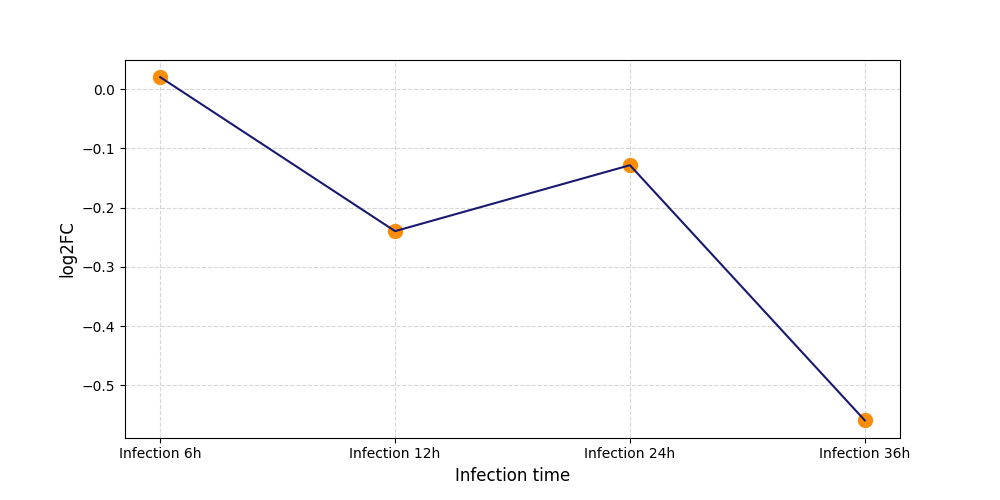 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T411
[9] |
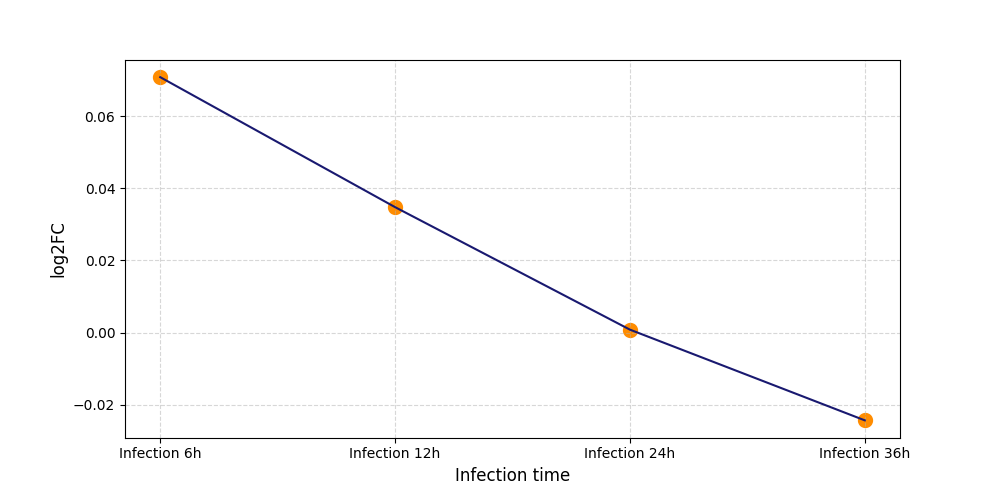 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T416
[9] |
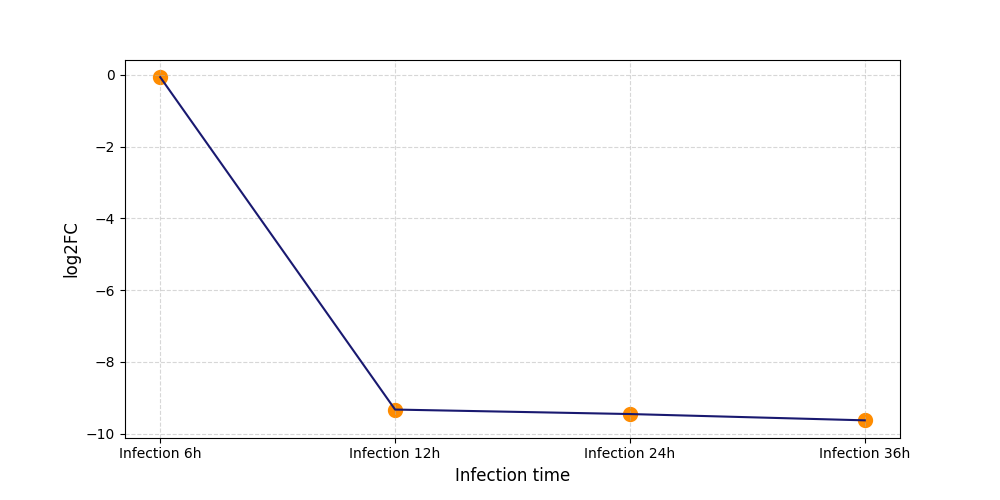 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T572
[9] |
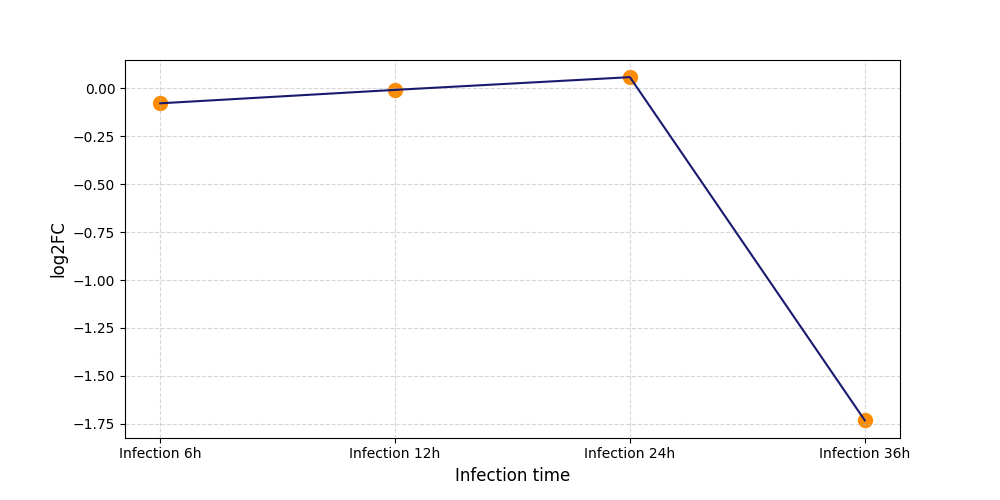 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T574
[9] |
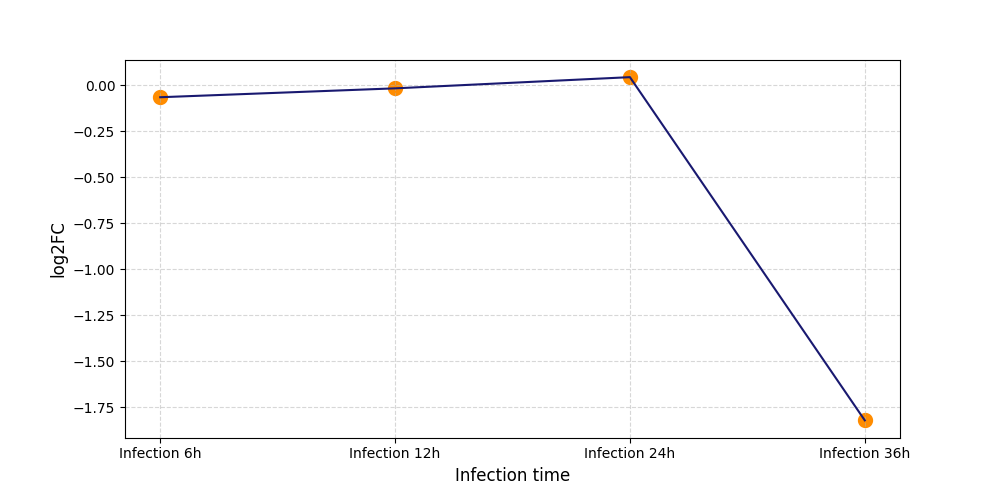 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T581
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T614
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T693
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T727
[9] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T778
[9] |
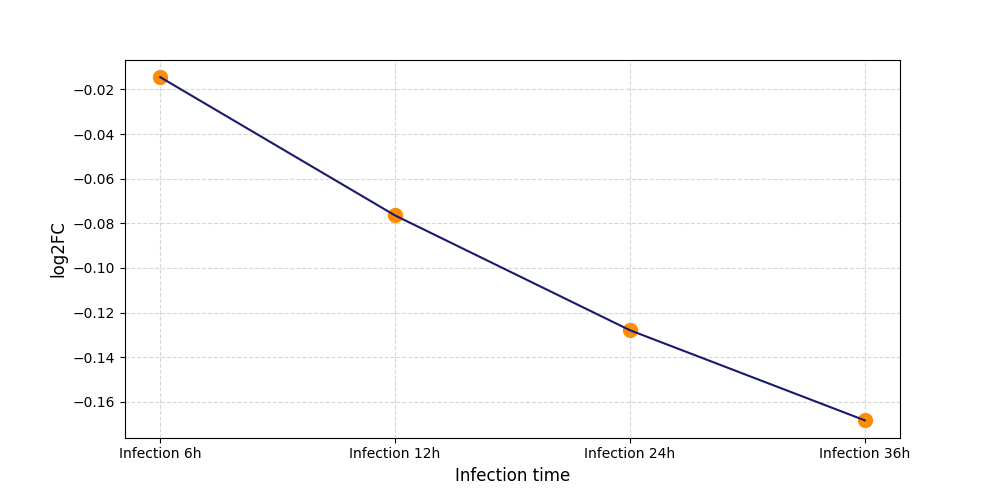 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T846
[9] |
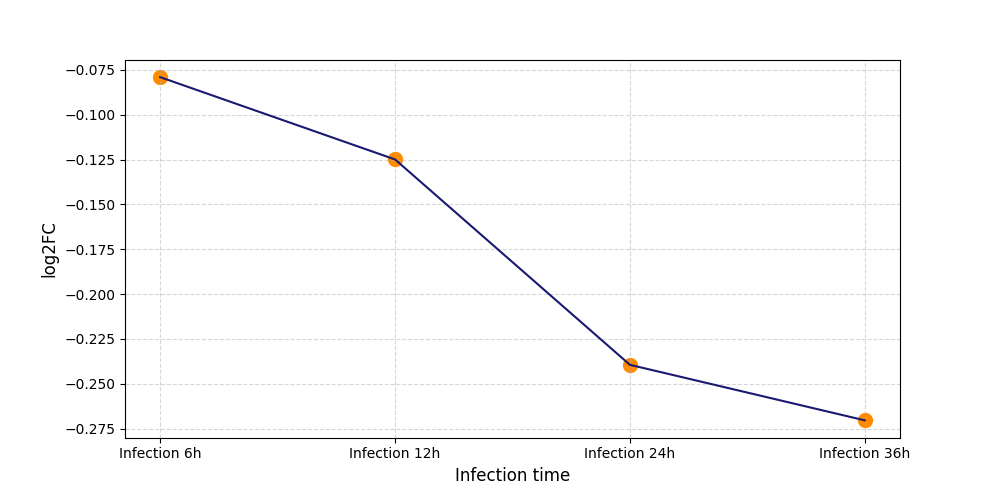 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T872
[9] |
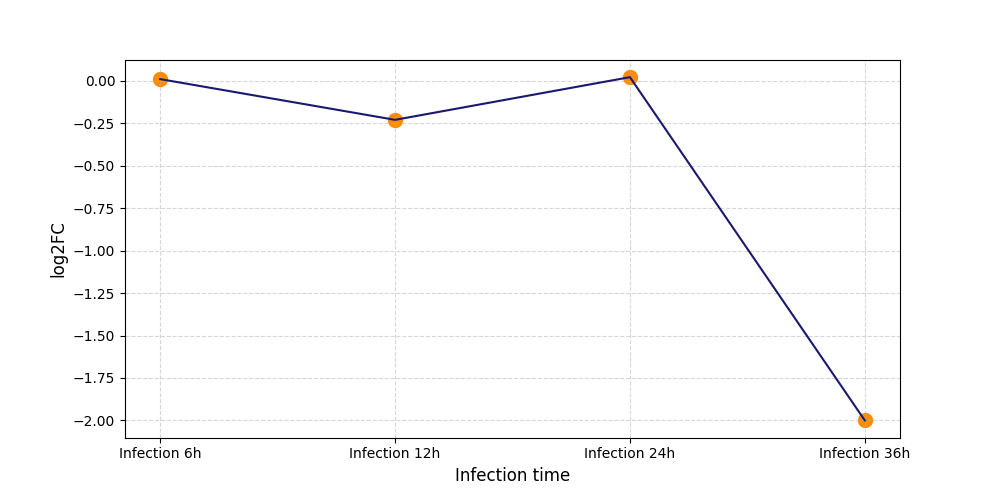 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Sequence Information |
MDAGFFRGTSAEQDNRFSNKQKKLLKQLKFAECLEKKVDMSKVNLEVIKPWITKRVTEILGFEDDVVIEFIFNQLEVKNPDSKMMQINLTGFLNGKNAREFMGELWPLLLSAQENIAGIPSAFLELKKEEIKQRQIEQEKLASMKKQDEDKDKRDKEEKESSREKRERSRSPRRRKSRSPSPRRRSSPVRRERKRSHSRSPRHRTKSRSPSPAPEKKEKTPELPEPSVKVKEPSVQEATSTSDILKVPKPEPIPEPKEPSPEKNSKKEKEKEKTRPRSRSRSKSRSRTRSRSPSHTRPRRRHRSRSRSYSPRRRPSPRRRPSPRRRTPPRRMPPPPRHRRSRSPVRRRRRSSASLSGSSSSSSSSRSRSPPKKPPKRTSSPPRKTRRLSPSASPPRRRHRPSPPATPPPKTRHSPTPQQSNRTRKSRVSVSPGRTSGKVTKHKGTEKRESPSPAPKPRKVELSESEEDKGGKMAAADSVQQRRQYRRQNQQSSSDSGSSSSSEDERPKRSHVKNGEVGRRRRHSPSRSASPSPRKRQKETSPRGRRRRSPSPPPTRRRRSPSPAPPPRRRRTPTPPPRRRTPSPPPRRRSPSPRRYSPPIQRRYSPSPPPKRRTASPPPPPKRRASPSPPPKRRVSHSPPPKQRSSPVTKRRSPSLSSKHRKGSSPSRSTREARSPQPNKRHSPSPRPRAPQTSSSPPPVRRGASSSPQRRQSPSPSTRPIRRVSRTPEPKKIKKAASPSPQSVRRVSSSRSVSGSPEPAAKKPPAPPSPVQSQSPSTNWSPAVPVKKAKSPTPSPSPPRNSDQEGGGKKKKKKKDKKHKKDKKHKKHKKHKKEKAVAAAAAAAVTPAAIAAATTTLAQEEPVAAPEPKKETESEAEDNLDDLEKHLREKALRSMRKAQVSPQS
Click to Show/Hide
|
|---|