Details of Host Protein
| Host Protein General Information (ID: PT1171) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name |
Treslin (TICRR)
|
Gene Name |
TICRR
|
||||||
| Host Species |
Homo sapiens
|
Uniprot Entry Name |
TICRR_HUMAN
|
||||||
| Protein Families |
Treslin family
|
||||||||
| Subcellular Location |
Nucleus
|
||||||||
| External Link | |||||||||
| NCBI Gene ID | |||||||||
| Uniprot ID | |||||||||
| Ensembl ID | |||||||||
| HGNC ID | |||||||||
| Function in Host |
Regulator of DNA replication and S/M and G2/M checkpoints. Regulates the triggering of DNA replication initiation via itsinteraction with TOPBP1 by participating in CDK2-mediated loading ofCDC45L onto replication origins. Required for the transition from pre-replication complex (pre-RC) to pre-initiation complex (pre-IC). Required to prevent mitotic entry after treatment with ionizingradiation.
[1]
Click to Show/Hide
|
||||||||
| 3D Structure |
|
||||||||
| Function of This Protein During Virus Infection | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Virus Name | SARS-COV-2 | Protein Function | Pro-viral | [2] | |||||
| Infected Tissue | Lung | Infection Time | 7-9 Days | ||||||
| Infected Cell | Calu-3 Cells (Human epithelial cell line) | Cellosaurus ID | CVCL_0609 | ||||||
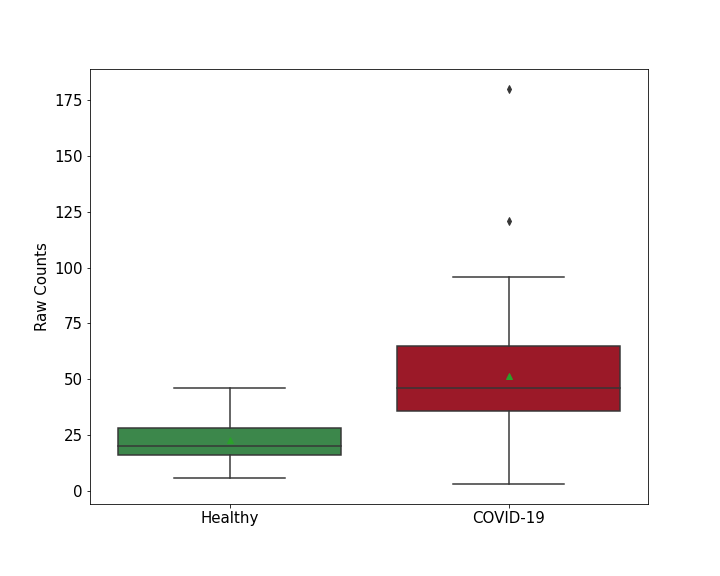
| Method Description | To detect the role of host protein TICRR in viral infection, TICRR protein knockout Calu-3 Cells were infected with SARS-COV-2 for 7 - 9 Days , and the effects on infection was detected through CRISPR-based genome-wide gene-knockout screen. | ||||||||
| Results | It is reported that knockout of TICRR leads to the decreased SARS-CoV-2 RNA levels compared with control group. | ||||||||
| Full List of Virus RNA Interacting with This Protien | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| RNA Region: 3'-UTR (hCoV-19/Wuhan-Hu-1/2019 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[3] | |||||||
| Strains Name |
hCoV-19/Wuhan-Hu-1/2019
|
||||||||
| Strains Family |
Beta (B.1.351)
|
||||||||
| RNA Binding Region |
3'-UTR
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Infection Cells | HEK293 Cells (Human embryonic kidney cell) (CVCL_0045 ) | ||||||||
| Cell Originated Tissue | Liver | ||||||||
| Infection Time | 48 h | ||||||||
| Interaction Score | Prot score = 29 | ||||||||
| Method Description | RNA-protein interaction detection (RaPID) assay; liquid chromatography with tandem mass spectrometry (LC-MS/MS) | ||||||||
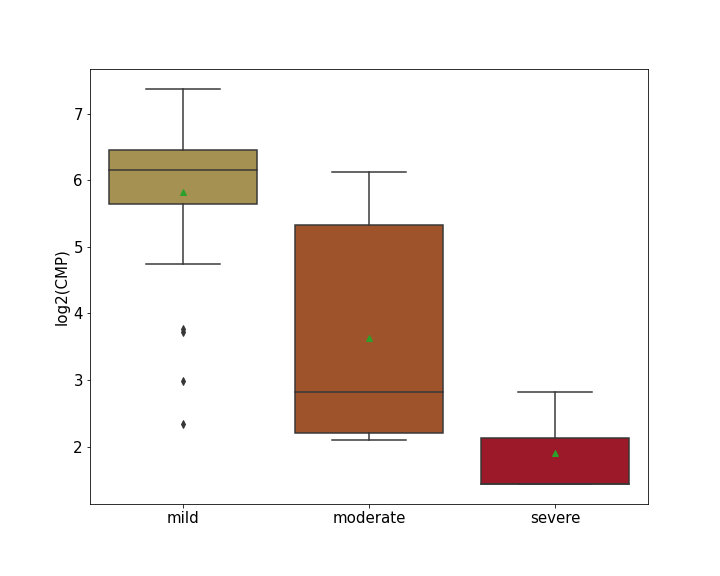
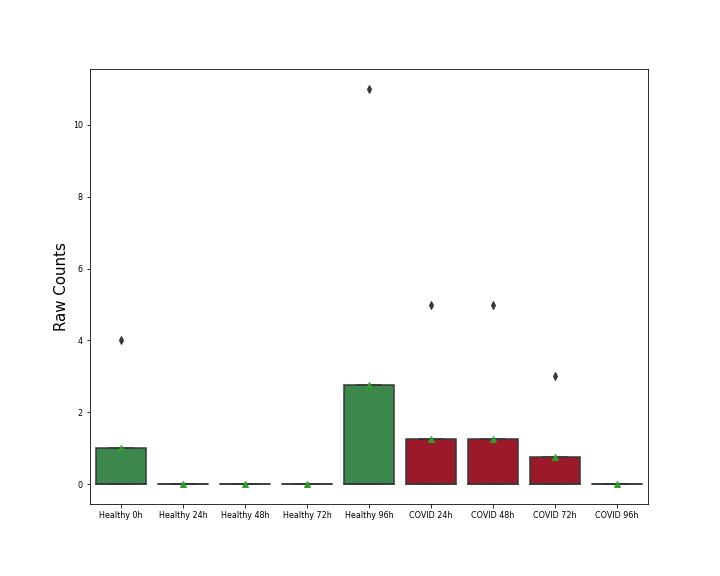
| Differential Gene Expression During SARS-COV-2 Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Phosphorylation after Virus Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
S1026
[4] |
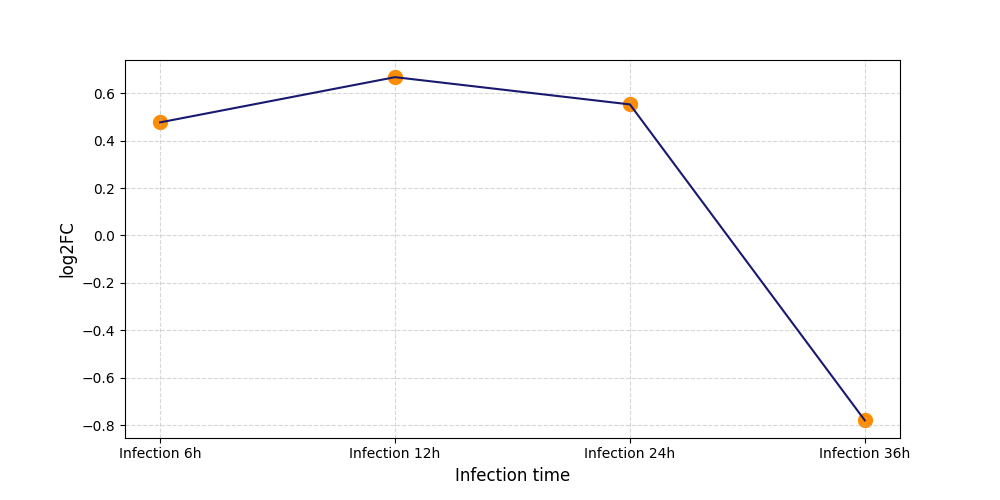 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1045
[4] |
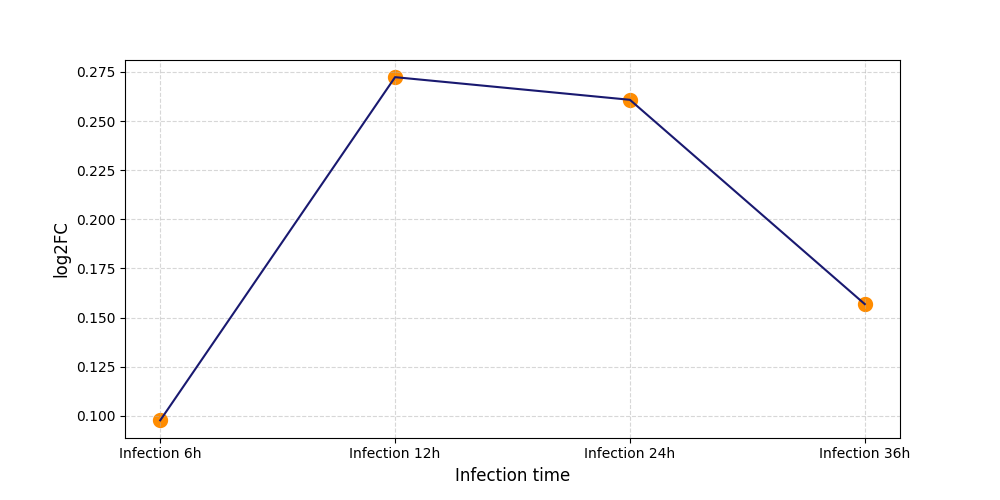 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1057
[4] |
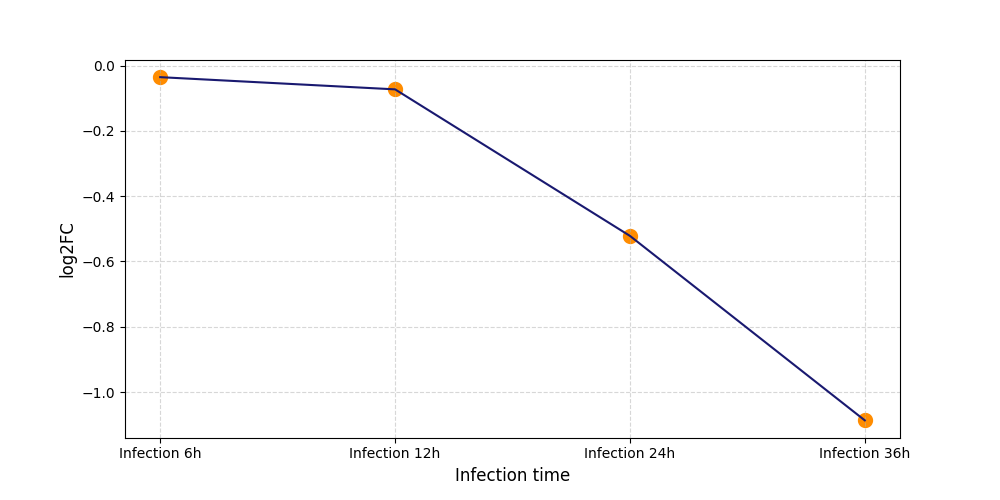 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1314
[4] |
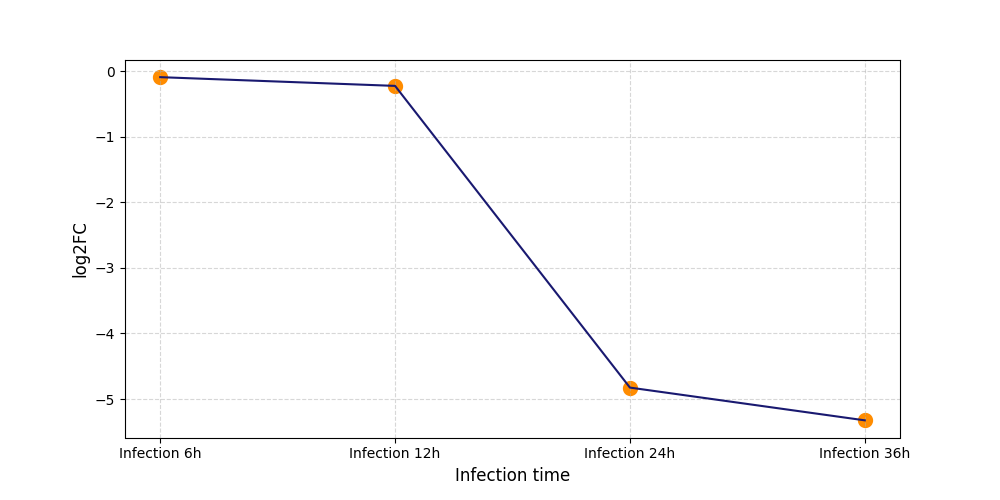 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1334
[4] |
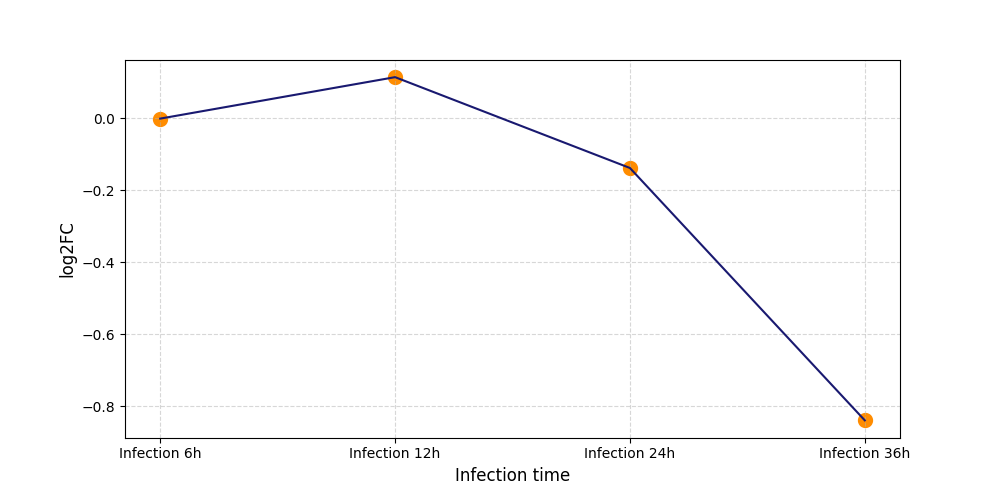 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1453
[4] |
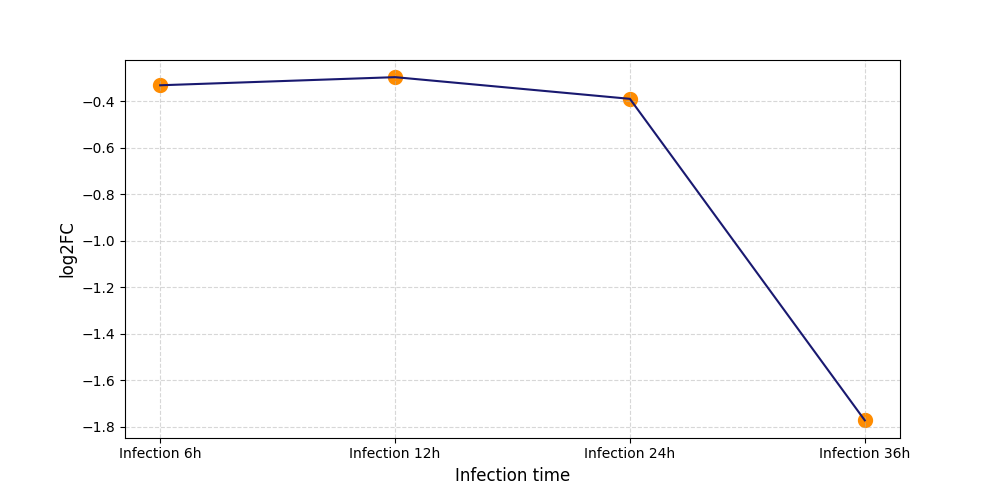 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1623
[4] |
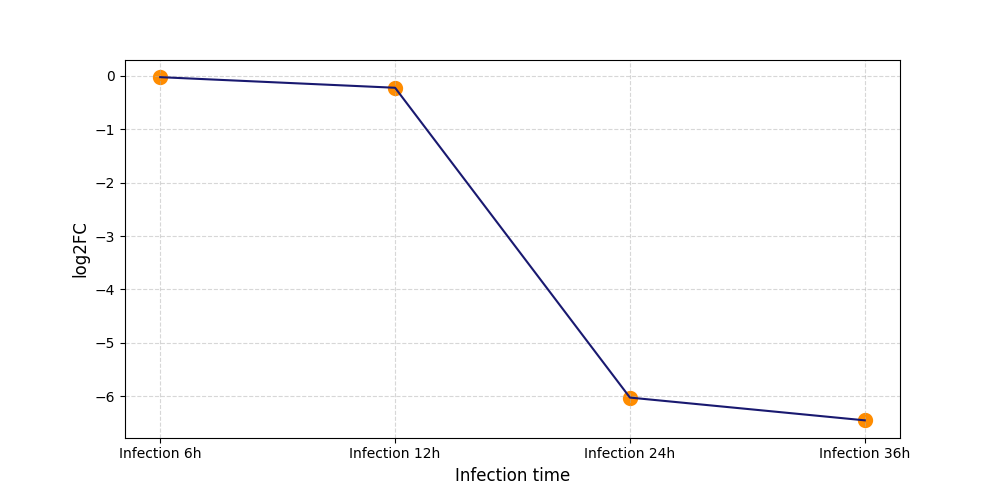 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1632
[4] |
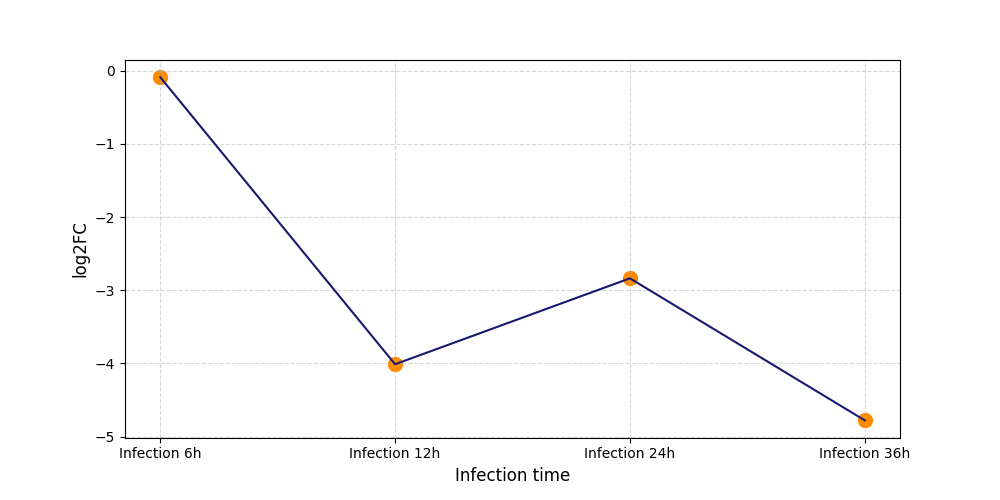 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1728
[4] |
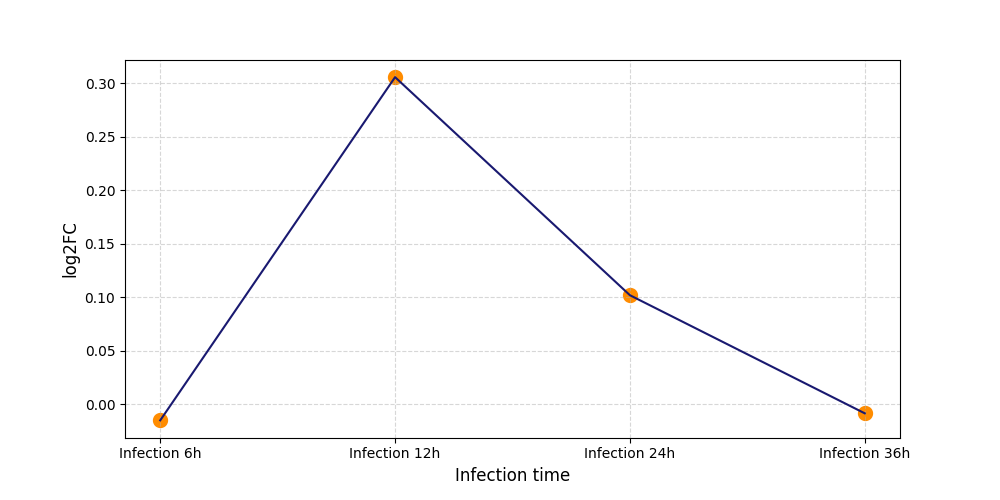 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1850
[4] |
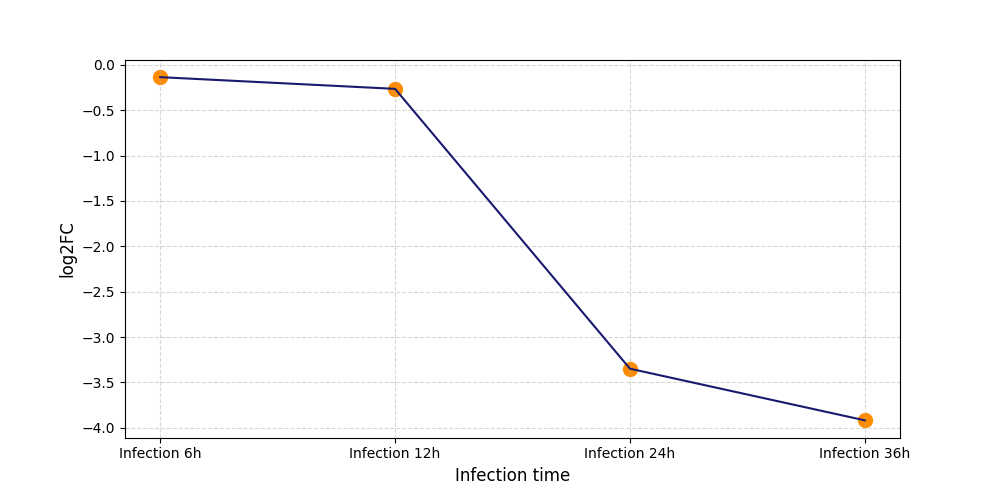 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S292
[4] |
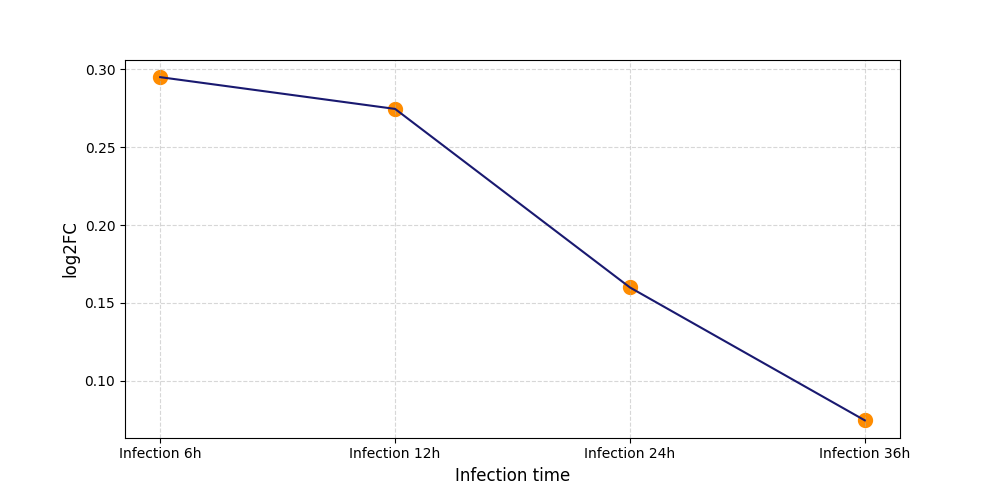 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S599
[4] |
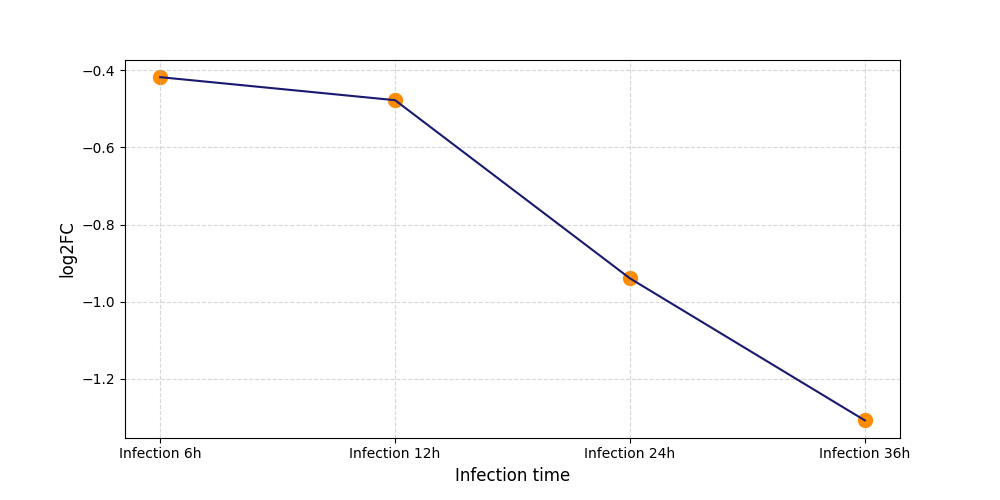 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S820
[4] |
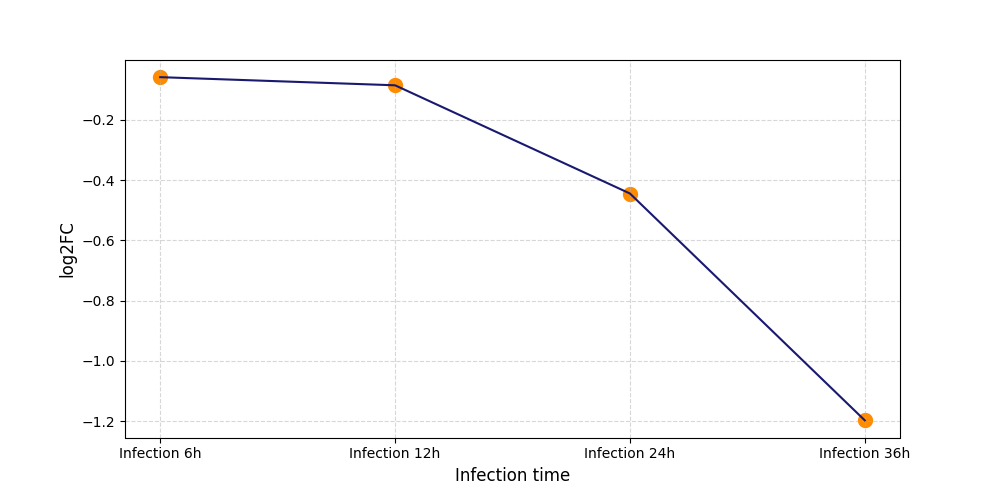 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S865
[4] |
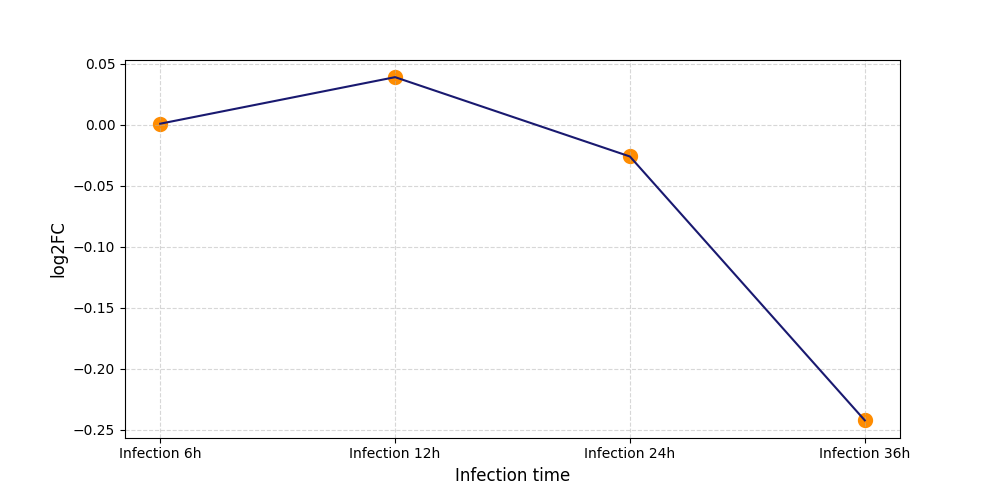 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S923
[4] |
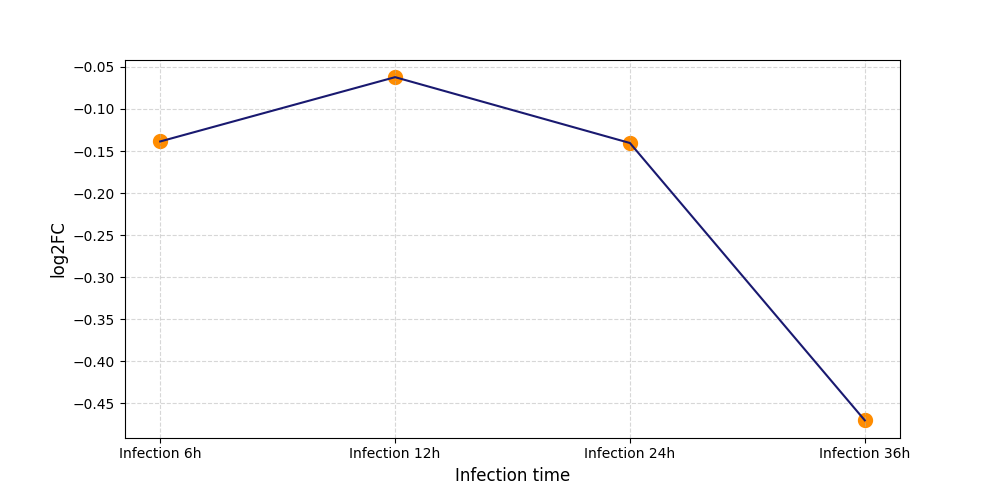 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S925
[4] |
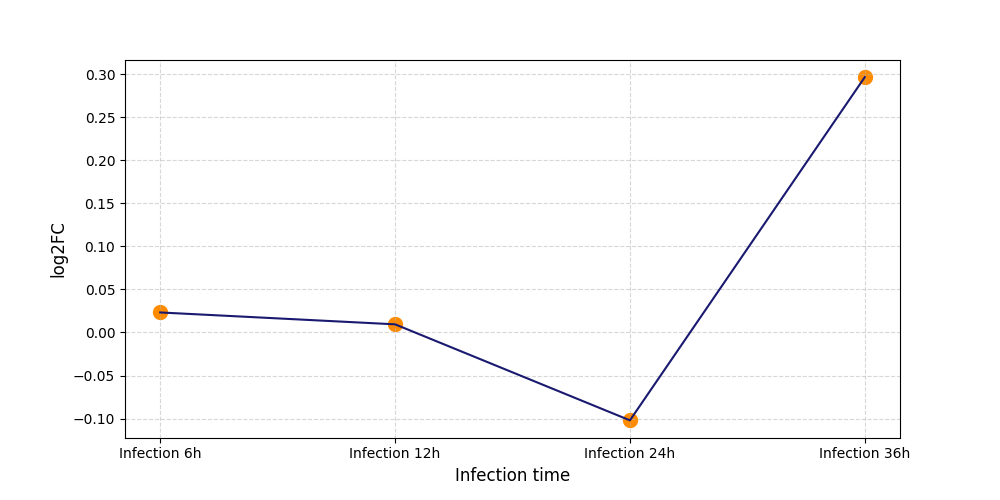 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S938
[4] |
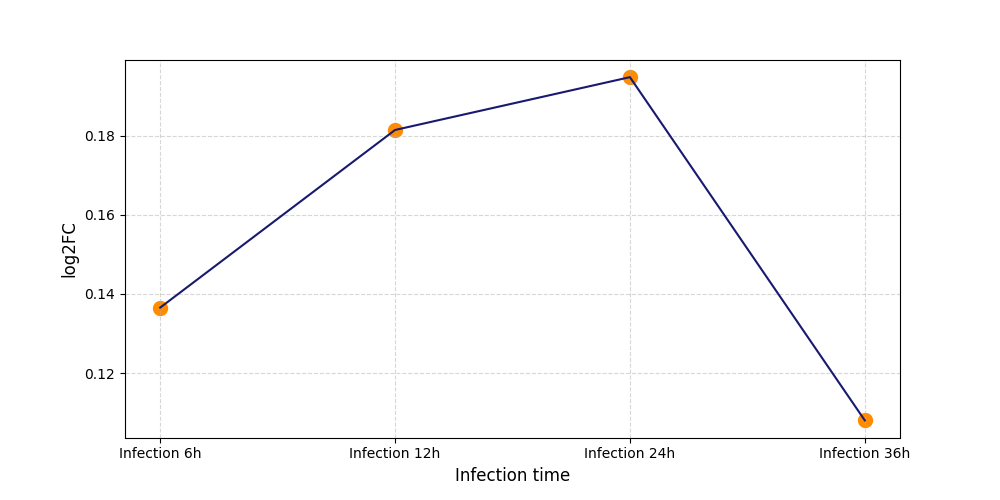 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
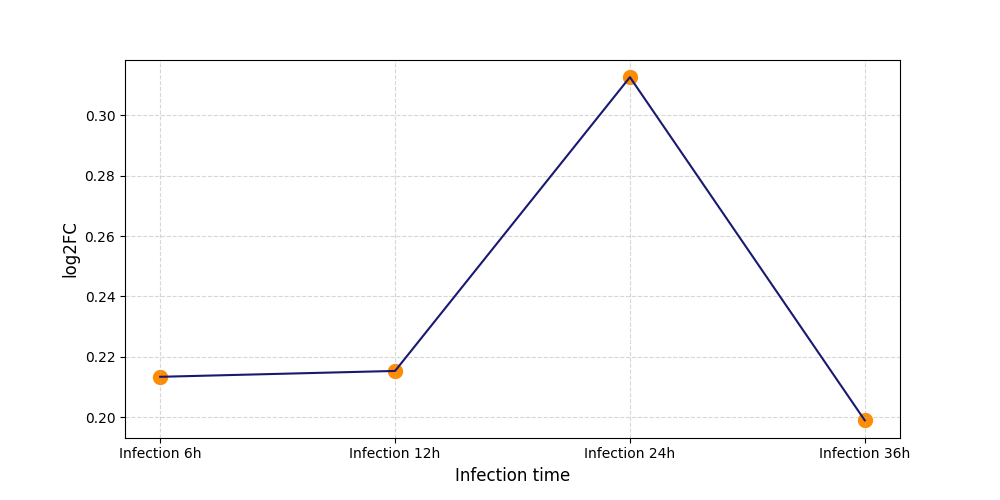
|
T1024
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
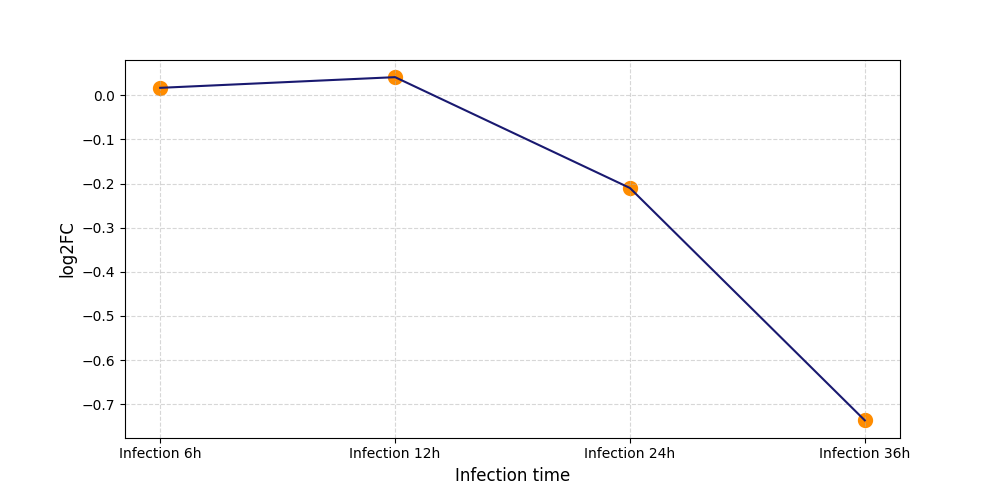
|
T1134
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
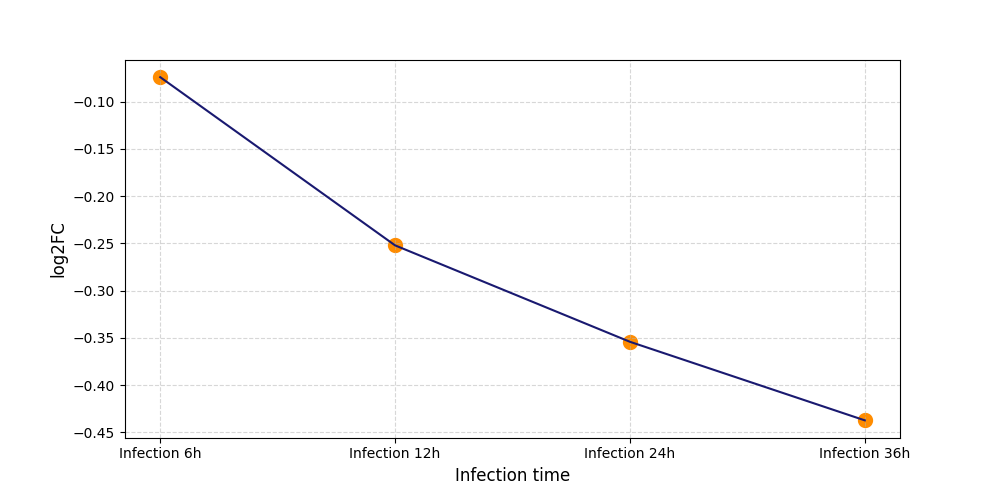
|
T1260
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
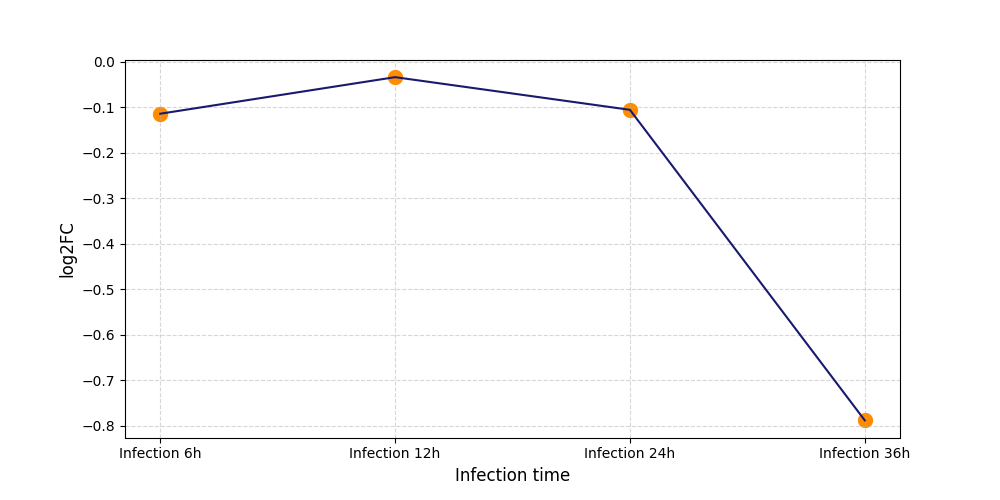
|
T1633
[4] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1848
[4] |
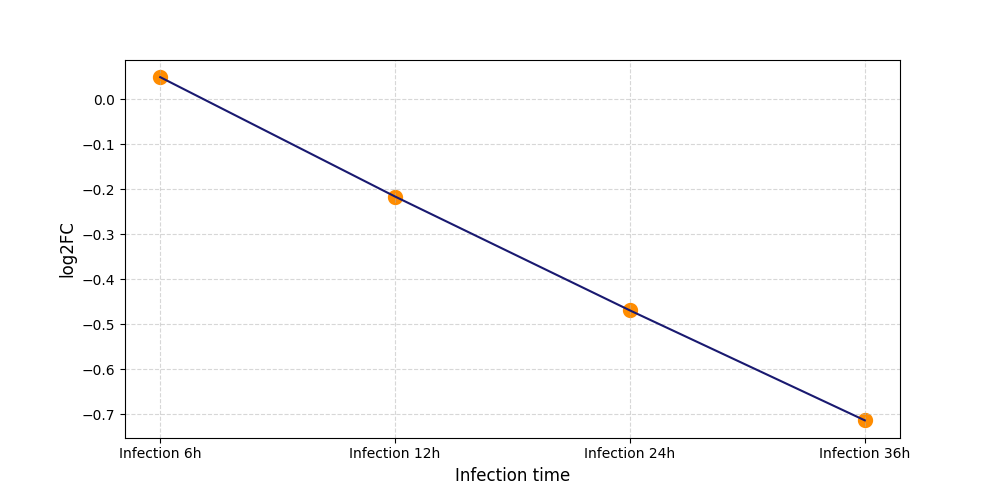 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T969
[4] |
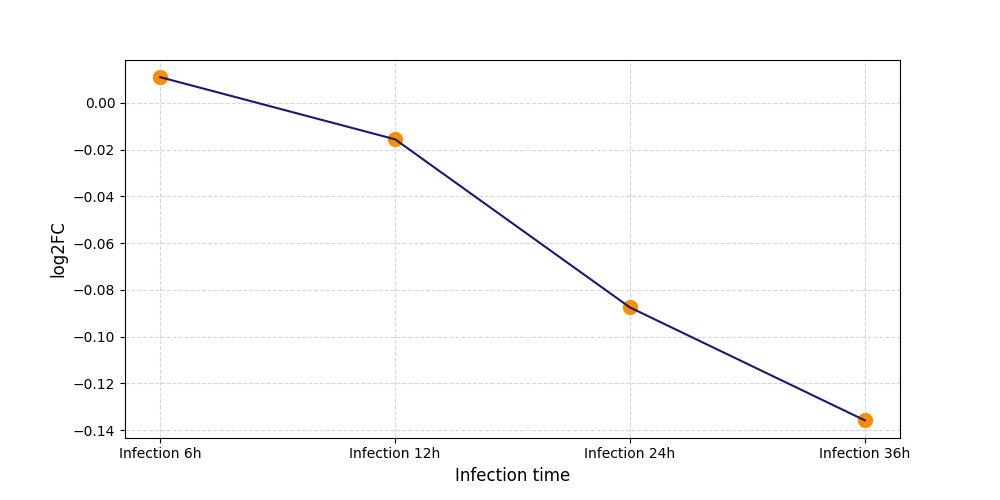 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Y1103
[4] |
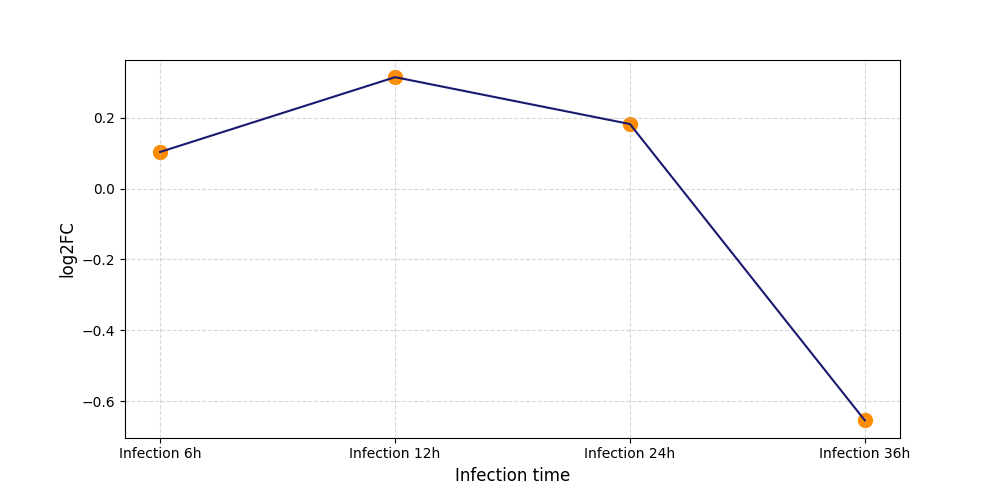 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Sequence Information |
MACCHKVMLLLDTAGGAARHSRVRRAALRLLTYLSCRFGLARVHWAFKFFDSQGARSRPSRVSDFRELGSRSWEDFEEELEARLEDRAHLPGPAPRATHTHGALMETLLDYQWDRPEITSPTKPILRSSGRRLLDVESEAKEAEAALGGLVNAVFLLAPCPHSQRELLQFVSGCEAQAQRLPPTPKQVMEKLLPKRVREVMVARKITFYWVDTTEWSKLWESPDHLGYWTVCELLHHGGGTVLPSESFSWDFAQAGEMLLRSGIKLSSEPHLSPWISMLPTDATLNRLLYNSPEYEASFPRMEGMLFLPVEAGKEIQETWTVTLEPLAMHQRHFQKPVRIFLKGSVAQWSLPTSSTLGTDSWMLGSPEESTATQRLLFQQLVSRLTAEELHLVADVDPGEGRPPITGVISPLSASAMILTVCRTKEAEFQRHVLQTAVADSPRDTASLFSDVVDSILNQTHDSLADTASAASPVPEWAQQELGHTTPWSPAVVEKWFPFCNISGASSDLMESFGLLQAASANKEESSKTEGELIHCLAELYQRKSREESTIAHQEDSKKKRGVPRTPVRQKMNTMCRSLKMLNVARLNVKAQKLHPDGSPDVAGEKGIQKIPSGRTVDKLEDRGRTLRSSKPKDFKTEEELLSYIRENYQKTVATGEIMLYACARNMISTVKMFLKSKGTKELEVNCLNQVKSSLLKTSKSLRQNLGKKLDKEDKVRECQLQVFLRLEMCLQCPSINESTDDMEQVVEEVTDLLRMVCLTEDSAYLAEFLEEILRLYIDSIPKTLGNLYNSLGFVIPQKLAGVLPTDFFSDDSMTQENKSPLLSVPFLSSARRSVSGSPESDELQELRTRSAKKRRKNALIRHKSIAEVSQNLRQIEIPKVSKRATKKENSHPAPQQPSQPVKDTVQEVTKVRRNLFNQELLSPSKRSLKRGLPRSHSVSAVDGLEDKLDNFKKNKGYHKLLTKSVAETPVHKQISKRLLHRQIKGRSSDPGPDIGVVEESPEKGDEISLRRSPRIKQLSFSRTHSASFYSVSQPKSRSVQRVHSFQQDKSDQRENSPVQSIRSPKSLLFGAMSEMISPSEKGSARMKKRSRNTLDSEVPAAYQTPKKSHQKSLSFSKTTPRRISHTPQTPLYTPERLQKSPAKMTPTKQAAFKESLKDSSSPGHDSPLDSKITPQKRHTQAGEGTSLETKTPRTPKRQGTQPPGFLPNCTWPHSVNSSPESPSCPAPPTSSTAQPRRECLTPIRDPLRTPPRAAAFMGTPQNQTHQQPHVLRAARAEEPAQKLKDKAIKTPKRPGNSTVTSSPPVTPKKLFTSPLCDVSKKSPFRKSKIECPSPGELDQKEPQMSPSVAASLSCPVPSTPPELSQRATLDTVPPPPPSKVGKRCRKTSDPRRSIVECQPDASATPGVGTADSPAAPTDSRDDQKGLSLSPQSPPERRGYPGPGLRSDWHASSPLLITSDTEHVTLLSEAEHHGIGDLKSNVLSVEEGEGLRTADAEKSSLSHPGIPPSPPSCGPGSPLMPSRDVHCTTDGRQCQASAQLDNLPASAWHSTDSASPQTYEVELEMQASGLPKLRIKKIDPSSSLEAEPLSKEESSLGEESFLPALSMPRASRSLSKPEPTYVSPPCPRLSHSTPGKSRGQTYICQACTPTHGPSSTPSPFQTDGVPWTPSPKHSGKTTPDIIKDWPRRKRAVGCGAGSSSGRGEVGADLPGSLSLLESEGKDHGLELSIHRTPILEDFELEGVCQLPDQSPPRNSMPKAEEASSWGQFGLSSRKRVLLAKEEADRGAKRICDLREDSEVSKSKEGSPSWSAWQLPSTGDEEVFVSGSTPPPSCAVRSCLSASALQALTQSPLLFQGKTPSSQSKDPRDEDVDVLPSTVEDSPFSRAFSRRRPISRTYTRKKLMGTWLEDL
Click to Show/Hide
|
|---|