Details of Host Protein
| Host Protein General Information (ID: PT0446) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name |
Filamin A (FLNA)
|
Gene Name |
FLNA
|
||||||
| Host Species |
Homo sapiens
|
Uniprot Entry Name |
FLNA_HUMAN
|
||||||
| Protein Families |
Filamin family
|
||||||||
| Subcellular Location |
Cytoplasm; cell cortex Cytoplasm
|
||||||||
| External Link | |||||||||
| NCBI Gene ID | |||||||||
| Uniprot ID | |||||||||
| Ensembl ID | |||||||||
| HGNC ID | |||||||||
| Function in Host |
Promotes orthogonal branching of actin filaments and linksactin filaments to membrane glycoproteins. Anchors varioustransmembrane proteins to the actin cytoskeleton and serves as ascaffold for a wide range of cytoplasmic signaling proteins. Interaction with FLNB may allow neuroblast migration from theventricular zone into the cortical plate. Tethers cell surface-localized furin, modulates its rate of internalization and directs itsintracellular trafficking. Involved in ciliogenesis. Plays a role in cell-cell contacts and adherens junctions during thedevelopment of blood vessels, heart and brain organs. Plays a role inplatelets morphology through interaction with SYK that regulatesITAM- and ITAM-like-containing receptor signaling, resulting in byplatelet cytoskeleton organization maintenance. Duringthe axon guidance process, required for growth cone collapse induced bySEMA3A-mediated stimulation of neurons.
[1-2]
Click to Show/Hide
|
||||||||
| Related KEGG Pathway | |||||||||
| Salmonella infection | hsa05132 |
Pathway Map 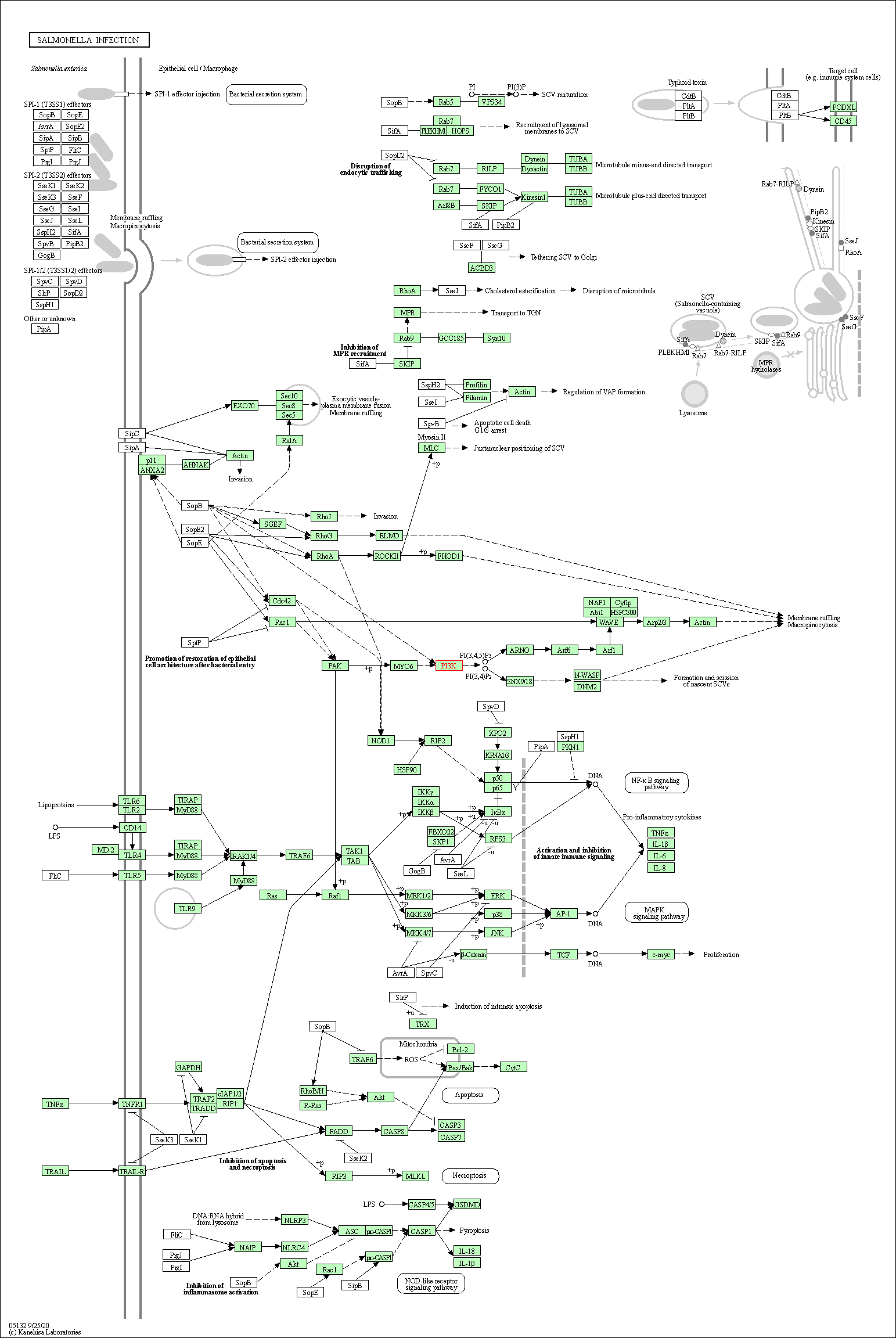
|
|||||||
| MAPK signaling pathway | hsa04010 |
Pathway Map 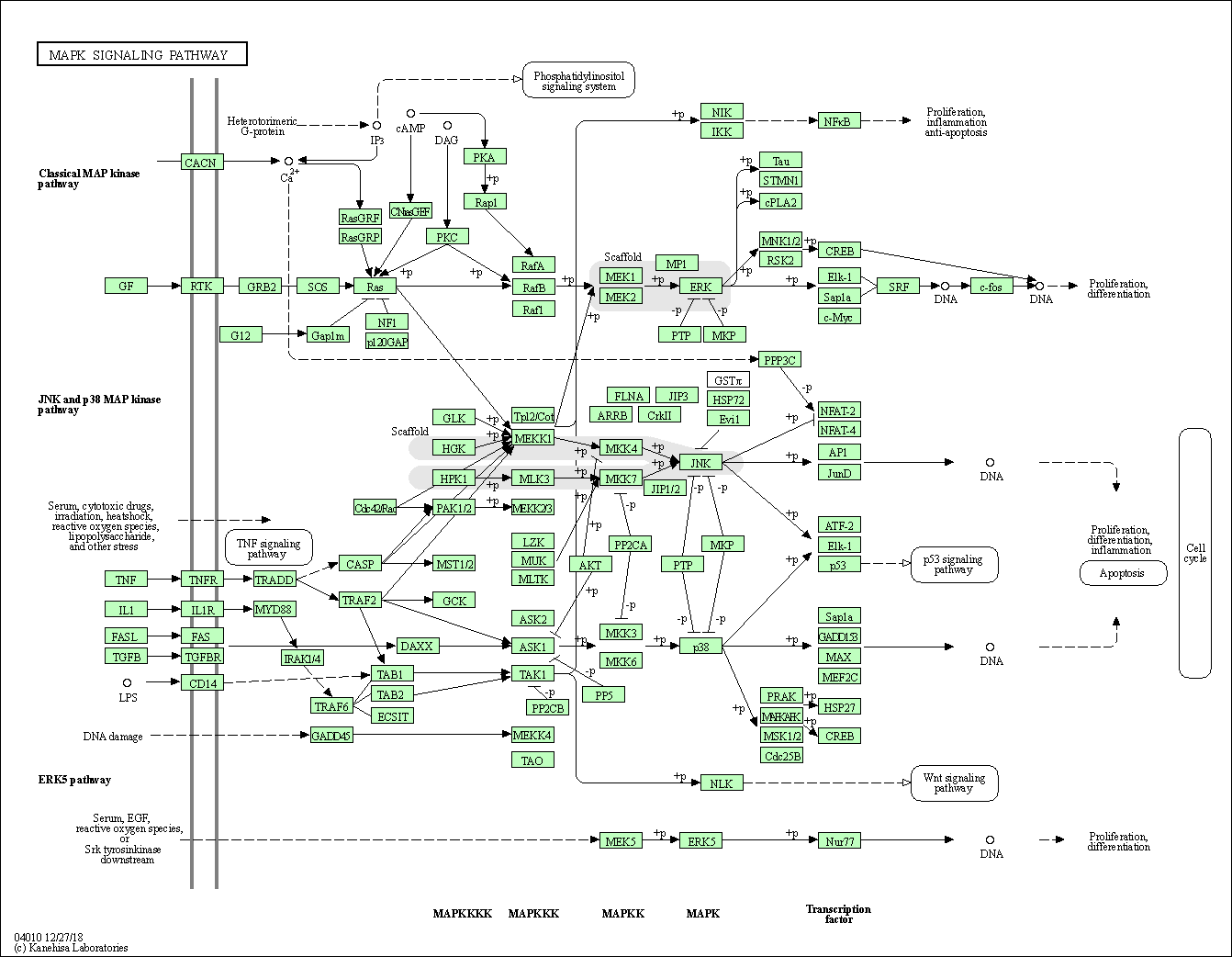
|
|||||||
| Focal adhesion | hsa04510 |
Pathway Map 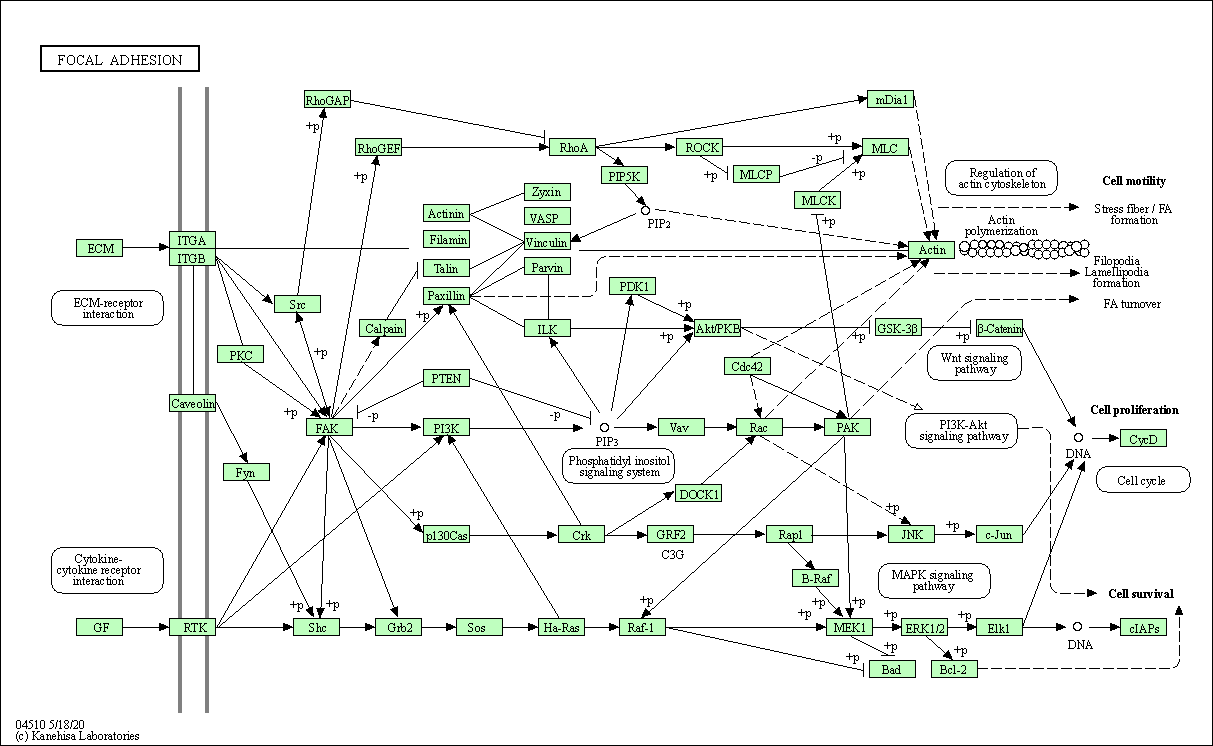
|
|||||||
| Proteoglycans in cancer | hsa05205 |
Pathway Map 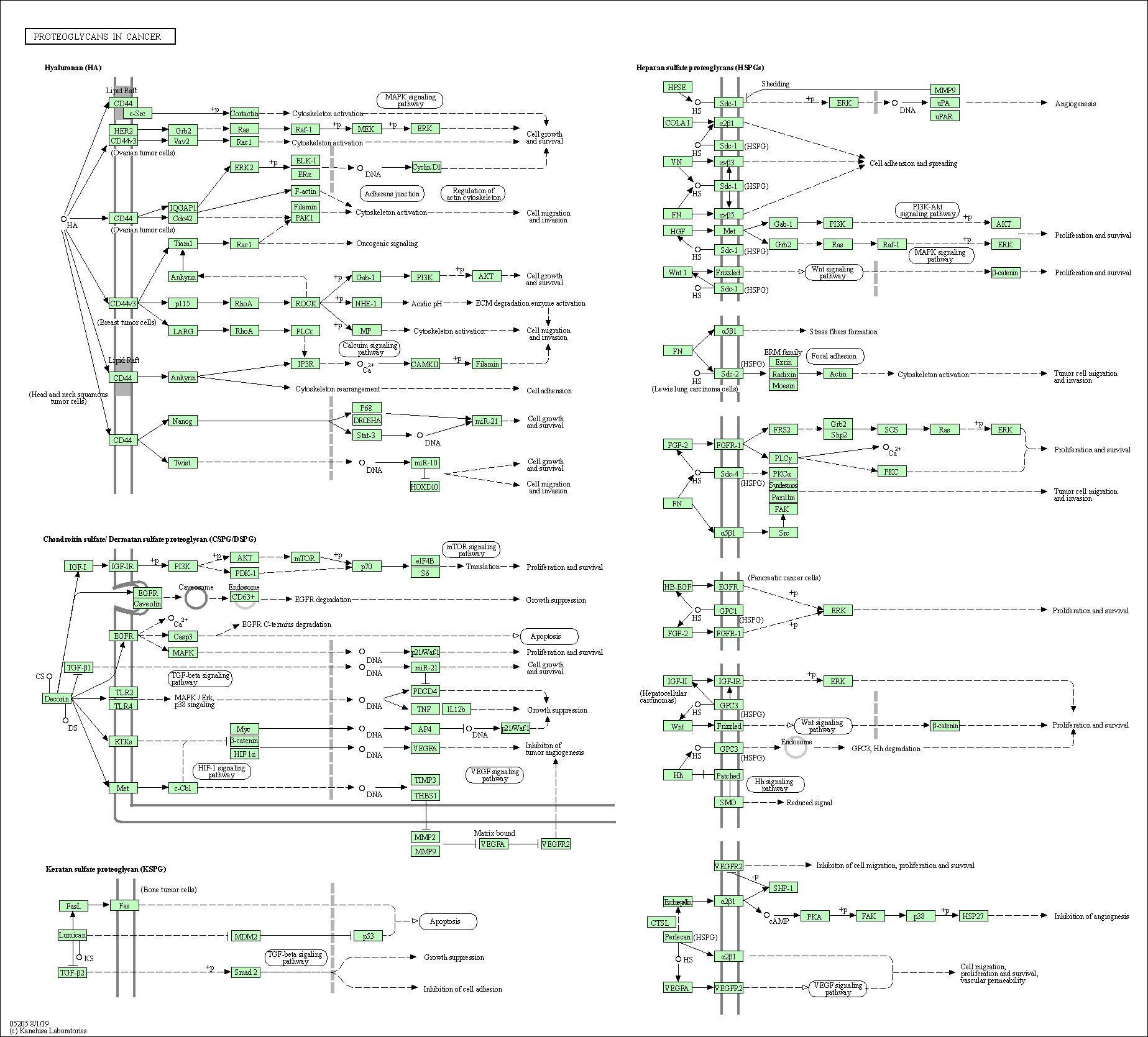
|
|||||||
| 3D Structure |
|
||||||||
| Function of This Protein During Virus Infection | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Virus Name | SARS-COV-2 | Protein Function | Anti-viral | [3] | |||||
| Infected Tissue | Lung | Infection Time | 7-9 Days | ||||||
| Infected Cell | Calu-3 Cells (Human epithelial cell line) | Cellosaurus ID | CVCL_0609 | ||||||
| Method Description | To detect the role of host protein FLNA in viral infection, FLNA protein knockout Calu-3 Cells were infected with SARS-COV-2 for 7 - 9 Days , and the effects on infection was detected through CRISPR-based genome-wide gene-knockout screen. | ||||||||
| Results | It is reported that knockout of FLNA increases SARS-CoV-2 RNA levels compared with control group. | ||||||||
| Host Protein - Virus RNA Network | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Full List of Virus RNA Interacting with This Protien | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| RNA Region: Not Specified Virus Region (hCoV-19/Not Specified Virus Strain ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[4] | |||||||
| Strains Name |
hCoV-19/Not Specified Virus Strain
|
||||||||
| RNA Binding Region |
Not Specified Virus Region
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Infection Cells | Huh7 cells (liver carcinoma cell) (CVCL_0336 ) | ||||||||
| Cell Originated Tissue | Liver | ||||||||
| Infection Time | 24h | ||||||||
| Interaction Score | log2FC = 3.01729E+14 | ||||||||
| Method Description | RNA antisense purification and quantitative mass spectrometry (RAP-MS); Tandem mass tag (TMT) labelling; liquid chromatography tandem mass spectrometry (LC-MS/MS); Westernblot | ||||||||
| RNA Region: Not Specified Virus Region (hCoV-19/France/IDF-220-95/2020 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[5] | |||||||
| Strains Name |
hCoV-19/France/IDF-220-95/2020
|
||||||||
| RNA Binding Region |
Not Specified Virus Region
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Interaction Type | Direct interaction | ||||||||
| Infection Cells | HEK293 Cells (Human embryonic kidney cell) HEK293 Cells (Human embryonic kidney cell) (CVCL_0045 ) | ||||||||
| Cell Originated Tissue | Kidney | ||||||||
| Infection Time | 48 h | ||||||||
| Interaction Score | SAINT score ≥ 0.79 | ||||||||
| Method Description | comprehensive identification of RNA-binding proteins by massspectrometry (ChIRP-MS) | ||||||||
| RNA Region: Not Specified Virus Region (hCoV-19/England/02/2020 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[6] | |||||||
| Strains Name |
hCoV-19/England/02/2020
|
||||||||
| Strains Family |
Beta (B.1.351)
|
||||||||
| RNA Binding Region |
Not Specified Virus Region
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Infection Cells | Calu-3 cells (Human lung cancer cell) Calu-3 cells (Human lung cancer cell) (CVCL_0609 ) | ||||||||
| Cell Originated Tissue | Lung | ||||||||
| Infection Time | 24 h | ||||||||
| Interaction Score | P-adjust = 0.008 | ||||||||
| Method Description | UV protein-RNA crosslinking; RNA interactome capture (cRIC); RNA antisense purification coupled with mass spectrometry (RAP-MS) | ||||||||
| Differential Gene Expression During SARS-COV-2 Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Phosphorylation after Virus Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
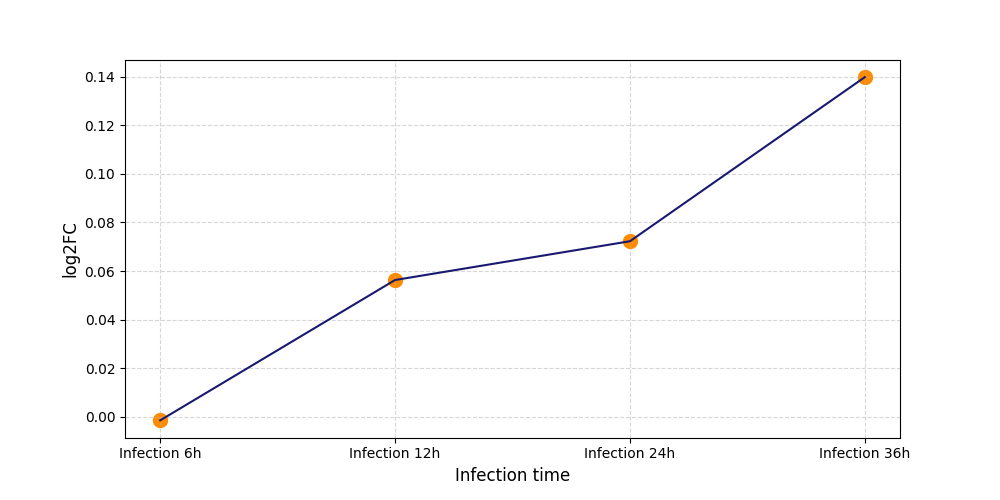
S1084
[7] |
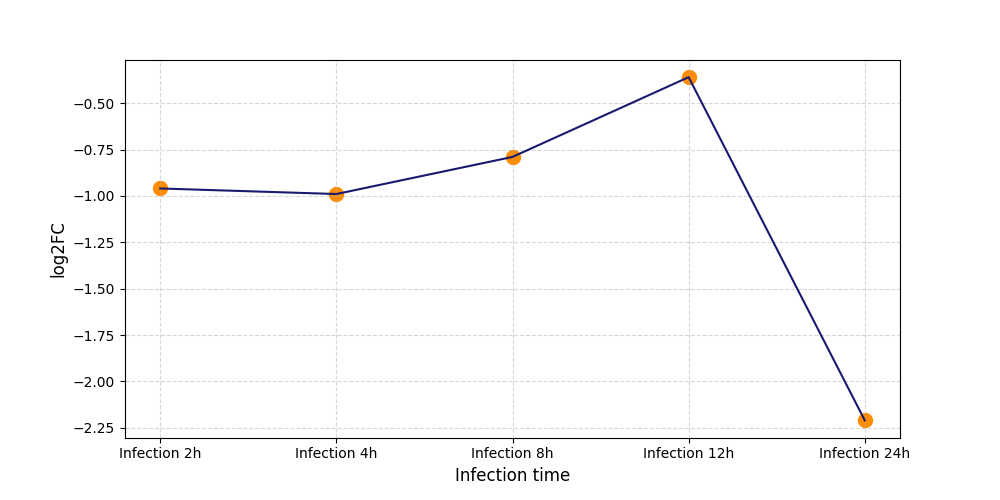 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
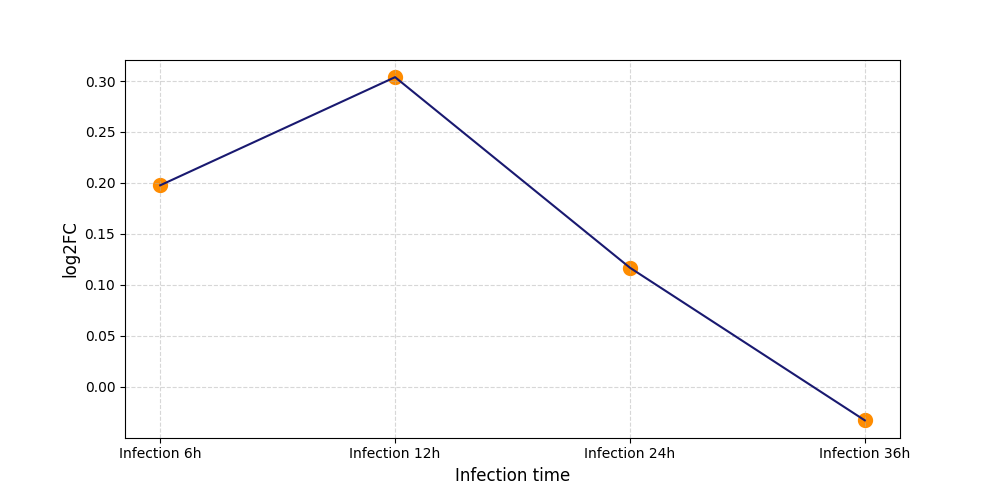
|
S1084
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
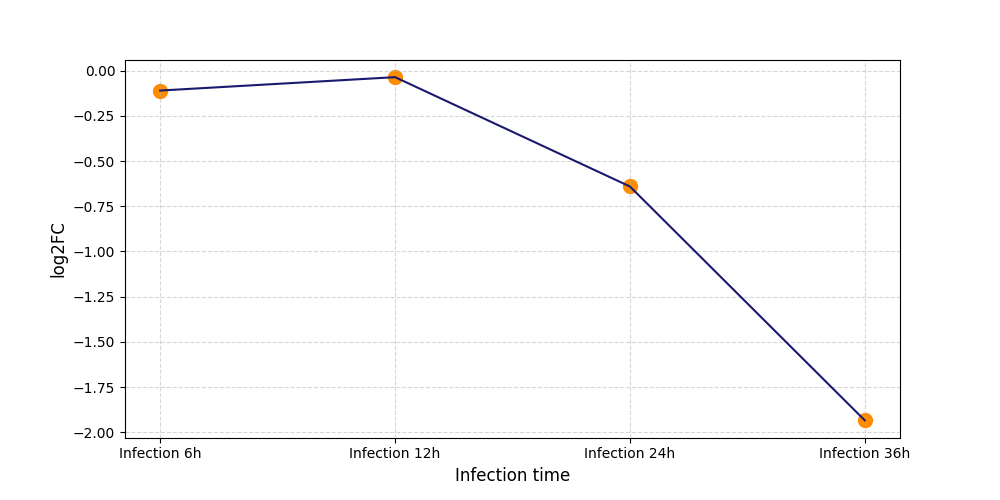
|
S1459
[7] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
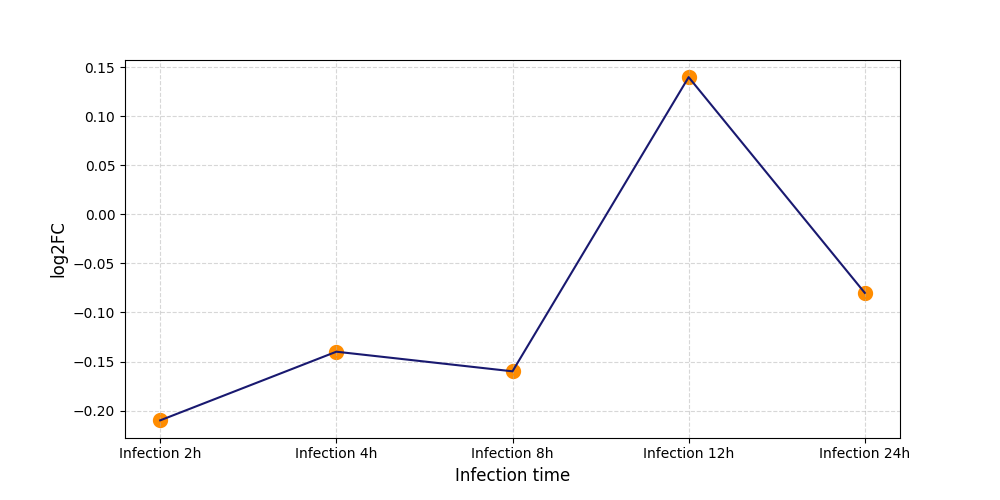
|
S1459
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1533
[8] |
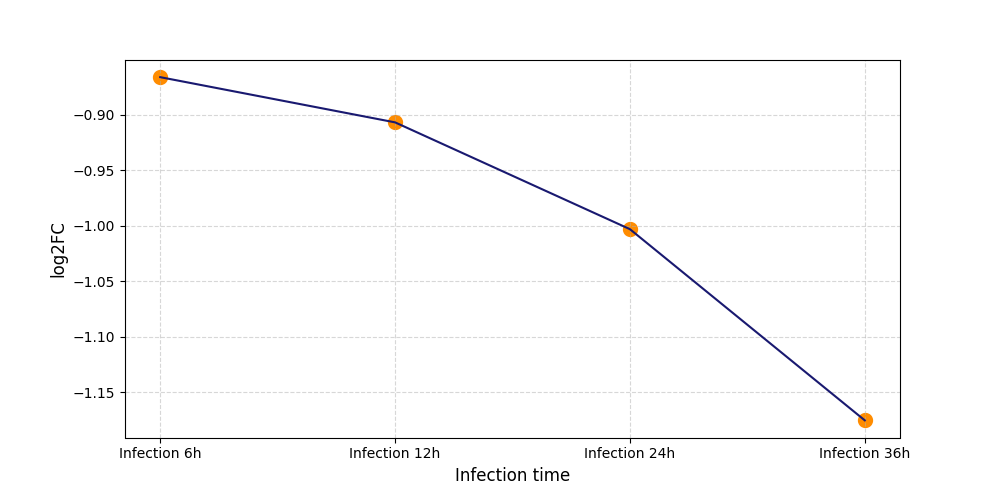 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1630
[7] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1630
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S1734
[8] |
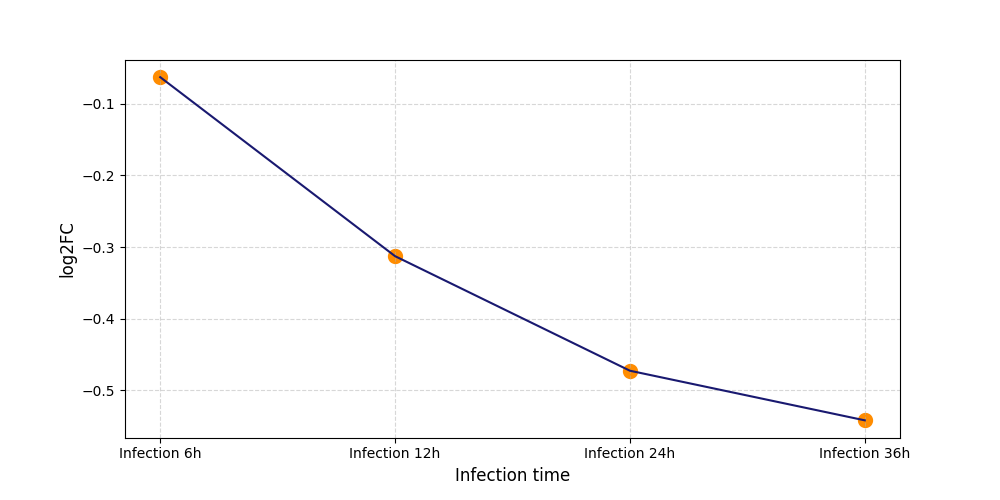 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2053
[8] |
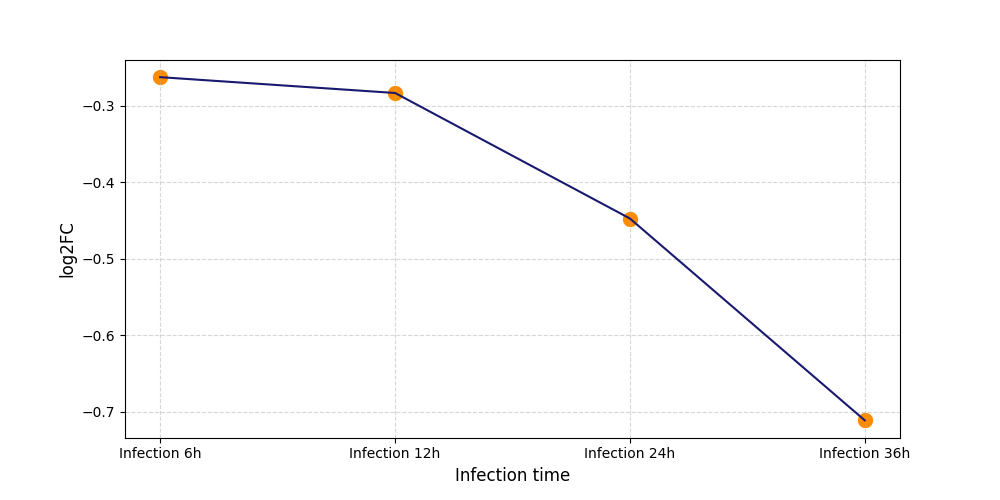 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2128
[7] |
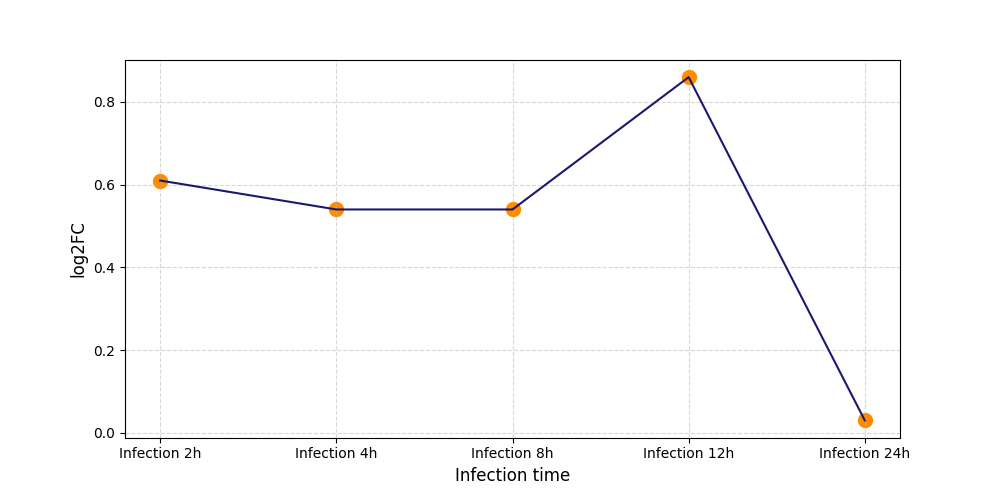 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2128
[8] |
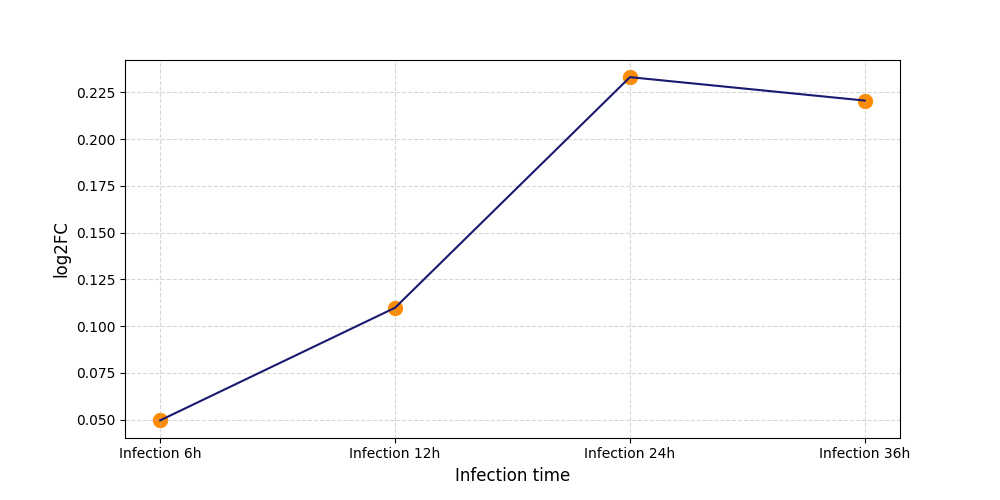 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2131
[8] |
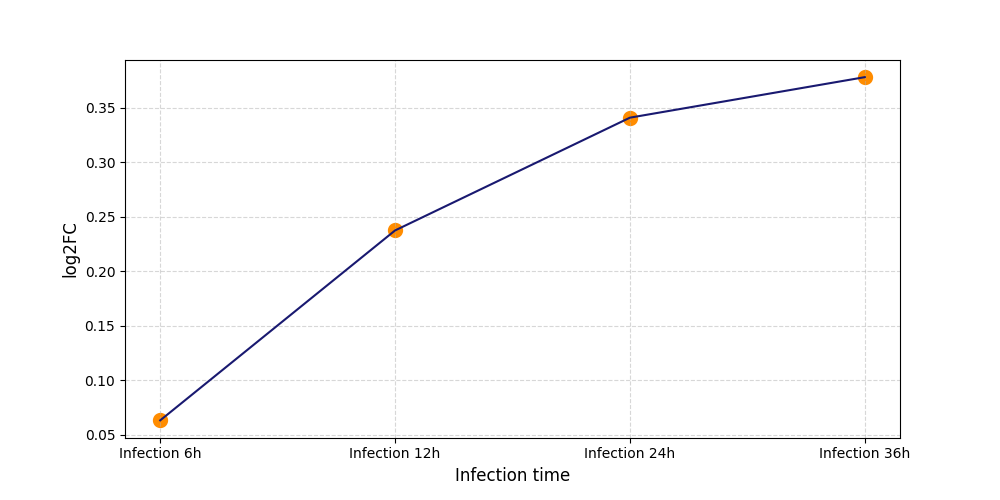 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2152
[8] |
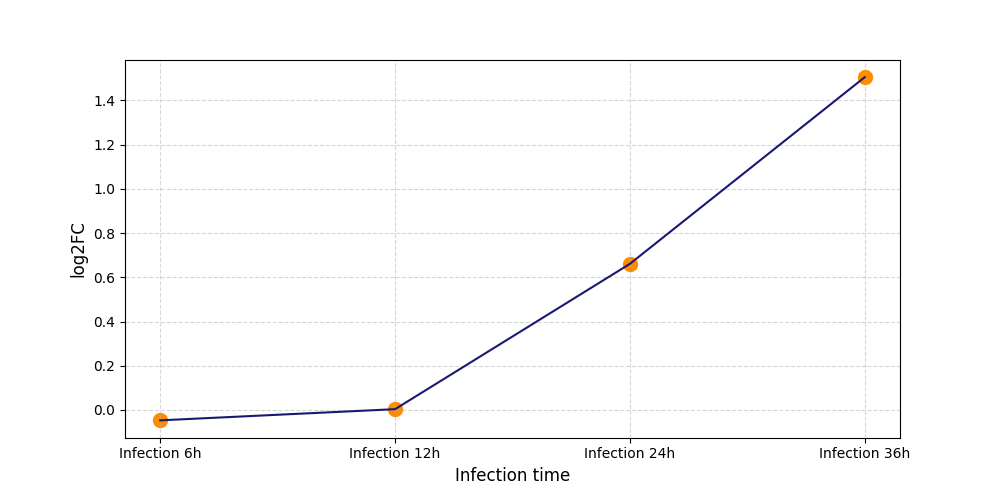 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2158
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2180
[8] |
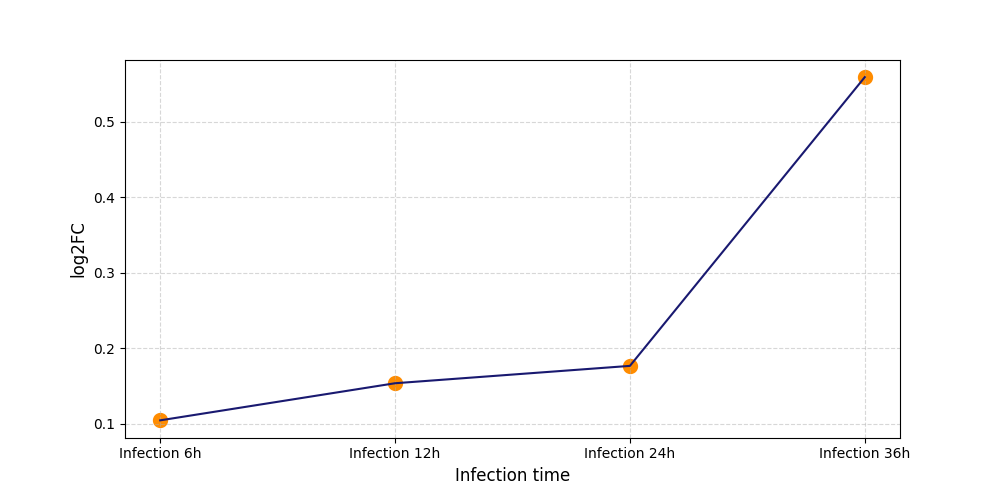 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2182
[8] |
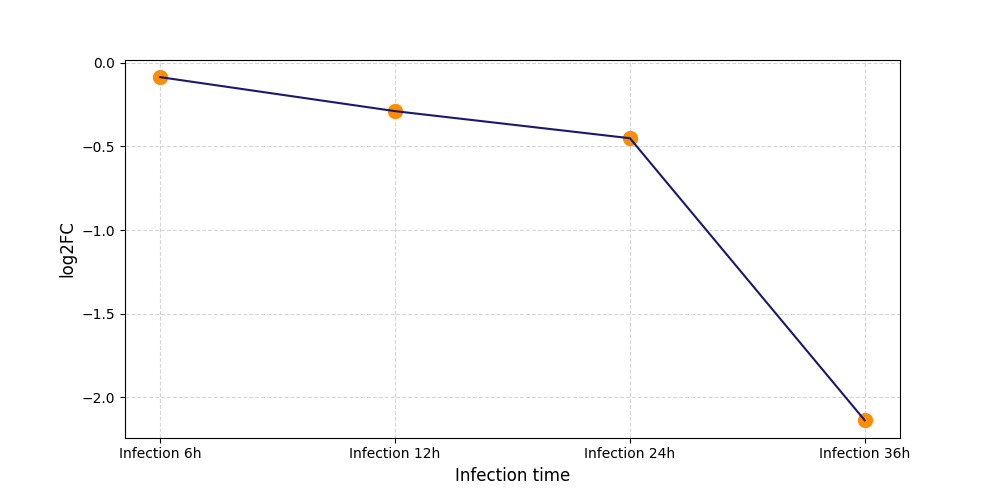 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
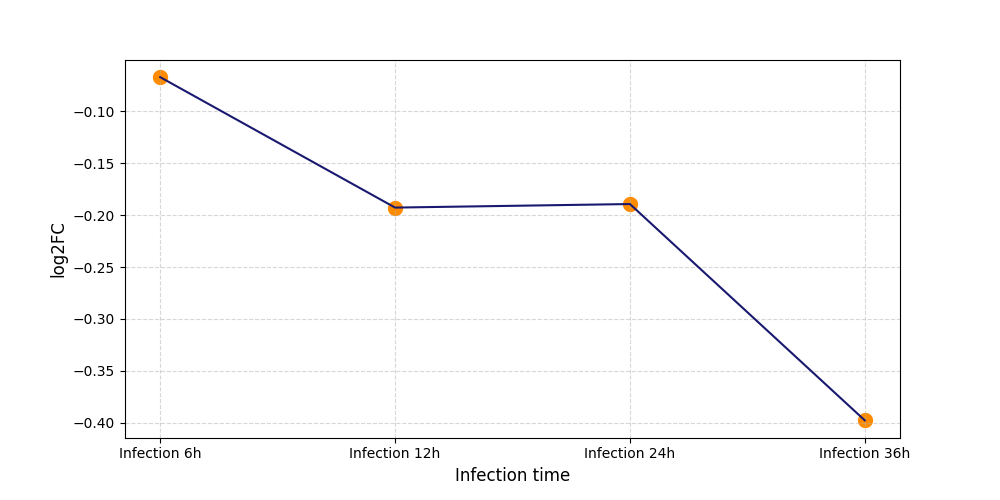
|
S2284
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
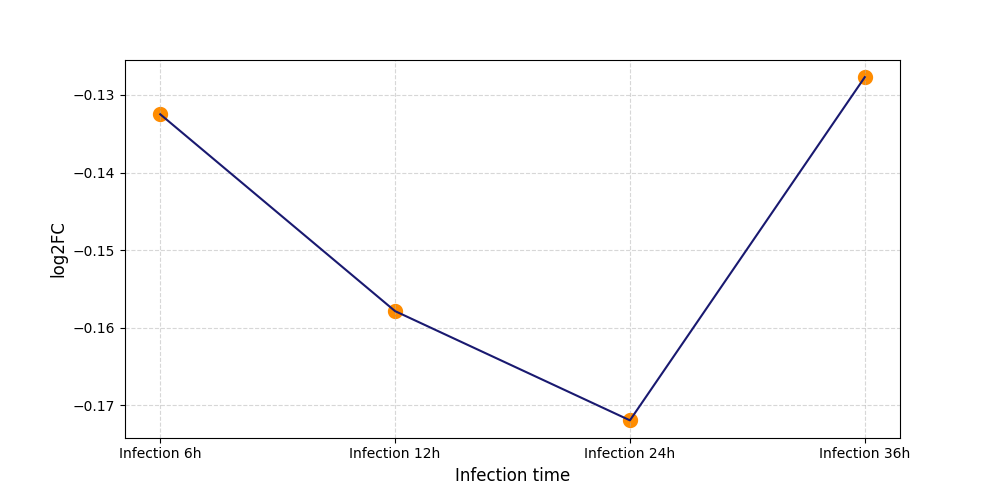
|
S2327
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2338
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
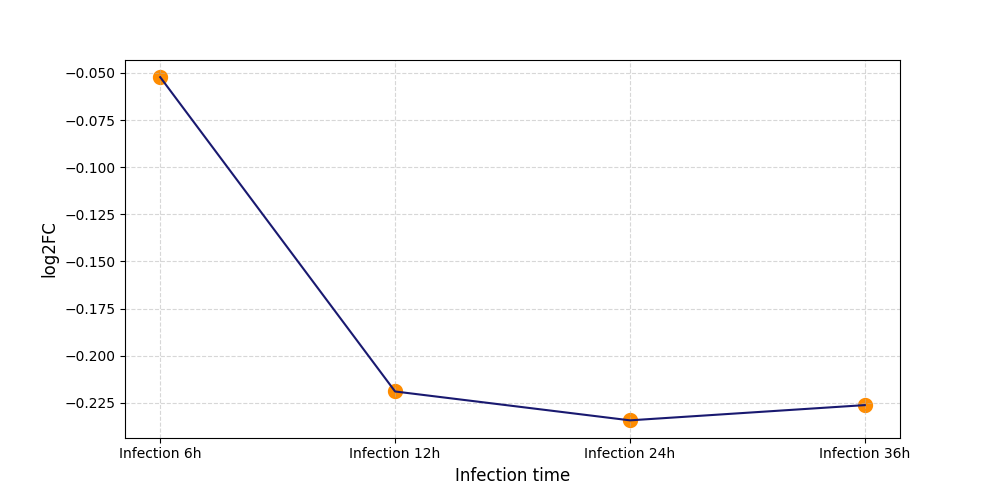
|
S2414
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
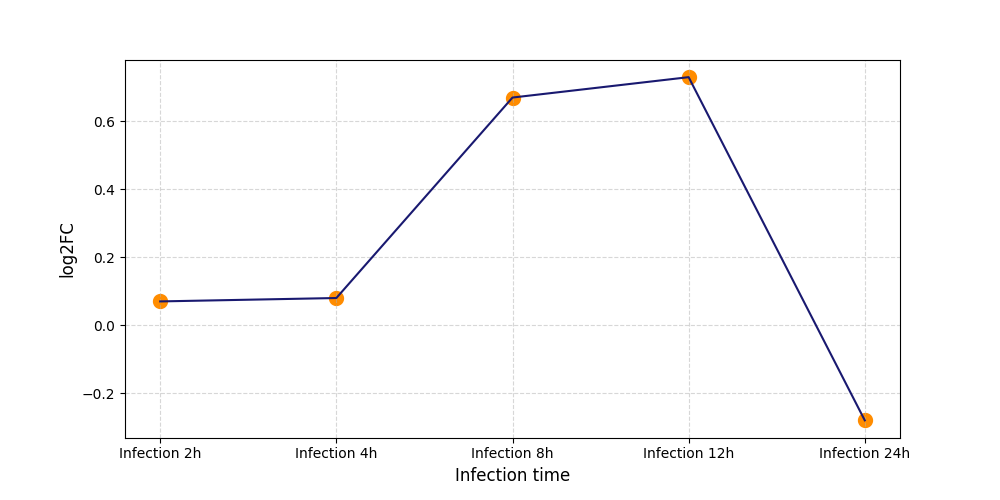
|
S2510
[7] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S2510
[8] |
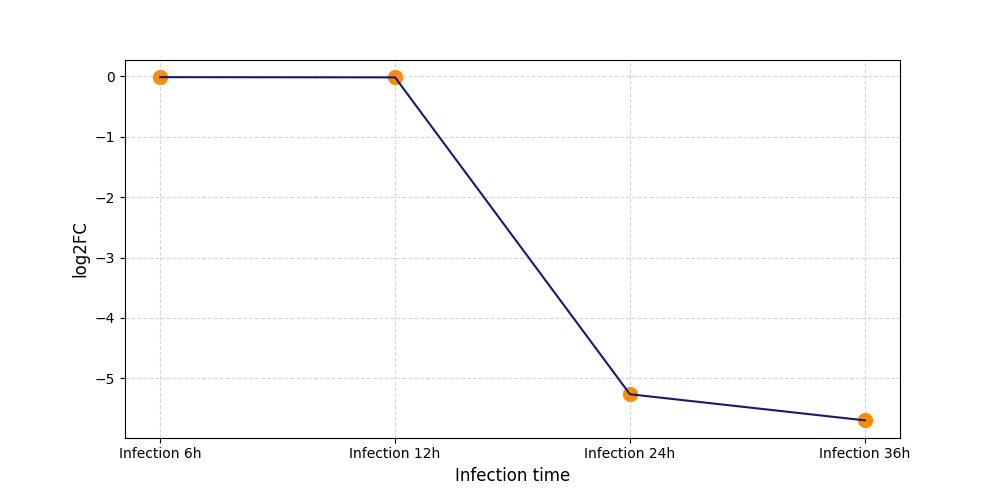 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S860
[8] |
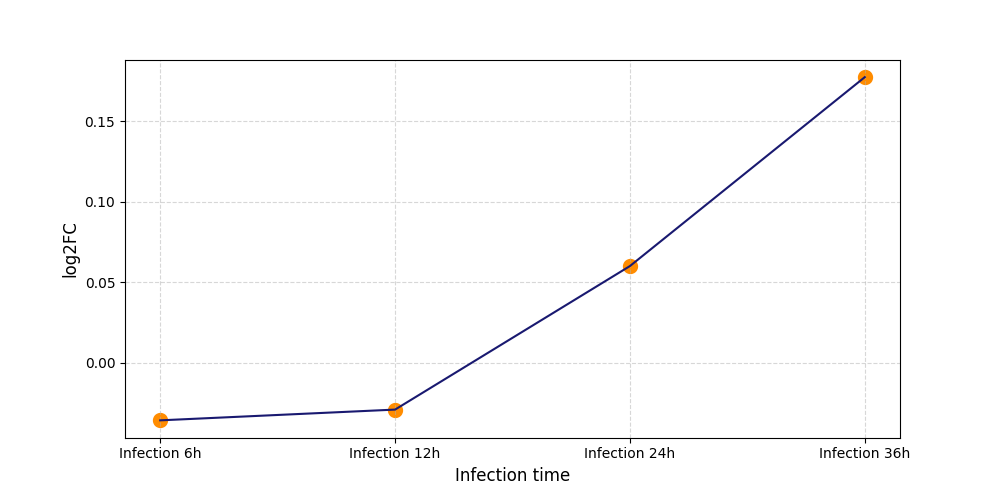 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S968
[8] |
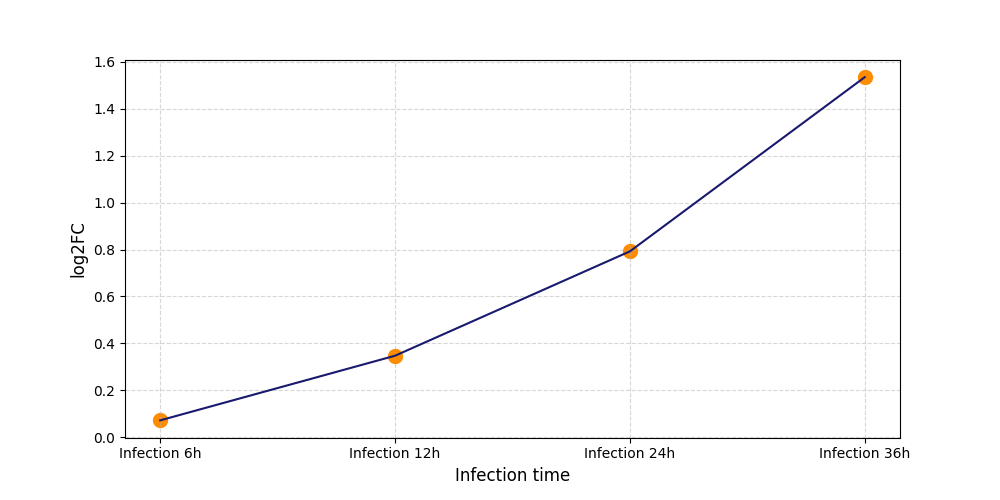 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S972
[8] |
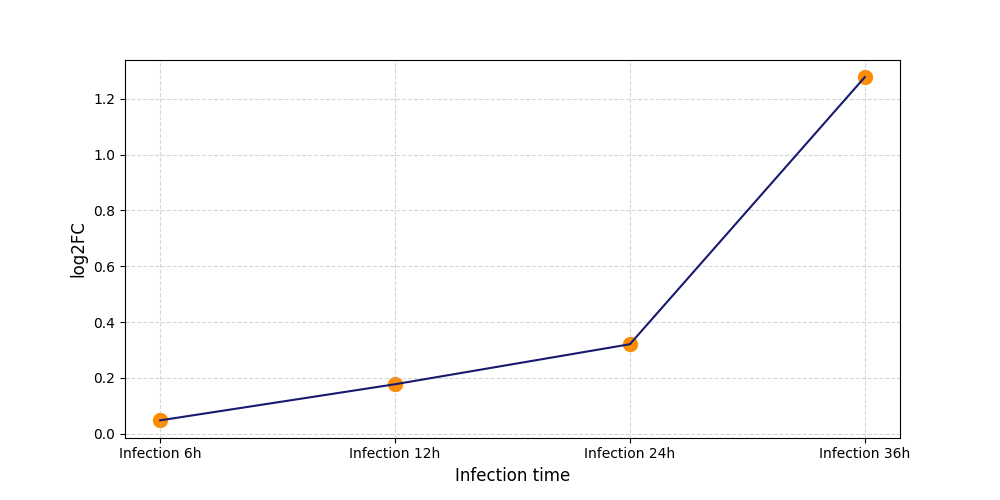 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T1739
[8] |
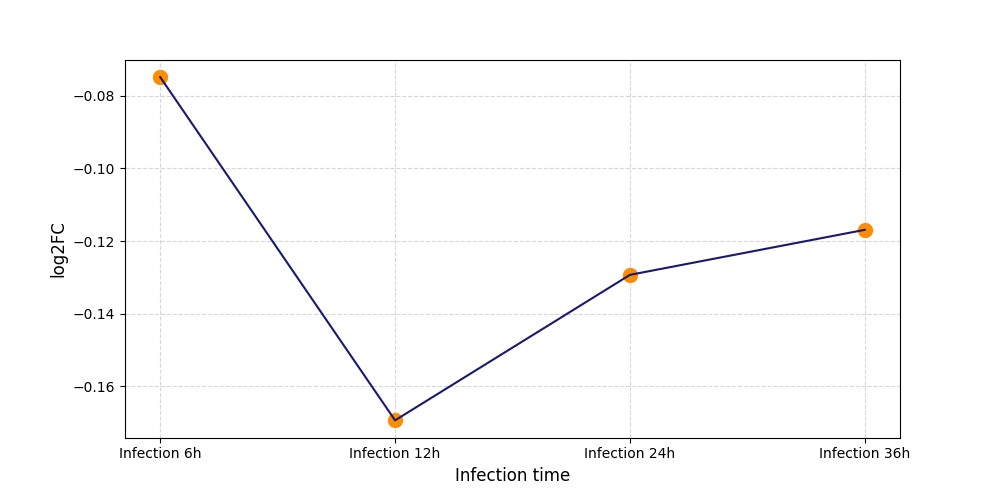 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2062
[8] |
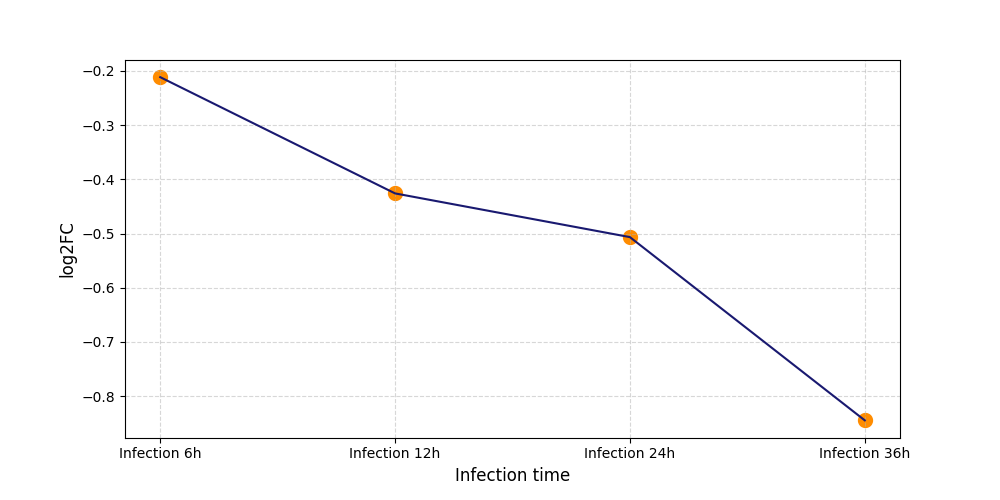 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2336
[8] |
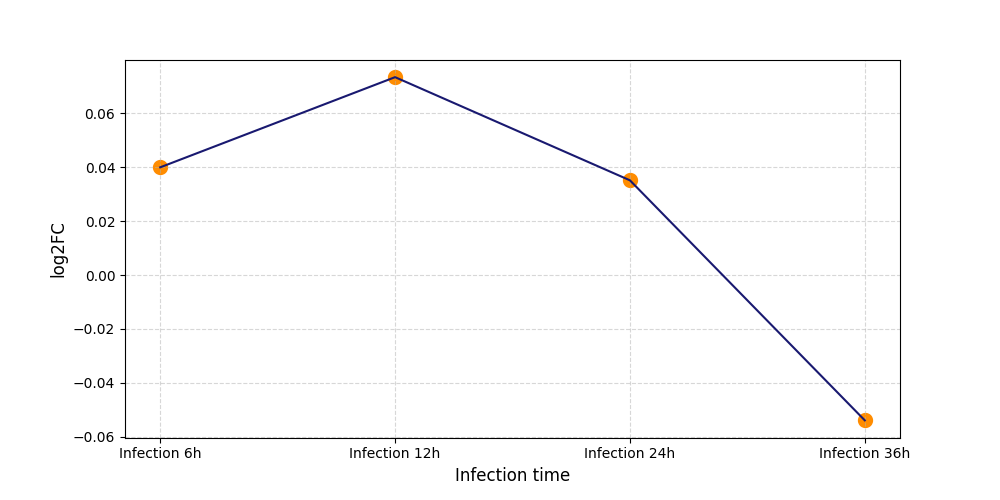 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2599
[7] |
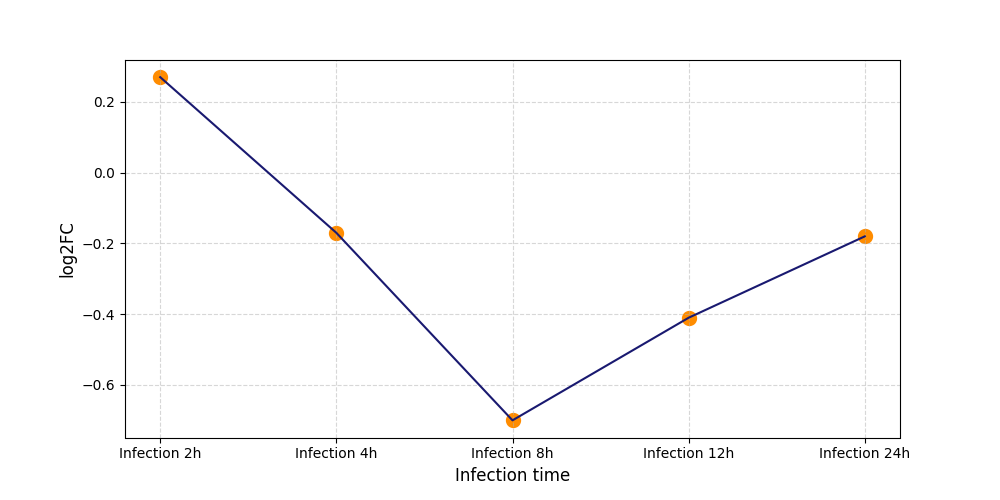 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T2599
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
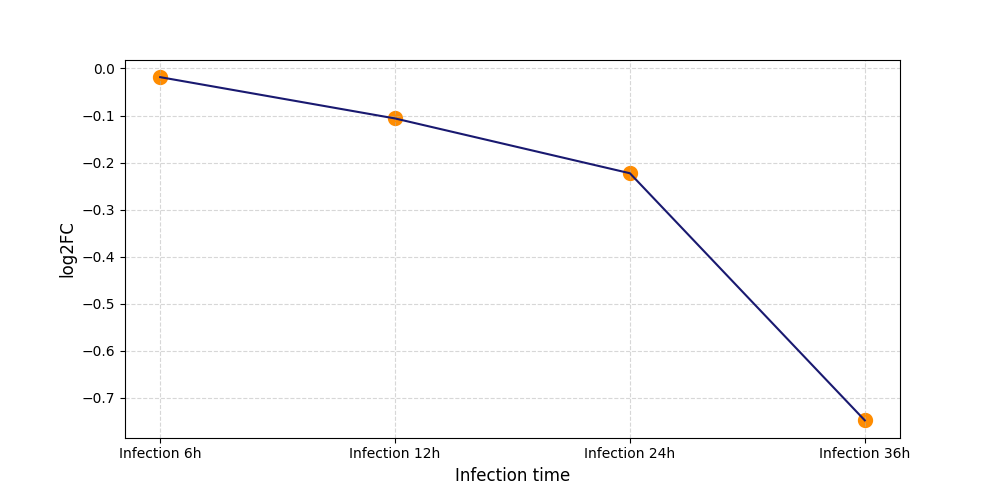
T859
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Y1525
[8] |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Sequence Information |
MSSSHSRAGQSAAGAAPGGGVDTRDAEMPATEKDLAEDAPWKKIQQNTFTRWCNEHLKCVSKRIANLQTDLSDGLRLIALLEVLSQKKMHRKHNQRPTFRQMQLENVSVALEFLDRESIKLVSIDSKAIVDGNLKLILGLIWTLILHYSISMPMWDEEEDEEAKKQTPKQRLLGWIQNKLPQLPITNFSRDWQSGRALGALVDSCAPGLCPDWDSWDASKPVTNAREAMQQADDWLGIPQVITPEEIVDPNVDEHSVMTYLSQFPKAKLKPGAPLRPKLNPKKARAYGPGIEPTGNMVKKRAEFTVETRSAGQGEVLVYVEDPAGHQEEAKVTANNDKNRTFSVWYVPEVTGTHKVTVLFAGQHIAKSPFEVYVDKSQGDASKVTAQGPGLEPSGNIANKTTYFEIFTAGAGTGEVEVVIQDPMGQKGTVEPQLEARGDSTYRCSYQPTMEGVHTVHVTFAGVPIPRSPYTVTVGQACNPSACRAVGRGLQPKGVRVKETADFKVYTKGAGSGELKVTVKGPKGEERVKQKDLGDGVYGFEYYPMVPGTYIVTITWGGQNIGRSPFEVKVGTECGNQKVRAWGPGLEGGVVGKSADFVVEAIGDDVGTLGFSVEGPSQAKIECDDKGDGSCDVRYWPQEAGEYAVHVLCNSEDIRLSPFMADIRDAPQDFHPDRVKARGPGLEKTGVAVNKPAEFTVDAKHGGKAPLRVQVQDNEGCPVEALVKDNGNGTYSCSYVPRKPVKHTAMVSWGGVSIPNSPFRVNVGAGSHPNKVKVYGPGVAKTGLKAHEPTYFTVDCAEAGQGDVSIGIKCAPGVVGPAEADIDFDIIRNDNDTFTVKYTPRGAGSYTIMVLFADQATPTSPIRVKVEPSHDASKVKAEGPGLSRTGVELGKPTHFTVNAKAAGKGKLDVQFSGLTKGDAVRDVDIIDHHDNTYTVKYTPVQQGPVGVNVTYGGDPIPKSPFSVAVSPSLDLSKIKVSGLGEKVDVGKDQEFTVKSKGAGGQGKVASKIVGPSGAAVPCKVEPGLGADNSVVRFLPREEGPYEVEVTYDGVPVPGSPFPLEAVAPTKPSKVKAFGPGLQGGSAGSPARFTIDTKGAGTGGLGLTVEGPCEAQLECLDNGDGTCSVSYVPTEPGDYNINILFADTHIPGSPFKAHVVPCFDASKVKCSGPGLERATAGEVGQFQVDCSSAGSAELTIEICSEAGLPAEVYIQDHGDGTHTITYIPLCPGAYTVTIKYGGQPVPNFPSKLQVEPAVDTSGVQCYGPGIEGQGVFREATTEFSVDARALTQTGGPHVKARVANPSGNLTETYVQDRGDGMYKVEYTPYEEGLHSVDVTYDGSPVPSSPFQVPVTEGCDPSRVRVHGPGIQSGTTNKPNKFTVETRGAGTGGLGLAVEGPSEAKMSCMDNKDGSCSVEYIPYEAGTYSLNVTYGGHQVPGSPFKVPVHDVTDASKVKCSGPGLSPGMVRANLPQSFQVDTSKAGVAPLQVKVQGPKGLVEPVDVVDNADGTQTVNYVPSREGPYSISVLYGDEEVPRSPFKVKVLPTHDASKVKASGPGLNTTGVPASLPVEFTIDAKDAGEGLLAVQITDPEGKPKKTHIQDNHDGTYTVAYVPDVTGRYTILIKYGGDEIPFSPYRVRAVPTGDASKCTVTVSIGGHGLGAGIGPTIQIGEETVITVDTKAAGKGKVTCTVCTPDGSEVDVDVVENEDGTFDIFYTAPQPGKYVICVRFGGEHVPNSPFQVTALAGDQPSVQPPLRSQQLAPQYTYAQGGQQTWAPERPLVGVNGLDVTSLRPFDLVIPFTIKKGEITGEVRMPSGKVAQPTITDNKDGTVTVRYAPSEAGLHEMDIRYDNMHIPGSPLQFYVDYVNCGHVTAYGPGLTHGVVNKPATFTVNTKDAGEGGLSLAIEGPSKAEISCTDNQDGTCSVSYLPVLPGDYSILVKYNEQHVPGSPFTARVTGDDSMRMSHLKVGSAADIPINISETDLSLLTATVVPPSGREEPCLLKRLRNGHVGISFVPKETGEHLVHVKKNGQHVASSPIPVVISQSEIGDASRVRVSGQGLHEGHTFEPAEFIIDTRDAGYGGLSLSIEGPSKVDINTEDLEDGTCRVTYCPTEPGNYIINIKFADQHVPGSPFSVKVTGEGRVKESITRRRRAPSVANVGSHCDLSLKIPEISIQDMTAQVTSPSGKTHEAEIVEGENHTYCIRFVPAEMGTHTVSVKYKGQHVPGSPFQFTVGPLGEGGAHKVRAGGPGLERAEAGVPAEFSIWTREAGAGGLAIAVEGPSKAEISFEDRKDGSCGVAYVVQEPGDYEVSVKFNEEHIPDSPFVVPVASPSGDARRLTVSSLQESGLKVNQPASFAVSLNGAKGAIDAKVHSPSGALEECYVTEIDQDKYAVRFIPRENGVYLIDVKFNGTHIPGSPFKIRVGEPGHGGDPGLVSAYGAGLEGGVTGNPAEFVVNTSNAGAGALSVTIDGPSKVKMDCQECPEGYRVTYTPMAPGSYLISIKYGGPYHIGGSPFKAKVTGPRLVSNHSLHETSSVFVDSLTKATCAPQHGAPGPGPADASKVVAKGLGLSKAYVGQKSSFTVDCSKAGNNMLLVGVHGPRTPCEEILVKHVGSRLYSVSYLLKDKGEYTLVVKWGDEHIPGSPYRVVVP
Click to Show/Hide
|
|---|