Details of Host Protein
| Host Protein General Information (ID: PT1018) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Protein Name |
Sequestosome-1 p62 (SQSTM1)
|
Gene Name |
SQSTM1
|
||||||
| Host Species |
Homo sapiens
|
Uniprot Entry Name |
SQSTM_HUMAN
|
||||||
| Subcellular Location |
Cytoplasm; cytosol Late endosome
|
||||||||
| External Link | |||||||||
| NCBI Gene ID | |||||||||
| Uniprot ID | |||||||||
| Ensembl ID | |||||||||
| HGNC ID | |||||||||
| Function in Host |
Autophagy receptor required for selective macroautophagy (aggrephagy). Functions as a bridge between polyubiquitinated cargo andautophagosomes. Interacts directly with both the cargo to becomedegraded and an autophagy modifier of the MAP1 LC3 family. Along with WDFY3, involved in the formation andautophagic degradation of cytoplasmic ubiquitin-containing inclusions (p62 bodies, ALIS/aggresome-like induced structures). Along with WDFY3, required to recruit ubiquitinated proteins to PML bodies in the nucleus. May regulate the activation ofNFKB1 by TNF-alpha, nerve growth factor (NGF) and interleukin-1. Mayplay a role in titin/TTN downstream signaling in muscle cells. Mayregulate signaling cascades through ubiquitination. Adapter thatmediates the interaction between TRAF6 and CYLD. May beinvolved in cell differentiation, apoptosis, immune response andregulation of K (+) channels. Involved in endosome organization byretaining vesicles in the perinuclear cloud: following ubiquitinationby RNF26, attracts specific vesicle-associated adapters, forming amolecular bridge that restrains cognate vesicles in the perinuclearregion and organizes the endosomal pathway for efficient cargotransport. Promotes relocalization of 'Lys-63'-linkedubiquitinated STING1 to autophagosomes. Acts as anactivator of the NFE2L2/NRF2 pathway via interaction with KEAP1:interaction inactivates the BCR (KEAP1) complex, promoting nuclearaccumulation of NFE2L2/NRF2 and subsequent expression of cytoprotectivegenes.
[1-7]
Click to Show/Hide
|
||||||||
| Related KEGG Pathway | |||||||||
| Cellular senescence | hsa04218 |
Pathway Map 
|
|||||||
| Shigellosis | hsa05131 |
Pathway Map 
|
|||||||
| Mitophagy - animal | hsa04137 |
Pathway Map 
|
|||||||
| Autophagy - animal | hsa04140 |
Pathway Map 
|
|||||||
| Necroptosis | hsa04217 |
Pathway Map 
|
|||||||
| Fluid shear stress and atherosclerosis | hsa05418 |
Pathway Map 
|
|||||||
| 3D Structure |
|
||||||||
| Host Protein - Virus RNA Network | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Full List of Virus RNA Interacting with This Protien | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| RNA Region: 5'-UTR (hCoV-19/South Korea/KCDC03/2020 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[8] | |||||||
| Strains Name |
hCoV-19/South Korea/KCDC03/2020
|
||||||||
| Strains Family |
Alpha (B.1.1.7)
|
||||||||
| RNA Binding Region |
5'-UTR
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Interaction Type | Directly bind to SARS-CoV-23 RNA's 5' UTR region | ||||||||
| Infection Cells | Vero cells (epithelial kidney cell) (CVCL_0059 ) | ||||||||
| Cell Originated Tissue | kidney | ||||||||
| Infection Time | 24 h | ||||||||
| Interaction Score | P-adjust < 0.05 | ||||||||
| Method Description | Modified RNA antisense purification coupled with mass spectrometry (RAP-MS); label-free quantification (LFQ); liquid chromatography with tandem mass spectrometry (LC-MS/MS) | ||||||||
| RNA Region: Not Specified Virus Region (hCov-OC43/VR-759 Quebec/2019 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[8] | |||||||
| Strains Name |
hCov-OC43/VR-759 Quebec/2019
|
||||||||
| Strains Family |
Beta
|
||||||||
| RNA Binding Region |
Not Specified Virus Region
|
||||||||
| Virus Name |
Human coronavirus OC43 (HCov-OC43)
|
||||||||
| Infection Cells | HCT-8 cells (Human ileocaecal adenocarcinoma cell) HCT-8 cells (Human ileocaecal adenocarcinoma cell) (CVCL_2478 ) | ||||||||
| Cell Originated Tissue | Ileocecum | ||||||||
| Infection Time | 12h; 24; 36h; 48h | ||||||||
| Interaction Score | p_value < 0.05; FDR < 10% | ||||||||
| Method Description | Modified RNA antisense purification coupled with mass spectrometry (RAP-MS); label-free quantification (LFQ); liquid chromatography with tandem mass spectrometry (LC-MS/MS) | ||||||||
| RNA Region: Not Specified Virus Region (hCoV-19/England/02/2020 ) | |||||||||
| RNA Region Details |
RNA Info
 Click to show the detail information of this RNA binding region
Click to show the detail information of this RNA binding region
|
[9] | |||||||
| Strains Name |
hCoV-19/England/02/2020
|
||||||||
| Strains Family |
Beta (B.1.351)
|
||||||||
| RNA Binding Region |
Not Specified Virus Region
|
||||||||
| Virus Name |
Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2)
|
||||||||
| Infection Cells | Calu-3 cells (Human lung cancer cell) Calu-3 cells (Human lung cancer cell) (CVCL_0609 ) | ||||||||
| Cell Originated Tissue | Lung | ||||||||
| Infection Time | 24 h | ||||||||
| Interaction Score | P-adjust = 0.004 | ||||||||
| Method Description | UV protein-RNA crosslinking; RNA interactome capture (cRIC); RNA antisense purification coupled with mass spectrometry (RAP-MS) | ||||||||
| Differential Gene Expression During SARS-COV-2 Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Phosphorylation after Virus Infection | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
P437
[10] |
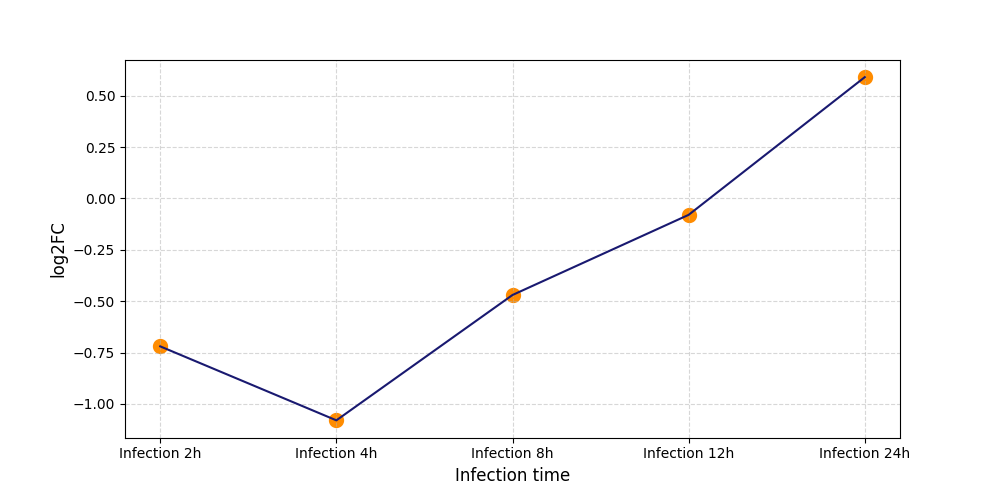 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S170
[11] |
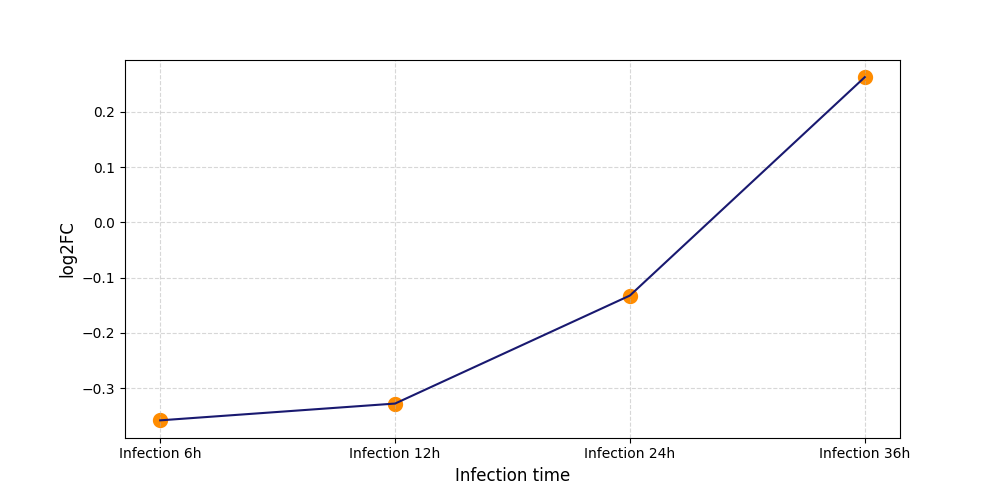 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S176
[11] |
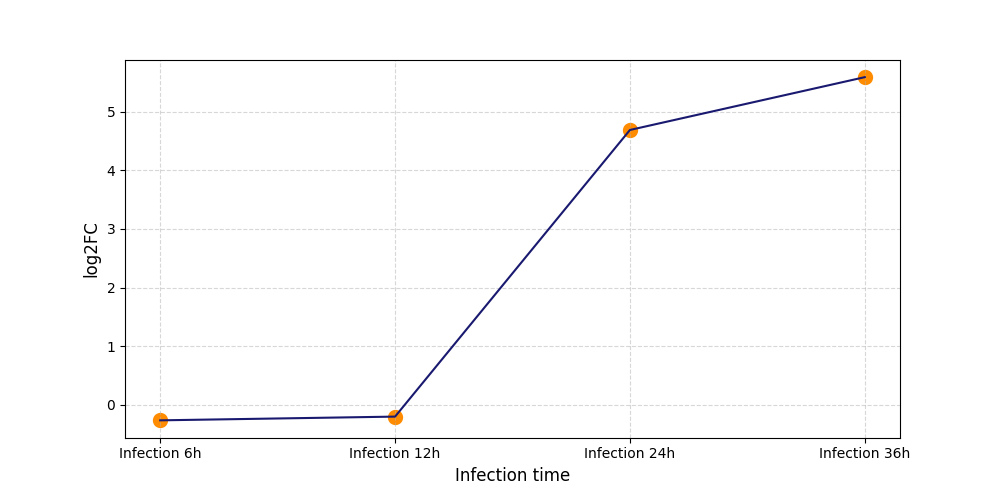 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S180
[11] |
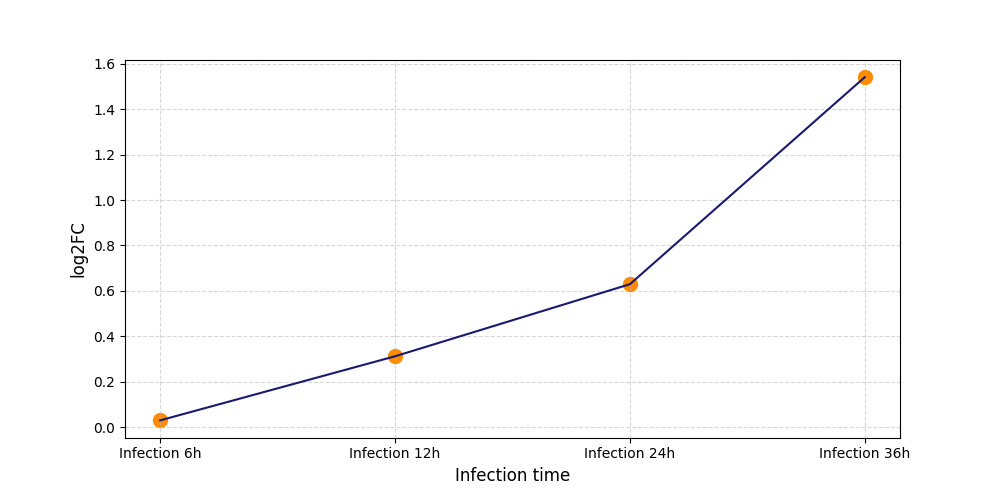 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S182
[11] |
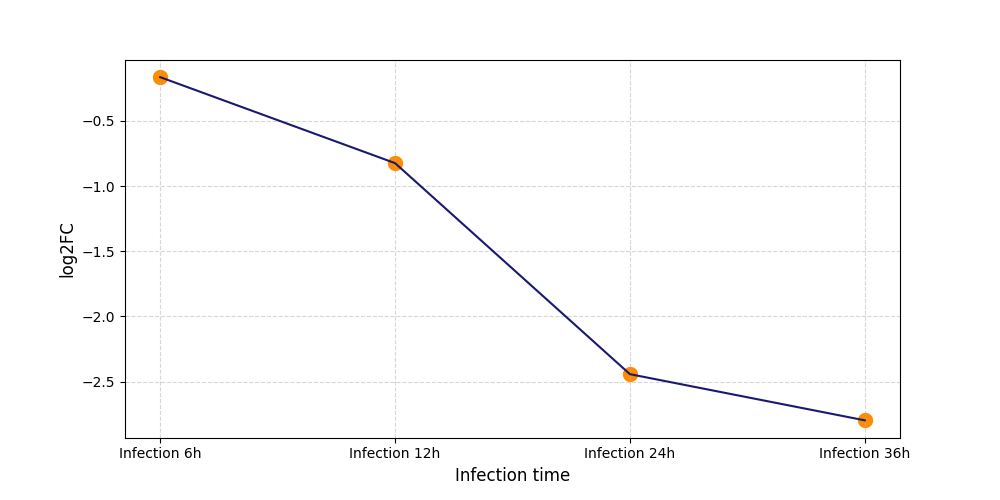 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S207
[10] |
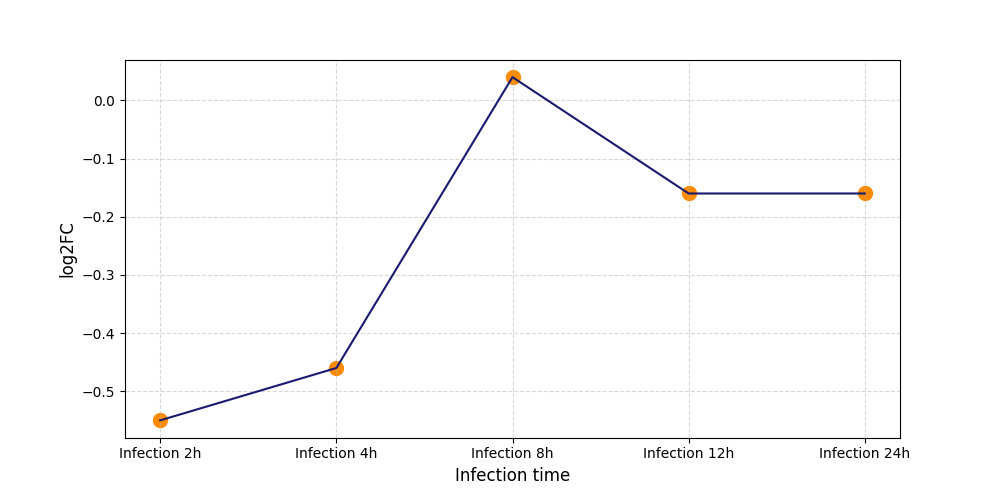 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S207
[11] |
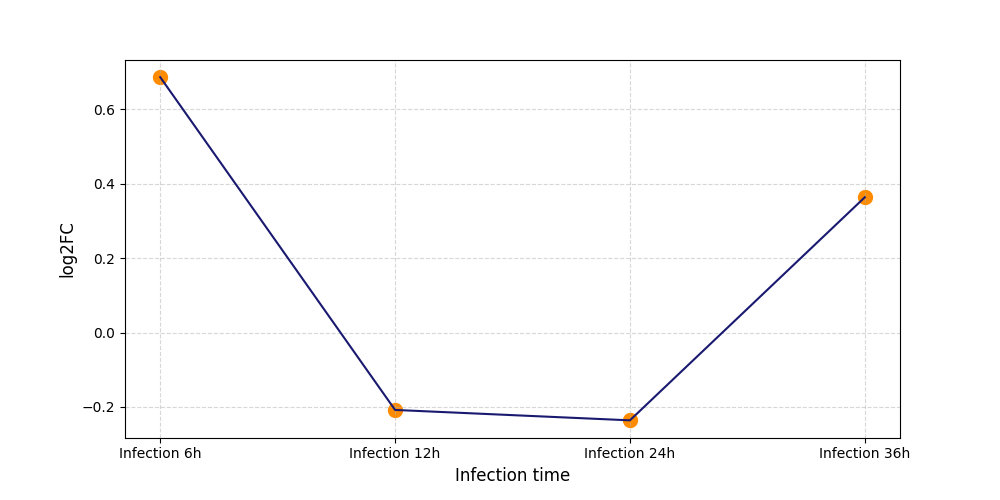 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S226
[11] |
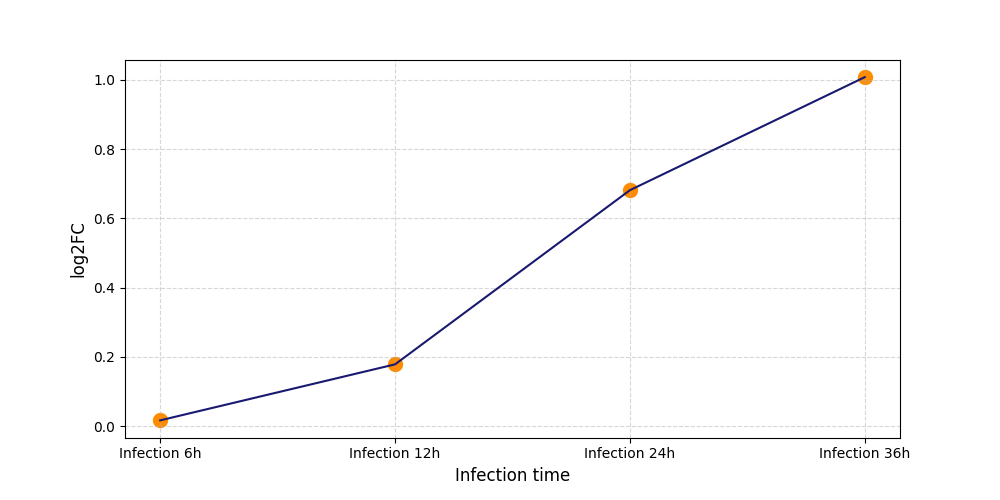 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S233
[11] |
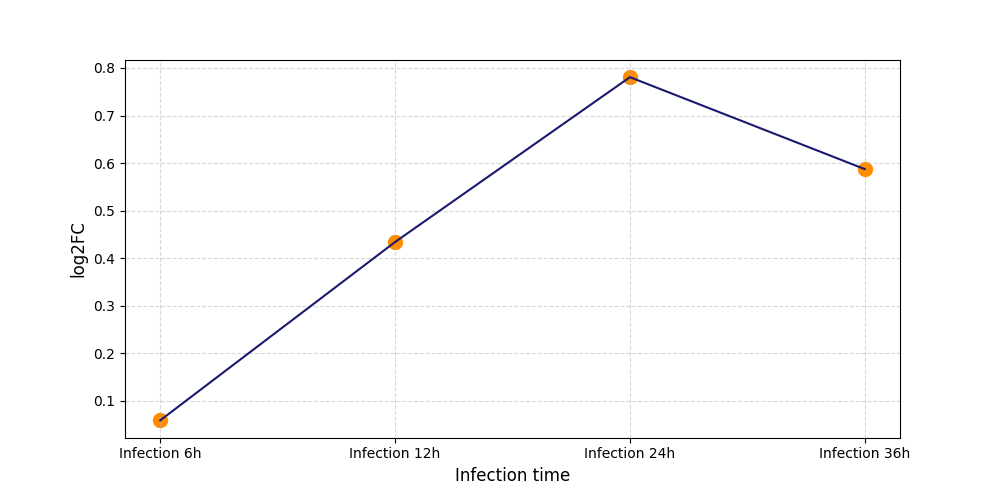 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S24
[11] |
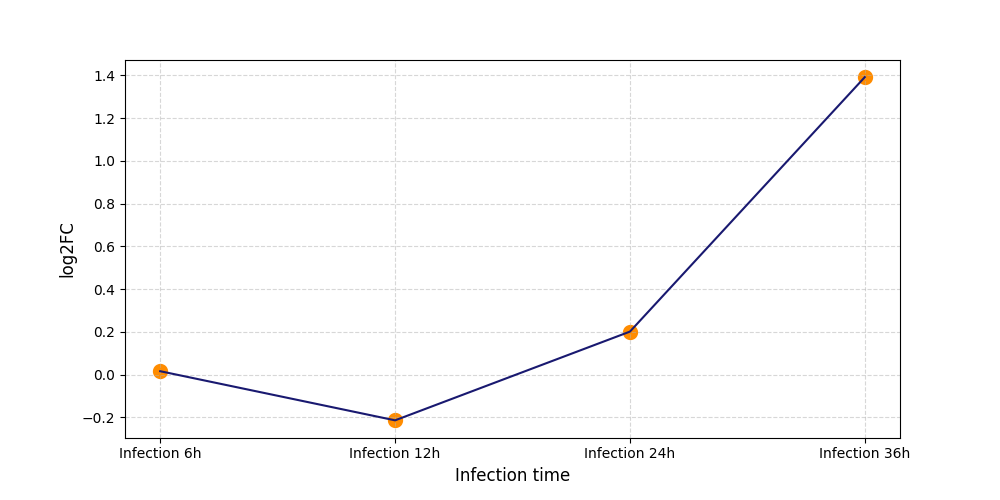 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S249
[11] |
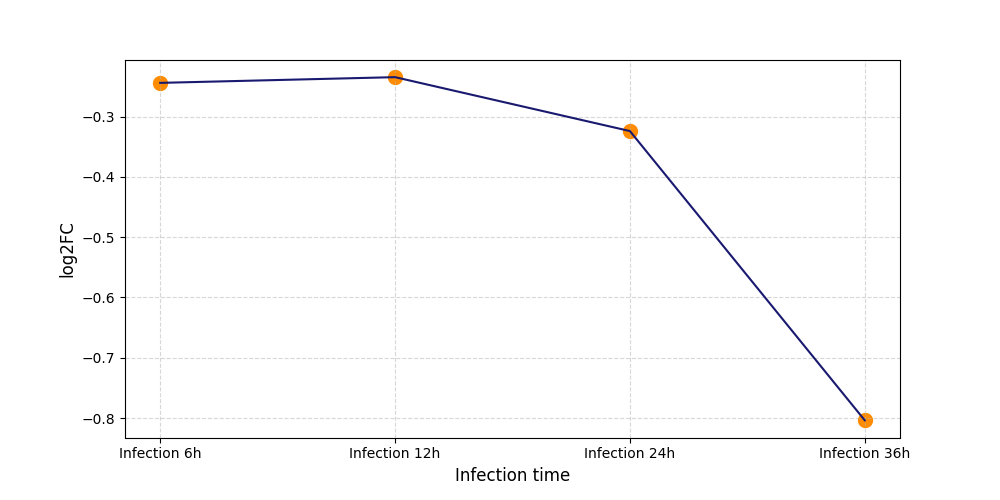 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S266
[11] |
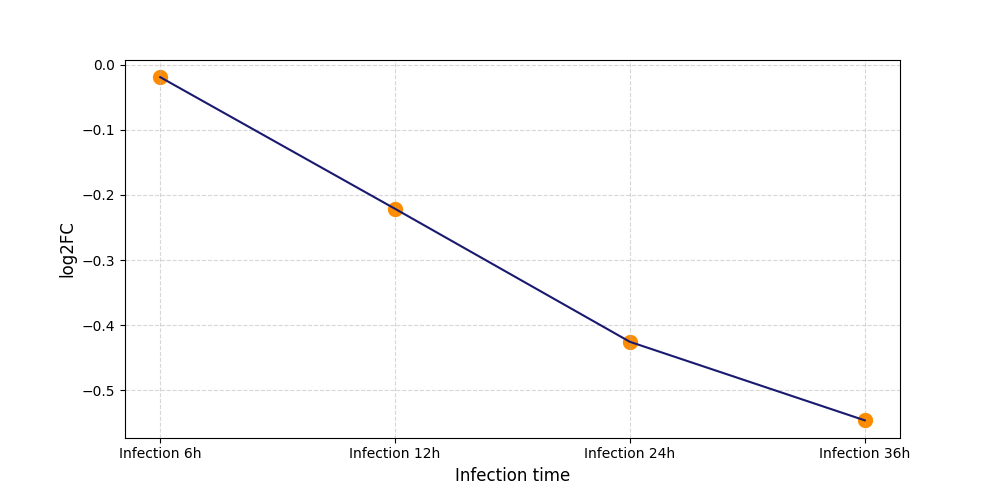 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S272
[10] |
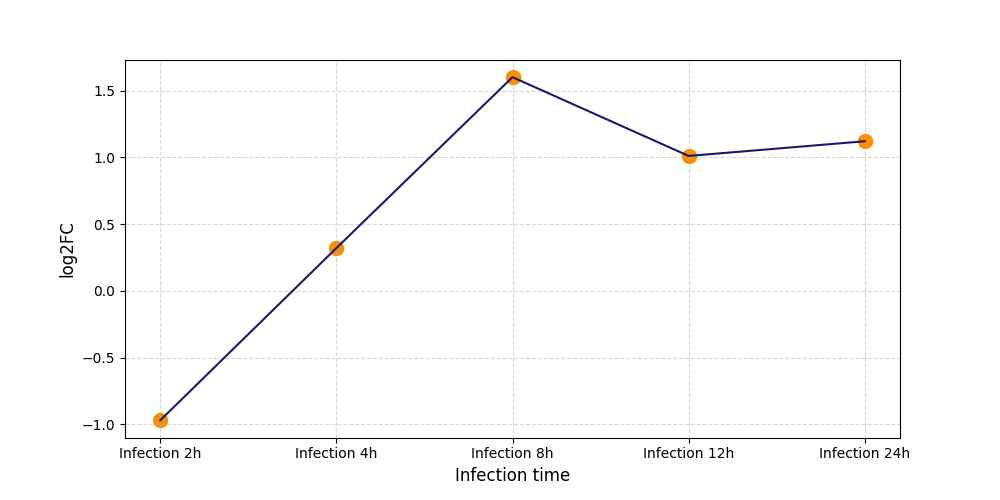 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S272
[11] |
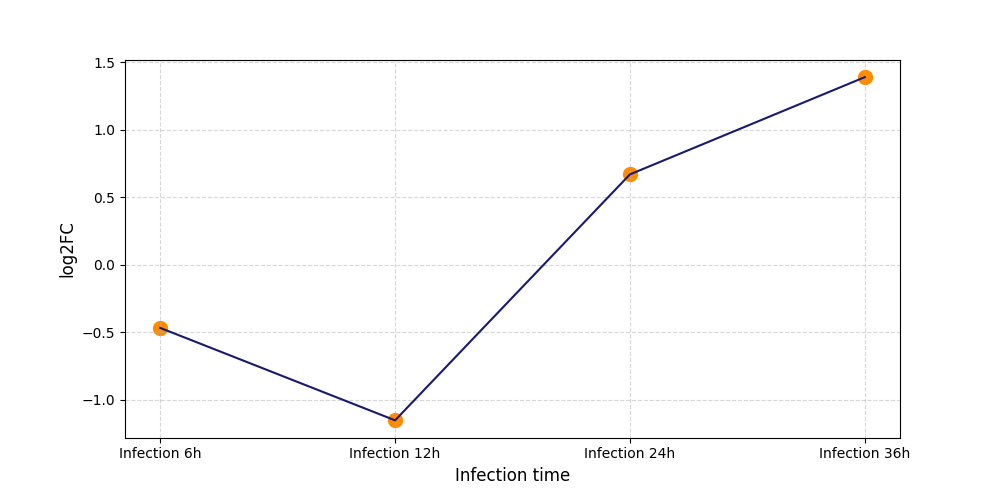 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S277
[11] |
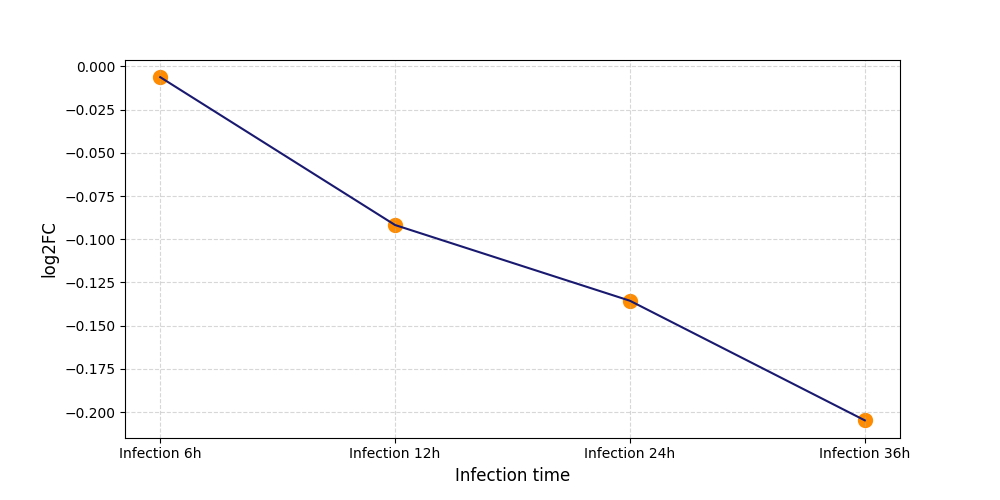 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S284
[11] |
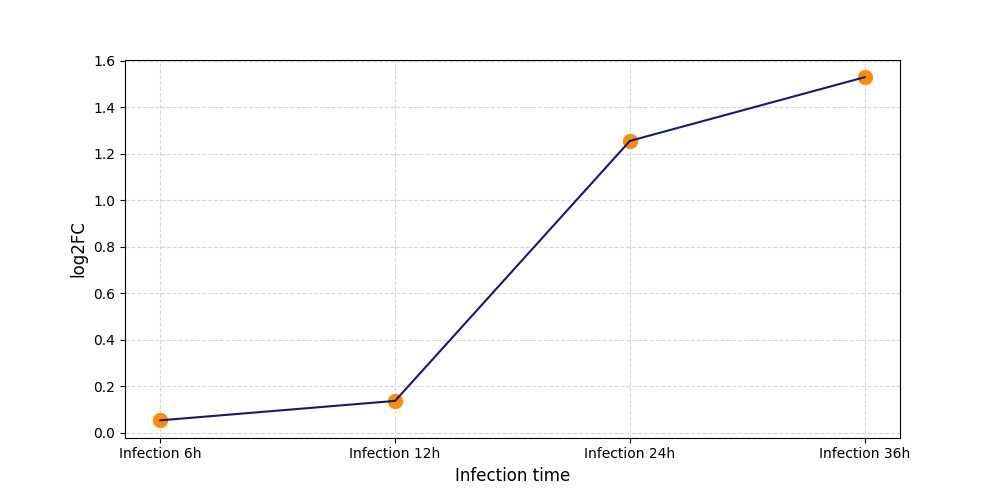 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S294
[11] |
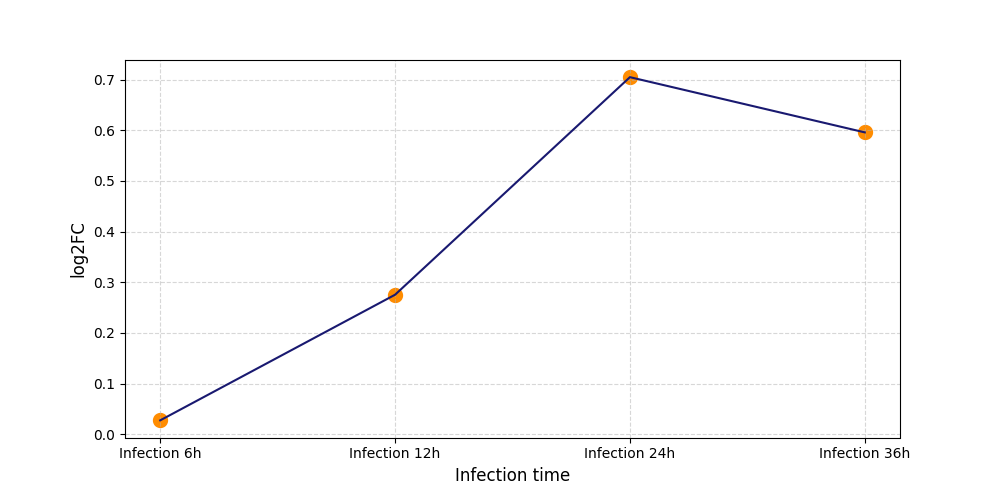 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S306
[11] |
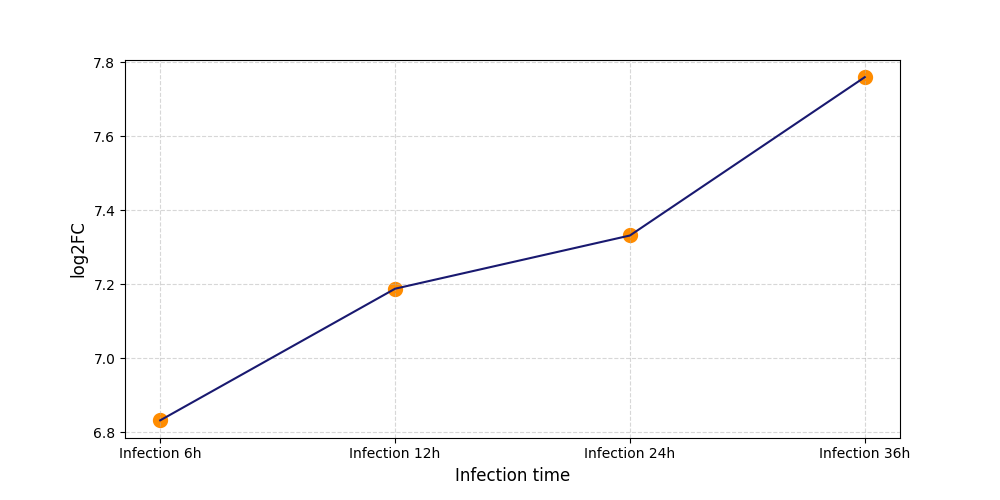 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S318
[11] |
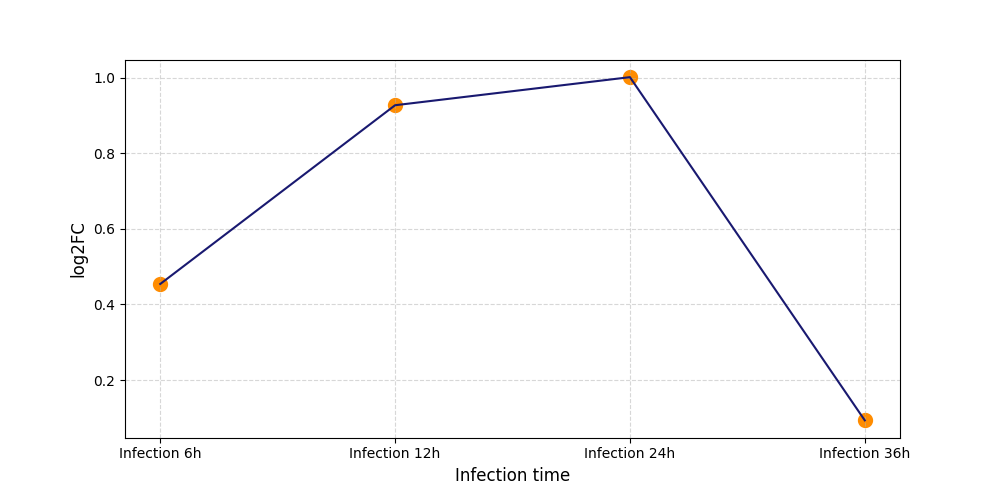 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S328
[11] |
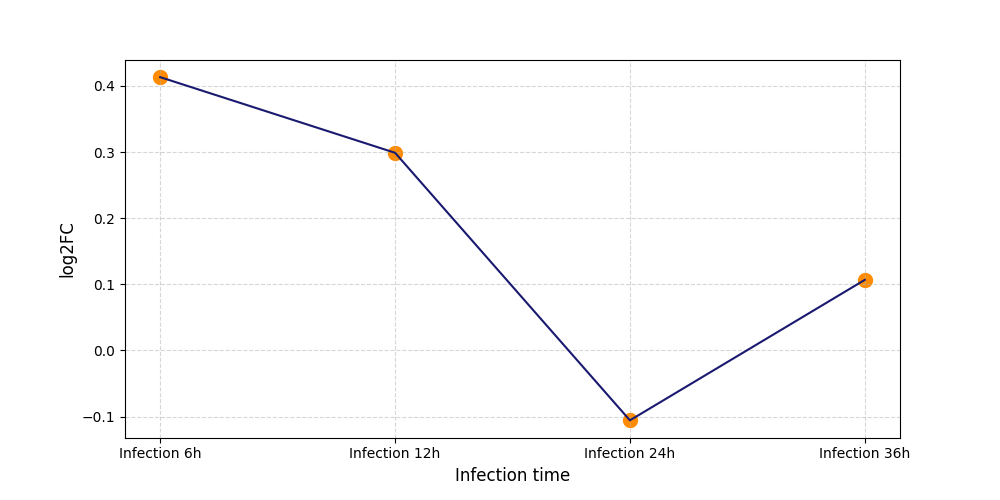 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S332
[11] |
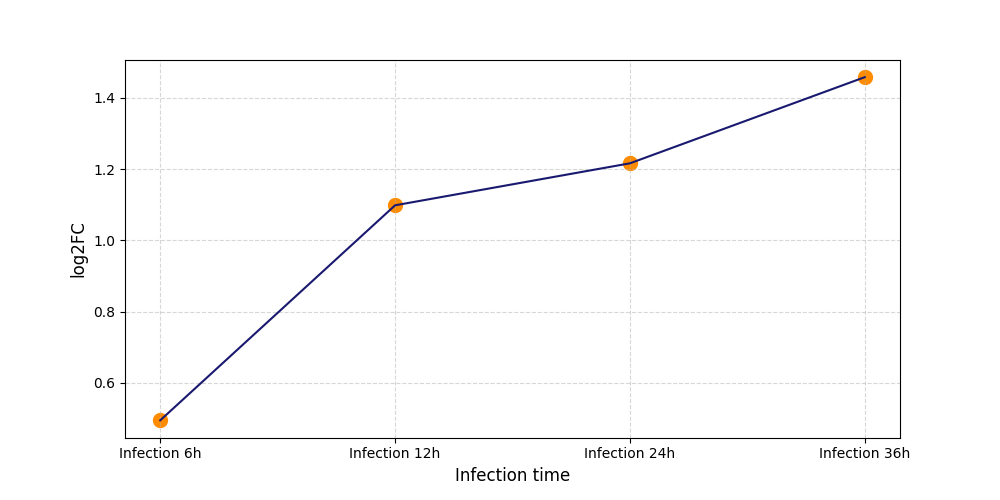 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S366
[11] |
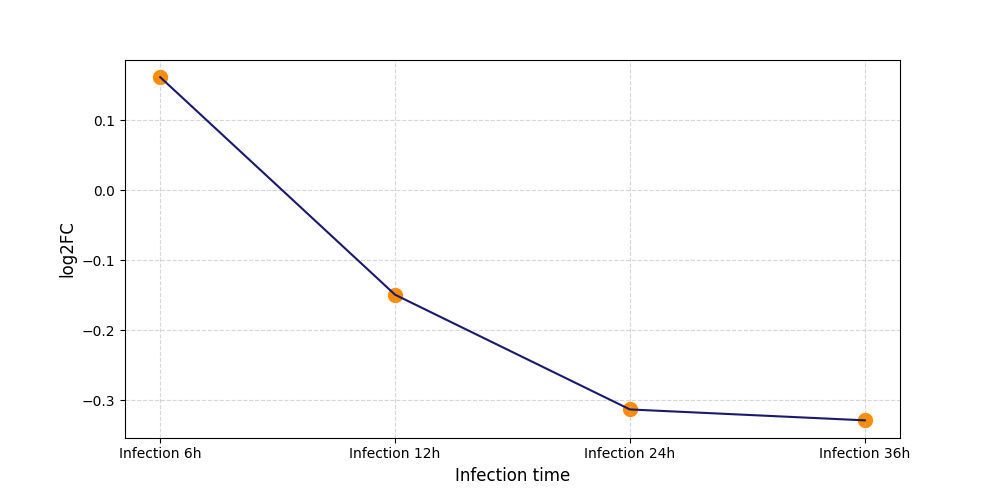 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
S403
[11] |
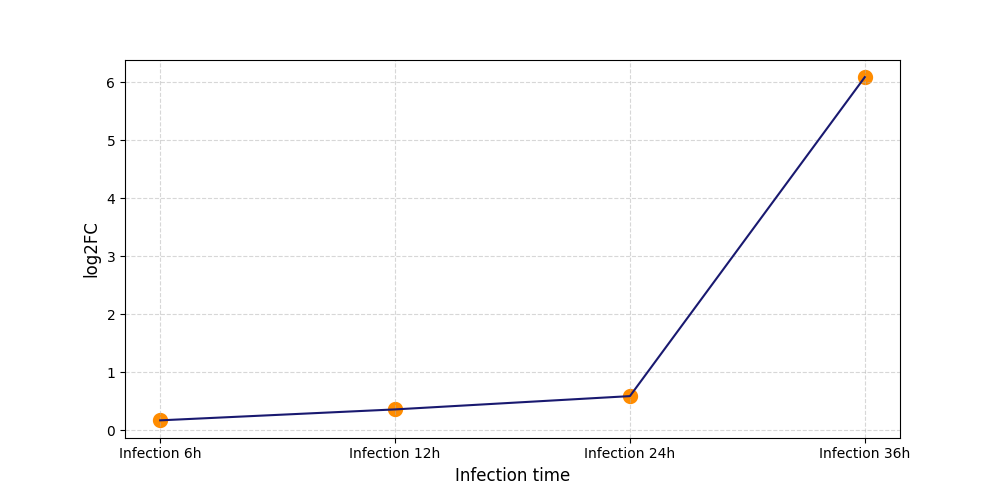 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T269
[11] |
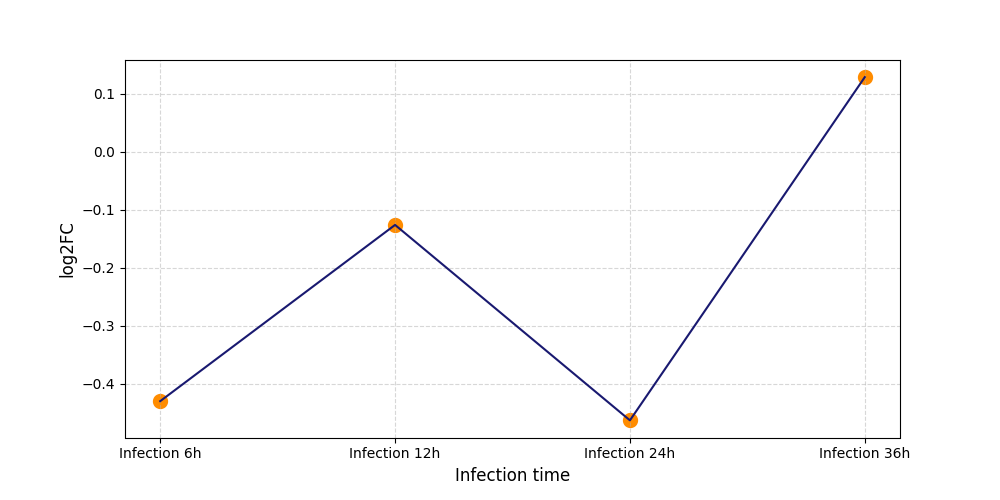 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
T278
[11] |
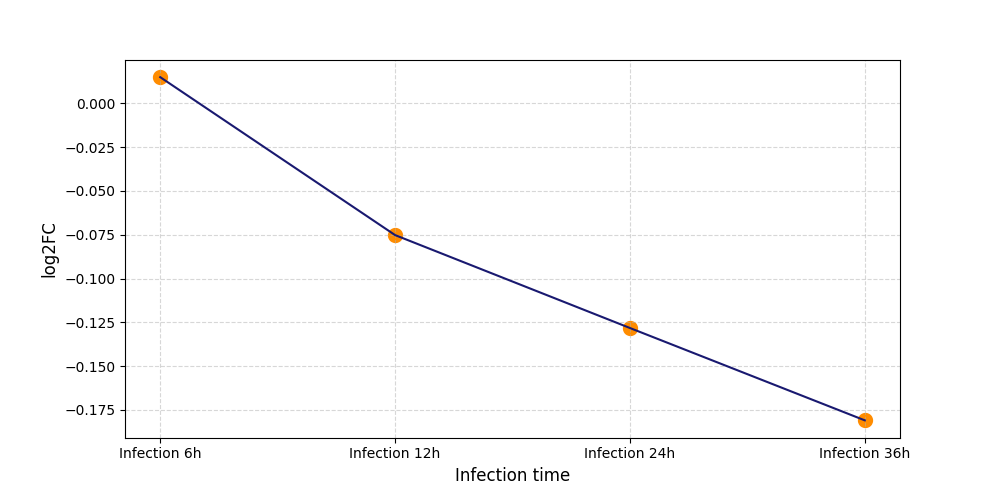 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein Sequence Information |
MASLTVKAYLLGKEDAAREIRRFSFCCSPEPEAEAEAAAGPGPCERLLSRVAALFPALRPGGFQAHYRDEDGDLVAFSSDEELTMAMSYVKDDIFRIYIKEKKECRRDHRPPCAQEAPRNMVHPNVICDGCNGPVVGTRYKCSVCPDYDLCSVCEGKGLHRGHTKLAFPSPFGHLSEGFSHSRWLRKVKHGHFGWPGWEMGPPGNWSPRPPRAGEARPGPTAESASGPSEDPSVNFLKNVGESVAAALSPLGIEVDIDVEHGGKRSRLTPVSPESSSTEEKSSSQPSSCCSDPSKPGGNVEGATQSLAEQMRKIALESEGRPEEQMESDNCSGGDDDWTHLSSKEVDPSTGELQSLQMPESEGPSSLDPSQEGPTGLKEAALYPHLPPEADPRLIESLSQMLSMGFSDEGGWLTRLLQTKNYDIGAALDTIQYSKHPPPL
Click to Show/Hide
|
|---|